Menslike hele eksoom-volgordebepaling
Diensvoordele
● Geteikende proteïenkoderende streek: deur die vaslegging en volgorde van proteïenkoderende streek, word hWES gebruik om variante wat met proteïenstruktuur verband hou, te openbaar;
● Hoë Akkuraatheid: met 'n hoë volgorde-diepte, hWES fasiliteer opsporing van algemene variante en skaars variante met frekwensies laer as 1%;
● Koste-effektief: hWES lewer ongeveer 85% van menslike siektemutasies uit 1% van menslike genoom;
● Vyf streng QC-prosedures wat die hele proses dek met Q30>85% gewaarborg.
Voorbeeld spesifikasies
| Platform
| Biblioteek
| Exon Capture Strategie
| Beveel volgordebepalingstrategie aan
|
|
Illumina NovaSeq-platform
| PE150 | Agilent SureSelect Human All Exon V6 IDT xGen Exome Hyb Panel V2 | 5 Gb 10 Gb |
Voorbeeldvereistes
| Voorbeeld tipe
| Bedrag(Qubit®)
| Volume
| Konsentrasie
| Suiwerheid (NanoDrop™) |
|
Genomiese DNA
| ≥ 300 ng | ≥ 15 μL | ≥ 20 ng/μL | OD260/280=1.8-2.0 geen agteruitgang, geen kontaminasie
|
Aanbevole volgorde-diepte
Vir Mendeliese afwykings/skaars siektes: effektiewe volgordebepalingsdiepte bo 50×
Vir tumormonsters: effektiewe volgordebepalingsdiepte bo 100×
Bioinformatika


Dienswerkvloei

Voorbeeld aflewering

DNA onttrekking

Biblioteek konstruksie

Opeenvolging

Data-analise

Data aflewering

Na-verkope dienste
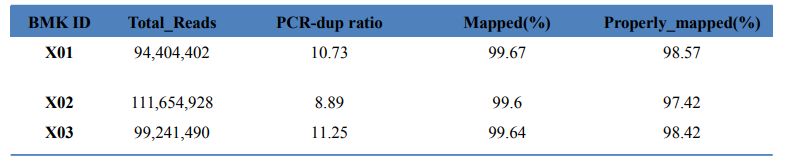
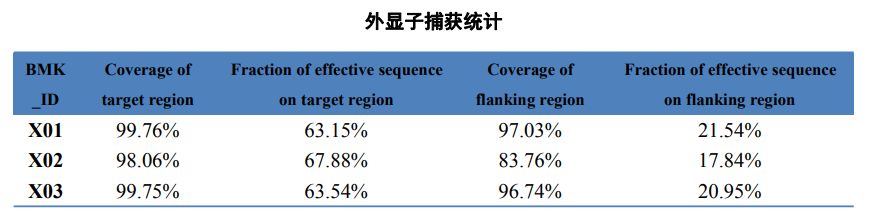
1.Belyningstatistieke
Tabel 1 Statistiek van kaartresultaat
Tabel 2 Statistiek van eksomvang
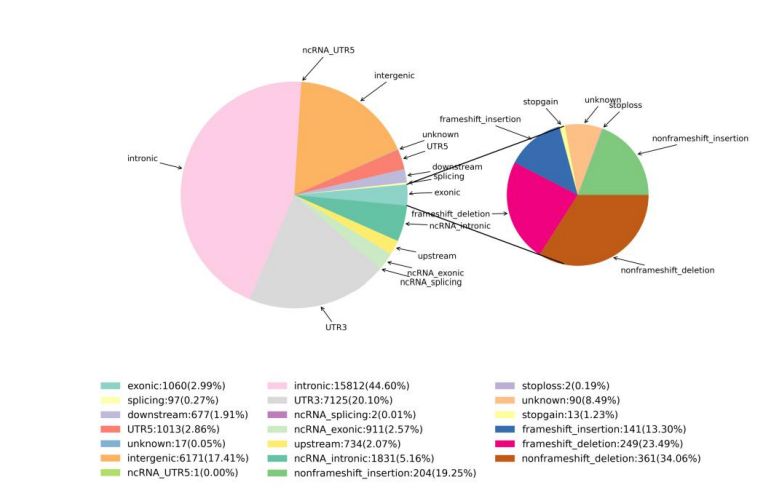
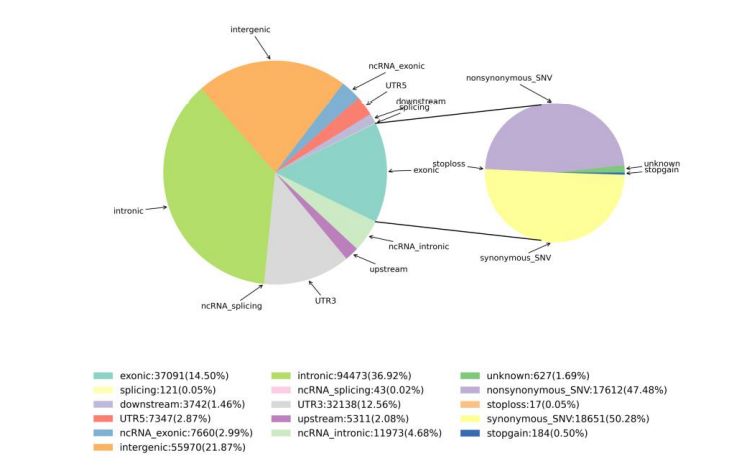
2.Variasie-opsporing
Figuur 1 Statistiek van SNV en InDel
3.Gevorderde Analise
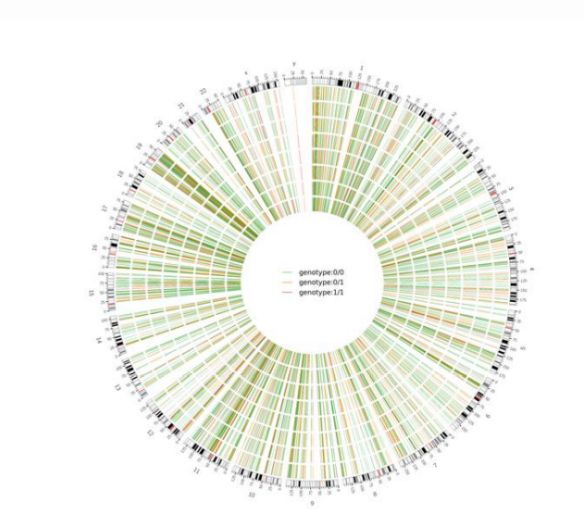
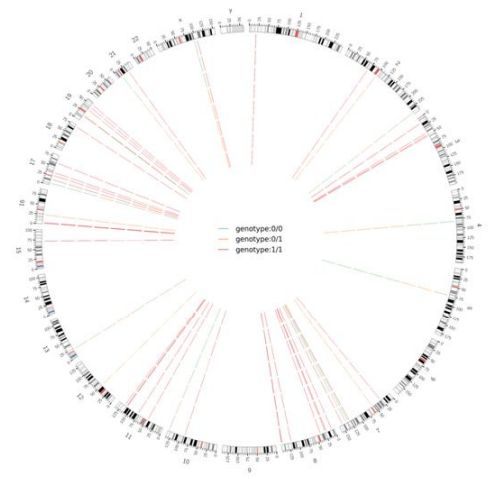
Figuur 2 Circos plot van genoomwye skadelike SNV en InDel
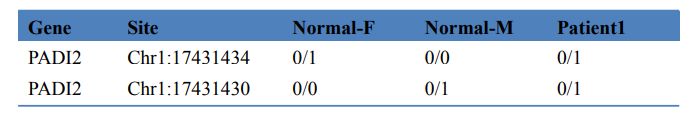
Tabel 3 Sifting van siekteveroorsakende gene