TRANSKRIPTOMIKER
natur
KOMMUNIKATIONER
Fuldlængde transkriptkarakterisering af SF3B1-mutation i kronisk lymfatisk leukæmi afslører nedregulering af tilbageholdte introner
Afskrifter i fuld længde|Nanopore-sekventering|Alternativ isoform analyse
Baggrund
Somatiske mutationer i splejsningsfaktor SF3B1 er blevet rapporteret i vid udstrækning at associere med forskellige kræftformer, herunder kronisk lymfatisk leukæmi (CLL), uveal melanom, brystkræft osv. Derudover har kortlæste transkriptomiske undersøgelser afsløret afvigende splejsningsmønstre induceret af SF3B1-mutationer.Imidlertid har undersøgelser af disse alternative splejsningsmønstre længe været begrænset til hændelsesniveau og mangel på viden på isoform-niveau på grund af begrænsningen af kortlæste samlede transskriptioner.Her blev nanopore-sekventeringsplatformen introduceret for at generere fuldlængde transkripter, som bemyndigede inverstigation på AS-isoformer.
Eksperimentelt design
Eksperimenter
Gruppering:1. CLL-SF3B1(WT) 2. CLL-SF3B1(K700E-mutation);3. Normale B-celler
Sekvenseringsstrategi:MinION 2D bibliotek sekventering, PromethION 1D bibliotek sekventering;kortlæste data fra samme prøver
Sekvenseringsplatform:ONT MinION;ONT PromethION;
Bioinformatisk analyse
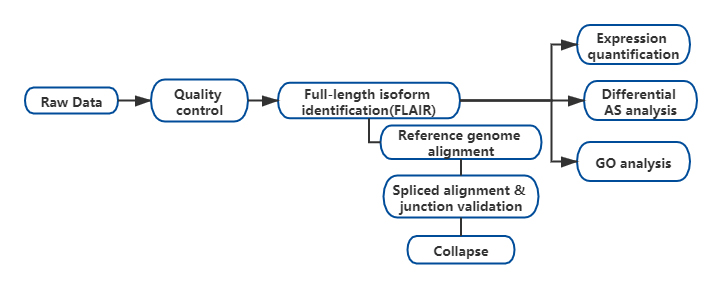
Resultater
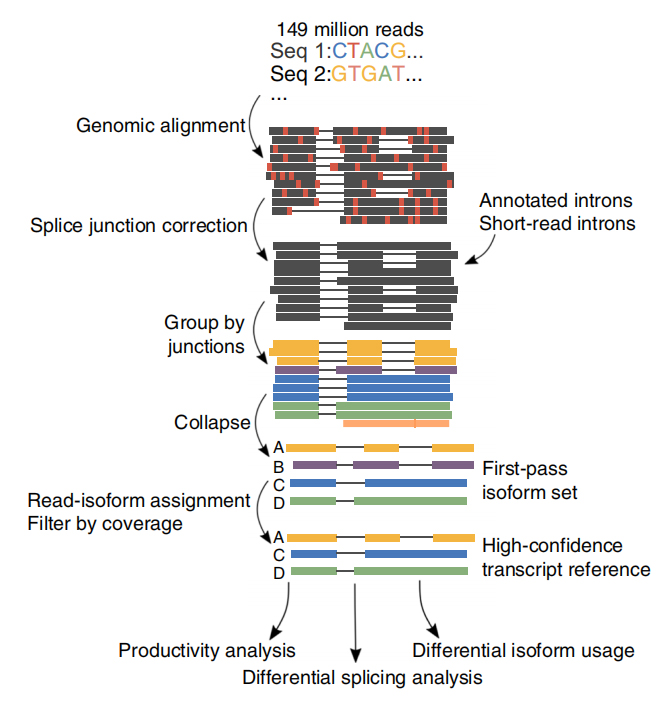
ENi alt 257 millioner aflæsninger blev genereret fra 6 CLL-prøver og 3 B-celler.I gennemsnit blev 30,5% af disse læsninger identificeret som fuldlængde transskriptioner.
FUL-længde alternativ isoformanalyse af RNA(FLAIR) blev udviklet til at generere et sæt højsikkerhedsisoformer.FLAIR kan opsummeres som:
Nanopore læser alignment: identificer generel transkriptionsstruktur baseret på referencegenom;
Splice-forbindelseskorrektion: korrekt sekvensfejl (rød) med splejsningssted fra enten kommenterede introner, introner fra kortlæste data eller begge dele;
Collapse: opsummer repræsentative isoformer baseret på splejsningsforbindelseskæder (førstegangssæt).Vælg isofrom med høj sikkerhed baseret på antallet af understøttende læsninger (Tærskel: 3).
Figur 1. FLAIR-analyse for at identificere fuld-længde isoformer forbundet med SF3B1-mutation i CLL
FLAIR identificerede 326.699 højsikkerhedssplejsede isoformer, hvoraf 90% er nye isoformer.De fleste af disse uannoterede isoformer viste sig at være nye kombinationer af kendte splejsningsforbindelser (142.971), mens resten af nye isoformer indeholdt enten tilbageholdt intron (21.700) eller ny exon (3594).
Long-read-sekvenser muliggør identifikation af mutant SF3B1-K700E-ændrede splejsningssteder på isoform-niveau.35 alternative 3'SS'er og 10 alternative 5'SS'er blev fundet at være signifikant differentielt splejset mellem SF3B1-K700E og SF3B1-WT.33 af de 35 ændringer blev nyopdaget af langlæste sekvenser.I Nanopore-data er fordelingen af afstanden mellem SF3B1-K700E-ændrede 3'SS'er til toppene af kanoniske steder omkring -20 bp, hvilket er væsentligt forskelligt fra en kontrolfordeling, svarende til hvad der er blevet rapporteret i CLL-kortaflæste sekvenser.Isoformer af ERGIC3-genet blev analyseret, hvor en ny isoform indeholdende det proksimale splejsningssted blev fundet mere rigeligt i SF3B1-K700E.Både proksimale og distale 3'SS var forbundet med distinkte AS-mønstre, der genererede flere isoformer.
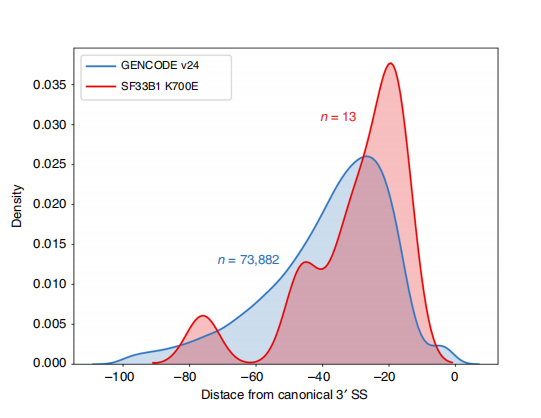
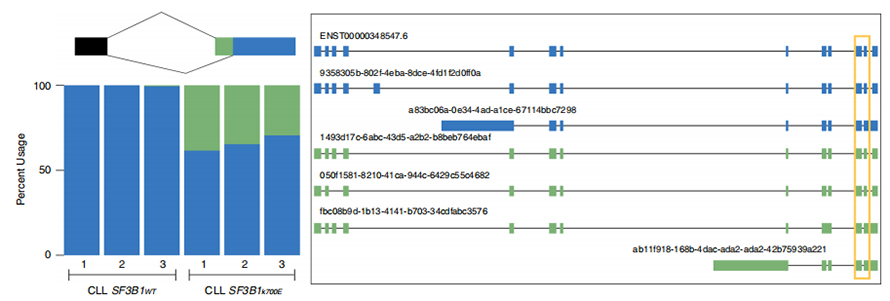
Figur 2. Alternative 3′ splejsningsmønstre identificeret med nanopore sekventeringsdata
IR-hændelsesbrugsanalyse har længe været begrænset i kortlæsningsbaseret analyse på grund af tillid til IR-identifikation og kvantificering.Ekspression af IR-isoformer i SF3B1-K700E og SF3B1-WT blev kvantificeret baseret på nanopore-sekvenser, hvilket afslørede en global nedregulering af IR-isoformer i SF3B1-K700E.
Figur 4. Landbrugsintensitet og netværksforbindelse på tværs af tre landbrugssystemer (A og B);Tilfældig skovanalyse(C) og sammenhæng mellem landbrugsintensitet og AMF-kolonisering (D)
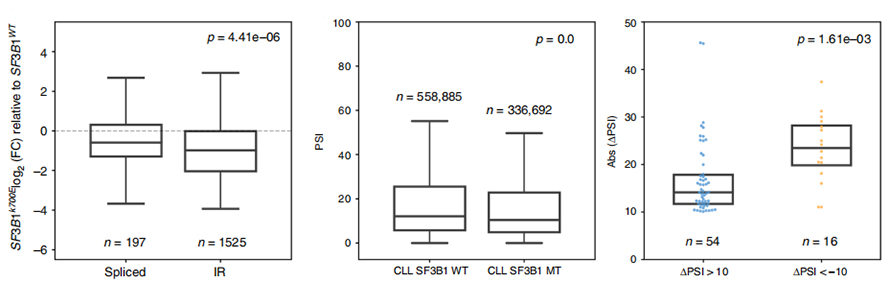
Figur 3. Intron-retentionshændelser er kraftigere nedreguleret i CLL SF3B1-K700E
Teknologi
Nanopore Long-read Sequencing
Nanopore sekventering er et enkelt molekyle i realtid elektrisk signal sekventeringsteknologi.
Double-strenged DNA eller RNA vil binde til nanoporøst protein indlejret i biofilmen og afvikles under føring af motorprotein.
DNA/RNA-strenge passerer gennem nanoporekanalprotein med en vis hastighed under påvirkning af spændingsforskel.
Molekuler genererer forskellige elektriske signaler i henhold til kemisk struktur.
Real-time detektion af sekvenser opnås ved base calling.
Udførelse af transkriptomsekventering i fuld længde
√ Datamætning
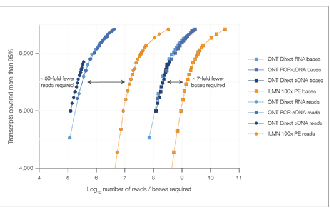
7 gange færre læsninger kræves for at opnå sammenlignelig datamætning.
√ Transskriptionsstrukturidentifikation
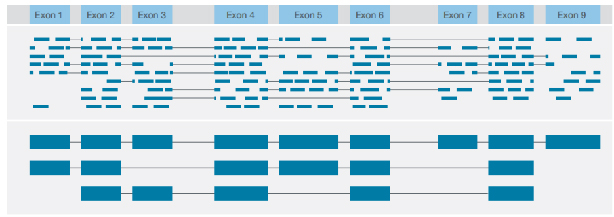
Identifikation af forskellige strukturelle varianter med konsensus i fuld længde af hver transkription
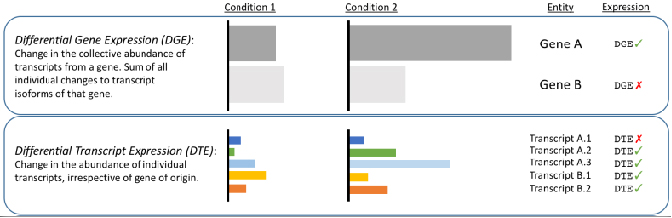
√ Differentialanalyse på transskriptionsniveau - Afslører ændringer skjult af korte læsninger
Reference
Tang AD, Soulette CM, Baren MJV, et al.Transkriptkarakterisering i fuld længde af SF3B1-mutation i kronisk lymfatisk leukæmi afslører nedregulering af tilbageholdte introner[J].Naturkommunikation.
Teknologi og højdepunkter sigter på at dele den seneste succesfulde anvendelse af forskellige high-throughput sekventeringsteknologier i forskellige forskningsarenaer samt geniale ideer inden for eksperimentelt design og datamining.
Indlægstid: Jan-08-2022

