Plante/dyrs helgenomsekventering
1. Servicefordele
Omfattende erfaring med genomsekventering for over 1000 arter.
Over 800 offentliggjorte sager med en akkumuleret effektfaktor på over 4000.
Omfattende bioinformatikanalyse på variationskald og funktionsanalyse.
2. Servicespecifikationer
| Platform | Bibliotek | Anbefalet sek. dybde | |
| Illumina | PE 150 | For SNP, InDel-kald ≥ 10x For SV, CNY-opkald ≥ 30x
| |
| Nanopore
| 8 kb | For SV, CNY-opkald ≥ 20x | |
| Pacbio | CCS | 15 kb | For SNP, InDel, SV, CNY opkald ≥ 10x |
3. Prøvekrav
| Platform | Konc.(ng/μL)
| Beløb (ng)
| Renhed
| Agarose gel
| ||
| OD260/280 | OD260/230 | 1. Ryd hovedbånd med ingen eller begrænsetnedbrydning observeret på gel. 2. Ingen eller begrænset RNA- eller proteinkontamination
| ||||
| Illumina | ≥1 | ≥30
| - | - | ||
| Nanopore
| ≥30 | Afhænger af dataudbytte 10μg/celle | 1,7-2,2 | ≥1,5 | ||
| Pacbio | CCS | ≥50 | 1,7-2,2 | 1,8-2,5 | ||
4. Bioinformationsanalyse
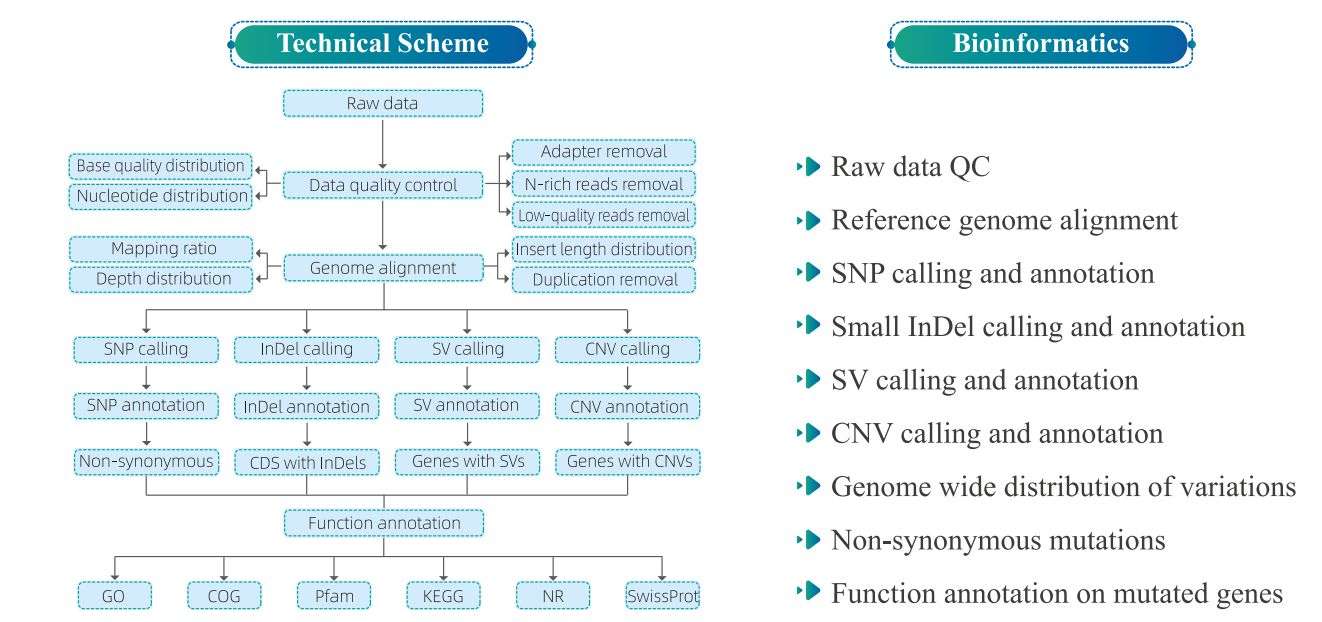
5. Service Work Flow

Prøve levering

DNA-ekstraktion

Biblioteksbyggeri

Sekvensering

Dataanalyse

Data levering
1) Statistik for genomkortlægning
Tabel 1 Statistik over kortlægningsresultat
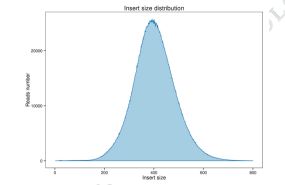
Figur 1 Fordeling af indsatsstørrelse og aflæser dækning.
2) Variationsdetektion
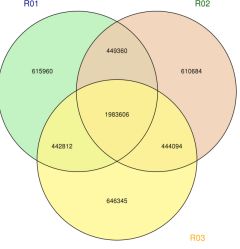
Figur 2 Statistik og annotering af SNP/INDEL/SV blandt prøver
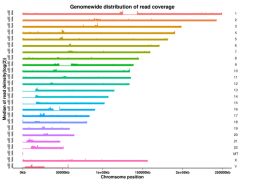
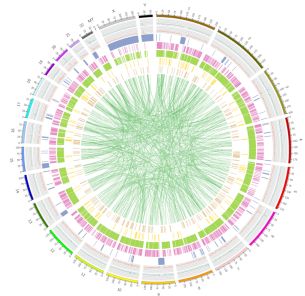
Figur 3 Genom-dækkende fordeling af vatiationer
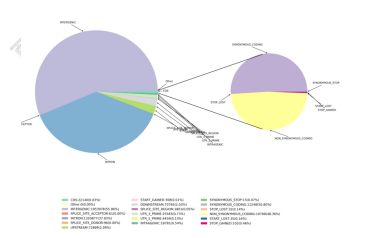
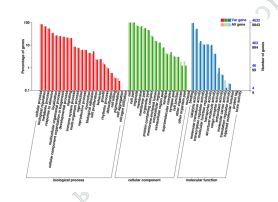
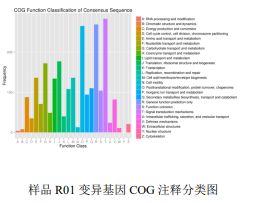
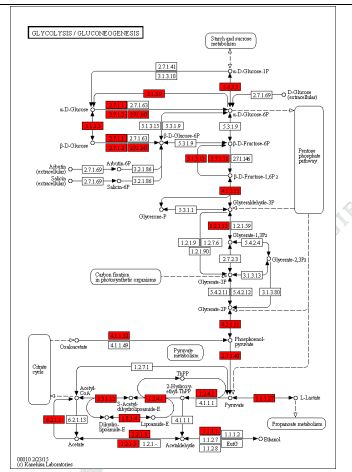
3) Funktionel annotering af variationer
| 2019 | Naturkommunikation | Resekventering af hele genomet afslører Brassica napus oprindelse og genetiske loci involveret i dets forbedring |
| 2020 | PNAS | Den evolutionære oprindelse og domesticeringshistorie af guldfisk (Carassius auratus) |
| 2021 | Plantebioteknologisk tidsskrift | Referencegenomer for de to dyrkede jutearter |
| 2021 | Plantebioteknologisk tidsskrift | Genomiske signaturer af vegetabilsk og oliefrø allopolyploid Brassicajuncea og genetiske loci, der kontrollerer ophobningen af glucosinolater |
| 2019 | Molekylær plante | Helgenom-resekventering af en verdensomspændende samling af rapsfrø afslører genetisk grundlag for deres økotypedivergens |
| 2022 | Havebrugsforskning | Genom-dækkende associationsanalyse giver molekylær indsigt i den naturlige variation af vandmelonfrøstørrelse |
| 2021 | Journal of Experimental Botany | Genom-dækkende associationsundersøgelse identificerer varianter af GhSAD1, der giver kuldetolerance i bomuld |
| 2021 | Journal of Experimental Botany | Genom-dækkende associationsundersøgelse og transkriptomsammenligning afslører nye QTL og kandidatgener, der kontrollerer kronbladstørrelsen i rapsfrø |