
16S/18S/ITS-Amplikon-Sequenzierung-NGS
Servicevorteile
● Isolationsfreie und schnelle Identifizierung der mikrobiellen Zusammensetzung in Umweltproben
● Hohe Auflösung bei gering vorkommenden Komponenten in Umweltproben
● Neuester QIIME2-Analysefluss mit vielfältigen Analysen in Bezug auf Datenbank, Annotation, OTU/ASV.
● Hoher Durchsatz, höhere Genauigkeit
● Anwendbar auf verschiedene mikrobielle Gemeinschaftsstudien
● BMK verfügt über umfassende Erfahrung mit über 100.000 Proben pro Jahr, die Boden, Wasser, Gas, Schlamm, Fäkalien, Därme, Haut, Fermentationsbrühe, Insekten, Pflanzen usw. abdecken.
● BMKCloud erleichterte die Dateninterpretation mit 45 personalisierten Analysetools
Leistungsbeschreibung
| SequenzierungPlattform | Bibliothek | Empfohlene Datenausbeute | Geschätzte Bearbeitungszeit |
| Illumina NovaSeq-Plattform | PE250 | 50K/100K/300K-Tags | 30 Tage |
Bioinformatische Analysen
● Qualitätskontrolle der Rohdaten
● OTU-Clustering/De-Noise (ASV)
● OTU-Anmerkung
● Alpha-Diversität
● Beta-Vielfalt
● Intergruppenanalyse
● Assoziationsanalyse anhand experimenteller Faktoren
● Funktionsgenvorhersage
Musteranforderungen und Lieferung
Probenanforderungen:
FürDNA-Extrakte:
| Beispielstyp | Menge | Konzentration | Reinheit |
| DNA-Extrakte | > 30 ng | > 1 ng/μl | AD260/280= 1,6-2,5 |
Für Umweltproben:
| Beispielstyp | Empfohlenes Probenahmeverfahren |
| Boden | Probenmenge: ca.5 g;Verbleibende verwelkte Substanz muss von der Oberfläche entfernt werden;Große Stücke zermahlen und durch einen 2-mm-Filter passieren;Aliquotierte Proben in sterilen EP-Röhrchen oder Zyrotöhrchen zur Reservierung. |
| Kot | Probenmenge: ca.5 g;Sammeln und aliquotieren Sie Proben zur Reservierung in sterilen EP-Röhrchen oder Kryoröhrchen. |
| Darminhalt | Proben müssen unter aseptischen Bedingungen verarbeitet werden.Gesammeltes Gewebe mit PBS waschen;Zentrifugieren Sie das PBS und sammeln Sie den Niederschlag in EP-Röhrchen. |
| Schlamm | Probenmenge: ca.5 g;Sammeln und aliquotieren Sie die Schlammprobe in einem sterilen EP-Röhrchen oder Kryoröhrchen zur Reservierung |
| Gewässer | Für Proben mit einer begrenzten Menge an Mikroben, wie z. B. Leitungswasser, Brunnenwasser usw., sammeln Sie mindestens 1 l Wasser und lassen Sie es durch einen 0,22-μm-Filter laufen, um die Mikroben auf der Membran anzureichern.Bewahren Sie die Membran in einem sterilen Röhrchen auf. |
| Haut | Kratzen Sie die Hautoberfläche vorsichtig mit einem sterilen Wattestäbchen oder einer chirurgischen Klinge ab und legen Sie sie in ein steriles Röhrchen. |
Empfohlene Musterlieferung
Frieren Sie die Proben 3-4 Stunden lang in flüssigem Stickstoff ein und lagern Sie sie zur Langzeitkonservierung in flüssigem Stickstoff oder bei -80 Grad.Der Probenversand mit Trockeneis ist erforderlich.
Service-Workflow

Musterlieferung

Bibliotheksbau

Sequenzierung

Datenanalyse

Kundendienst
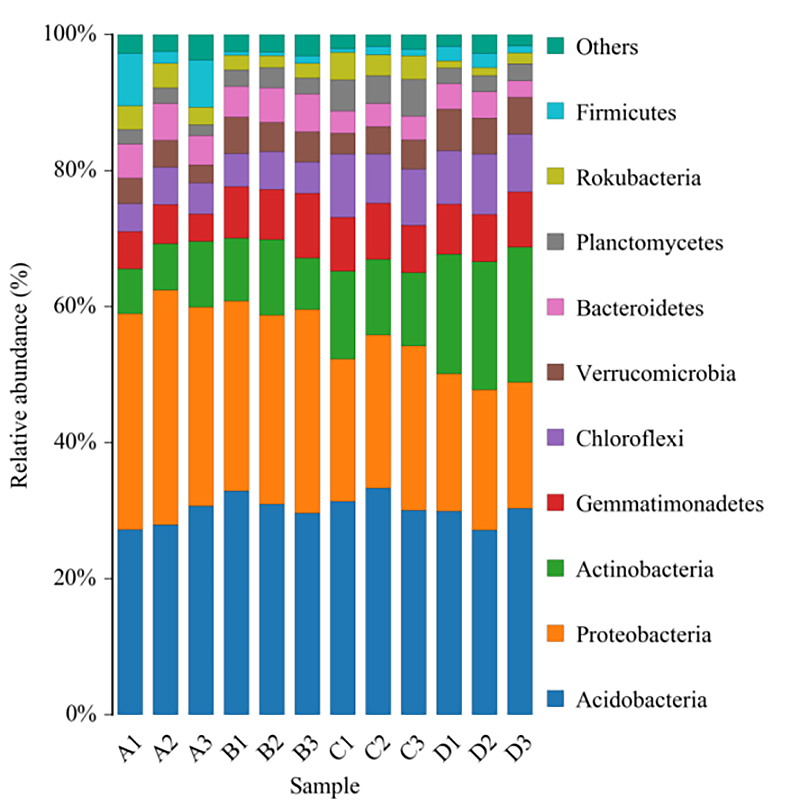
1. Artenverteilung
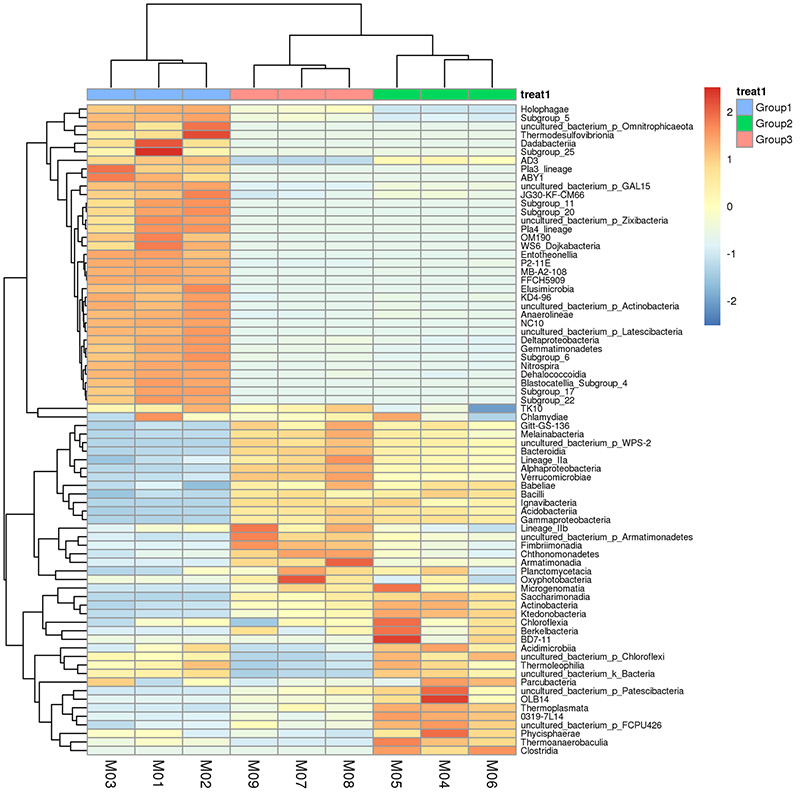
2.Heatmap: Clusterung des Artenreichtums
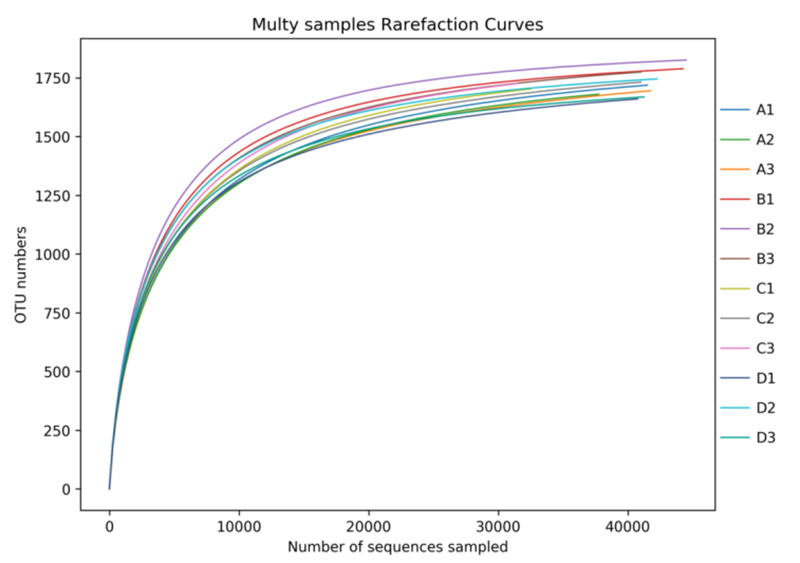
3.Seltene Fraktionskurve
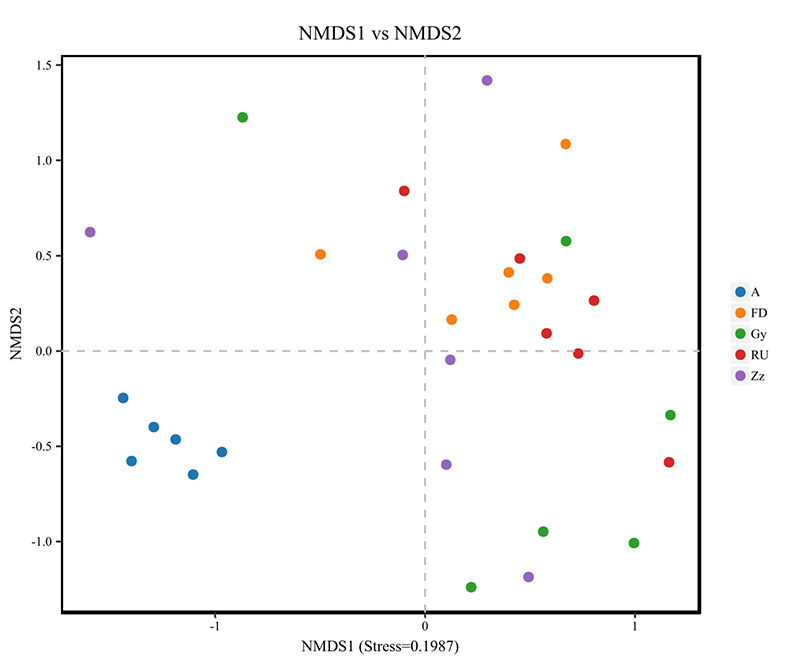
4.NMDS-Analyse
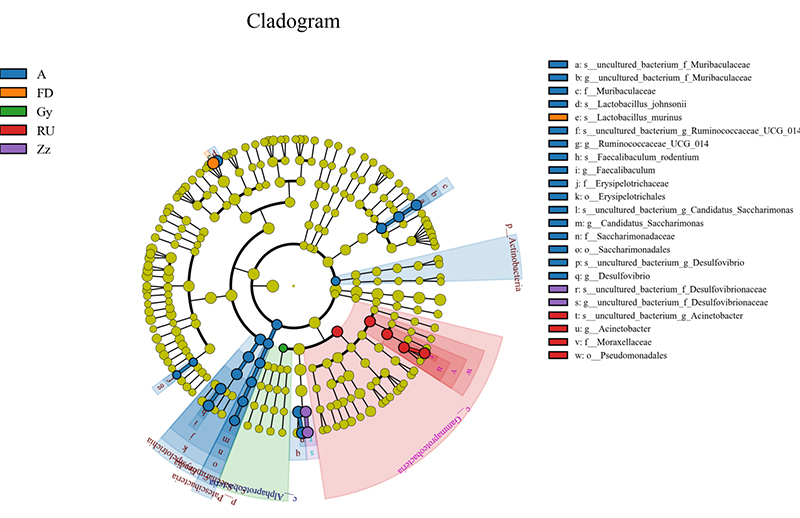
5. Lefse-Analyse
BMK-Fall
Übergewichtige Personen mit und ohne Typ-2-Diabetes weisen eine unterschiedliche mikrobielle Funktionsfähigkeit und Zusammensetzung des Darms auf
Veröffentlicht:Zellwirt & Mikrobe, 2019
Sequenzierungsstrategie:
Schlanke Nicht-Diabetiker (n=633);Übergewichtige Nicht-Diabetiker (n=494);Fettleibiger Typ-2-Diabetes (n=153);
Zielregion: 16S rDNA V1-V2
Plattform: Illumina Miseq (NGS-basierte Amplikonsequenzierung)
Eine Teilmenge der DNA-Extrakte wurde einer metagenomischen Sequenzierung auf Illumina Hiseq unterzogen
Wichtigste Ergebnisse
Mikrobielle Profile dieser Stoffwechselerkrankungen konnten erfolgreich differenziert werden.
Durch den Vergleich mikrobieller Merkmale, die durch 16S-Sequenzierung generiert wurden, wurde festgestellt, dass Fettleibigkeit mit Veränderungen der mikrobiellen Zusammensetzung und einzelnen Merkmalen, insbesondere einer signifikanten Abnahme von Akkermansia, Faecalibacterium, Oscillibacter, Alistipes usw., verbunden ist. Darüber hinaus wurde festgestellt, dass T2D mit einem Anstieg von Escherichia/Shigella verbunden ist .
Referenz
Thingholm, LB, et al.„Übergewichtige Personen mit und ohne Typ-2-Diabetes zeigen unterschiedliche mikrobielle Funktionsfähigkeit und Zusammensetzung im Darm.“Zellwirt und Mikrobe26.2 (2019).











