
Metagenomische Sequenzierung – NGS
Servicevorteile
● Isolations- und kultivierungsfrei für die Profilierung mikrobieller Gemeinschaften
● Hohe Auflösung bei der Erkennung von Arten mit geringer Häufigkeit in Umweltproben
● Die Idee des „Meta-“ integriert alle biologischen Merkmale auf Funktionsebene, Artenebene und Genebene, was eine dynamische Sichtweise widerspiegelt, die näher an der Realität ist.
● BMK verfügt mit über 10.000 verarbeiteten Proben über umfangreiche Erfahrung in verschiedenen Probentypen.
Leistungsbeschreibung
| Plattform | Sequenzierung | Empfohlene Daten | Seitenwechsel |
| Illumina NovaSeq-Plattform | PE150 | 6G/10G/20G | 45 Arbeitstage |
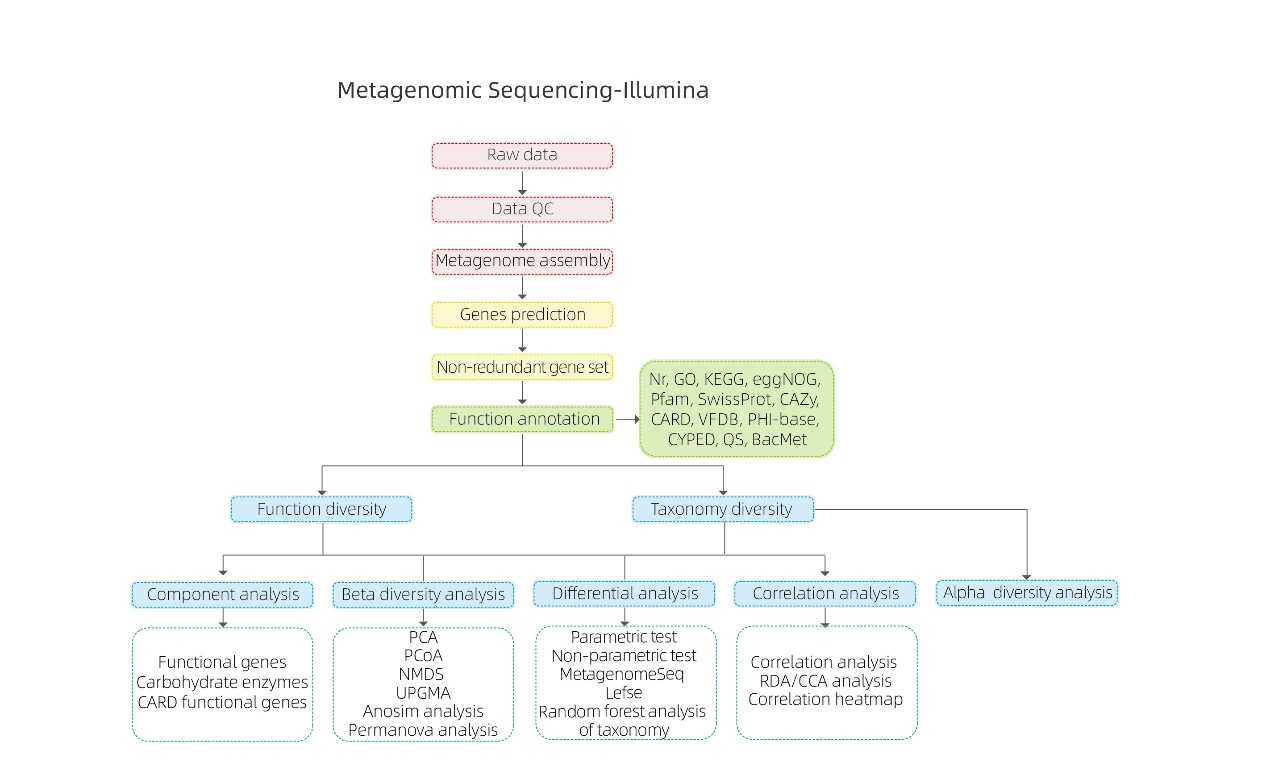
Bioinformatische Analysen
● Qualitätskontrolle der Rohdaten
● Metagenom-Assemblierung
● Nicht-redundanter Gensatz und Annotation
● Analyse der Artenvielfalt
● Analyse der genetischen Funktionsvielfalt
● Intergruppenanalyse
● Assoziationsanalyse anhand experimenteller Faktoren
Musteranforderungen und Lieferung
Probenanforderungen:
FürDNA-Extrakte:
| Beispielstyp | Menge | Konzentration | Reinheit |
| DNA-Extrakte | > 30 ng | > 1 ng/μl | AD260/280= 1,6-2,5 |
Für Umweltproben:
| Beispielstyp | Empfohlenes Probenahmeverfahren |
| Boden | Probenmenge: ca.5 g;Verbleibende verwelkte Substanz muss von der Oberfläche entfernt werden;Große Stücke zermahlen und durch einen 2-mm-Filter passieren;Aliquotierte Proben in sterilen EP-Röhrchen oder Zyrotöhrchen zur Reservierung. |
| Kot | Probenmenge: ca.5 g;Sammeln und aliquotieren Sie Proben zur Reservierung in sterilen EP-Röhrchen oder Kryoröhrchen. |
| Darminhalt | Proben müssen unter aseptischen Bedingungen verarbeitet werden.Gesammeltes Gewebe mit PBS waschen;Zentrifugieren Sie das PBS und sammeln Sie den Niederschlag in EP-Röhrchen. |
| Schlamm | Probenmenge: ca.5 g;Sammeln und aliquotieren Sie die Schlammprobe in einem sterilen EP-Röhrchen oder Kryoröhrchen zur Reservierung |
| Gewässer | Für Proben mit einer begrenzten Menge an Mikroben, wie z. B. Leitungswasser, Brunnenwasser usw., sammeln Sie mindestens 1 l Wasser und lassen Sie es durch einen 0,22-μm-Filter laufen, um die Mikroben auf der Membran anzureichern.Bewahren Sie die Membran in einem sterilen Röhrchen auf. |
| Haut | Kratzen Sie die Hautoberfläche vorsichtig mit einem sterilen Wattestäbchen oder einer chirurgischen Klinge ab und legen Sie sie in ein steriles Röhrchen. |
Empfohlene Musterlieferung
Frieren Sie die Proben 3-4 Stunden lang in flüssigem Stickstoff ein und lagern Sie sie zur Langzeitkonservierung in flüssigem Stickstoff oder bei -80 Grad.Der Probenversand mit Trockeneis ist erforderlich.
Service-Workflow

Musterlieferung

Bibliotheksbau

Sequenzierung

Datenanalyse

Kundendienst
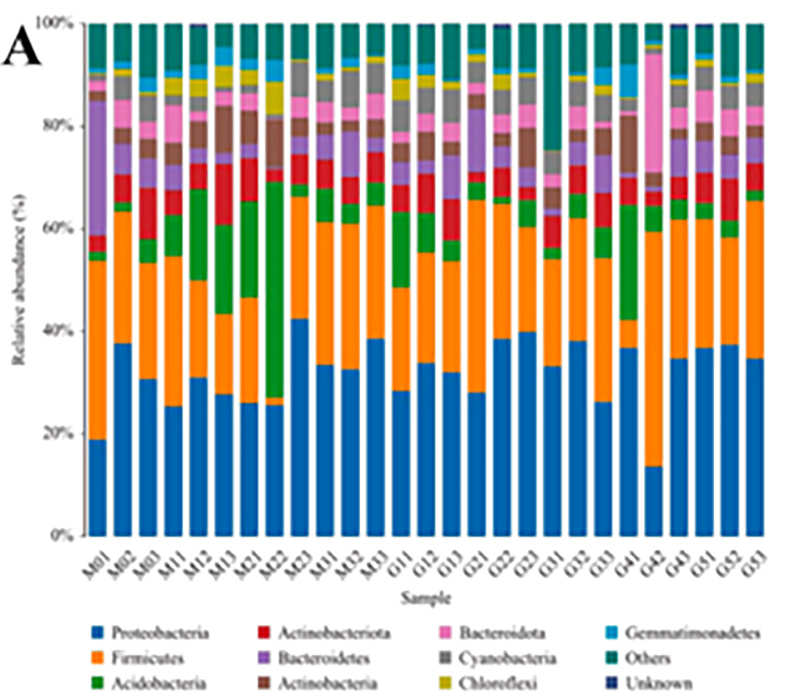
1.Histogramm: Artenverteilung
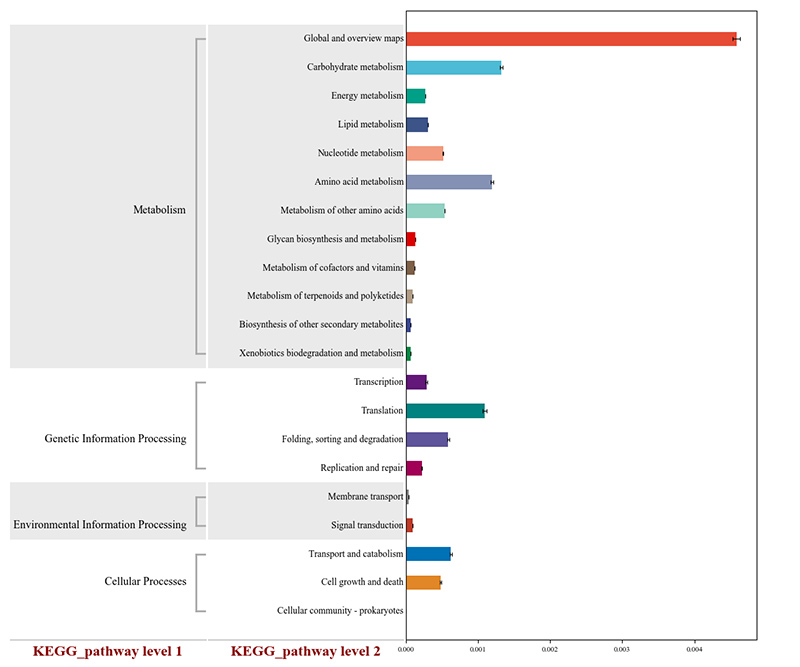
2.Funktionelle Gene, die mit KEGG-Stoffwechselwegen verknüpft sind
3.Heatmap: Differentialfunktionen basierend auf der relativen Genhäufigkeit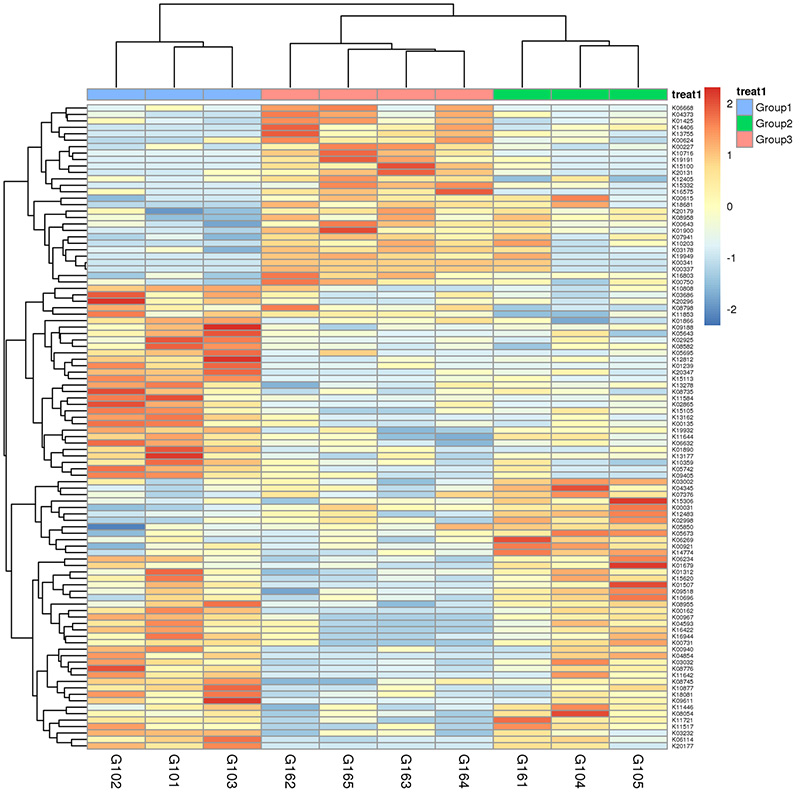 4.Circos von CARD-Antibiotikaresistenzgenen
4.Circos von CARD-Antibiotikaresistenzgenen
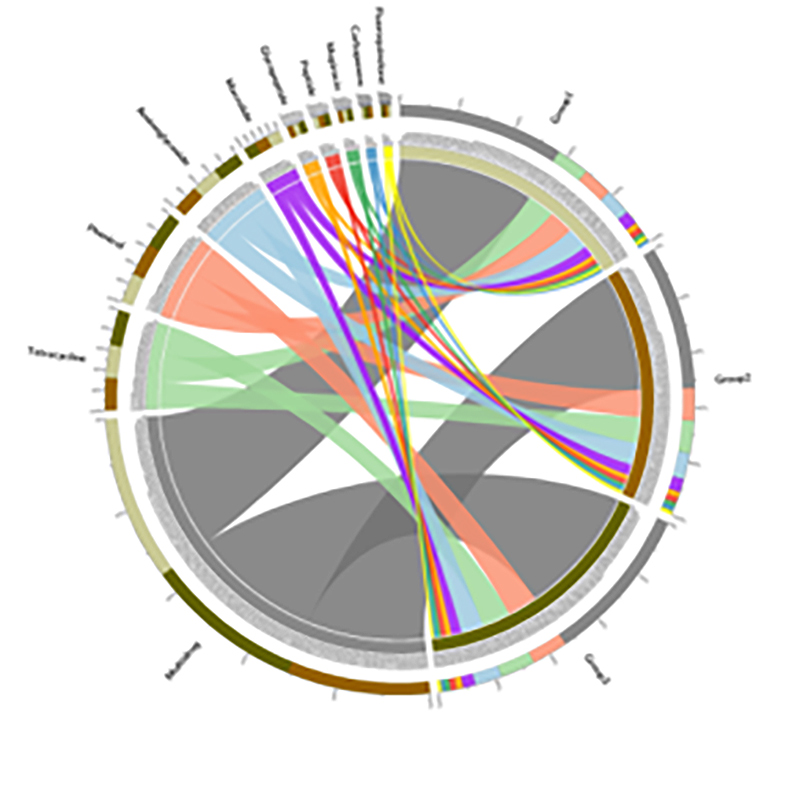
BMK-Fall
Prävalenz von Antibiotikaresistenzgenen und bakteriellen Krankheitserregern entlang des Boden-Mangrovenwurzel-Kontinuums
Veröffentlicht:Journal of Hazardous Materials, 2021
Sequenzierungsstrategie:
Materialien: DNA-Extrakte von vier Fragmenten von Mangrovenwurzel-assoziierten Proben: unbepflanzter Boden, Rhizosphären-, Episphären- und Endosphärenkompartimente
Plattform: Illumina HiSeq 2500
Ziele: Metagenom
16S-rRNA-Gen-V3-V4-Region
Wichtigste Ergebnisse
Metagenomische Sequenzierung und Metabarcoding-Profilierung am Boden-Wurzel-Kontinuum von Mangrovensetzlingen wurden durchgeführt, um die Verbreitung von Antibiotikaresistenzgenen (ARGs) vom Boden in Pflanzen zu untersuchen.Metagenomische Daten zeigten, dass 91,4 % der Antibiotikaresistenzgene in allen vier oben genannten Bodenkompartimenten häufig identifiziert wurden, was eine kontinuierliche Tendenz zeigte.Die 16S-rRNA-Amplikonsequenzierung erzeugte 29.285 Sequenzen, die 346 Arten repräsentierten.In Kombination mit der Artprofilierung durch Amplikonsequenzierung wurde festgestellt, dass diese Verbreitung unabhängig von wurzelassoziierten Mikrobiota erfolgt, sie könnte jedoch durch die Übertragung genetischer Elemente erleichtert werden.Diese Studie identifizierte den Fluss von ARGs und Krankheitserregern vom Boden in die Pflanzen durch ein miteinander verbundenes Boden-Wurzel-Kontinuum.
Referenz
Wang, C., Hu, R., Strong, PJ, Zhuang, W. und Shu, L.(2020).Prävalenz von Antibiotikaresistenzgenen und bakteriellen Krankheitserregern entlang des Boden-Mangrovenwurzel-Kontinuums.Zeitschrift für gefährliche Materialien, 408, 124985.











