Sequenzierung des gesamten Transkriptoms – Illumina
Merkmale
● Duale Bibliothek zur Sequenzierung des gesamten Transkriptoms: rRNA-Depletion, gefolgt von der Vorbereitung der PE150-Bibliothek und Größenauswahl, gefolgt von der Vorbereitung der SE50-Bibliothek
● Vollständige Bioinformatik-Analyse von mRNA, lncRNA, circRNA und miRNA in separaten Bioinformatik-Berichten
● Gemeinsame Analyse der gesamten RNA-Expression in einem kombinierten Bericht, einschließlich ceRNA-Netzwerkanalyse.
Servicevorteile
●Eingehende Analyse regulatorischer Netzwerke: Die ceRNA-Netzwerkanalyse wird durch die gemeinsame Sequenzierung von mRNA, lncRNA, circRNA und miRNA sowie durch einen umfassenden bioinformatischen Workflow ermöglicht.
●Umfassende Anmerkung: Wir verwenden mehrere Datenbanken, um die differentiell exprimierten Gene (DEGs) funktionell zu annotieren und die entsprechende Anreicherungsanalyse durchzuführen, um Einblicke in die zellulären und molekularen Prozesse zu gewinnen, die der Transkriptomantwort zugrunde liegen.
●Umfangreiches Fachwissen: Mit einer Erfolgsbilanz beim erfolgreichen Abschluss von über 2000 Gesamttranskriptomprojekten in verschiedenen Forschungsbereichen bringt unser Team einen großen Erfahrungsschatz in jedes Projekt ein.
●Strenge Qualitätskontrolle: Wir implementieren zentrale Kontrollpunkte in allen Phasen, von der Proben- und Bibliotheksvorbereitung bis hin zur Sequenzierung und Bioinformatik.Diese sorgfältige Überwachung gewährleistet die Lieferung gleichbleibend hochwertiger Ergebnisse.
● Umfassende Anmerkung: Wir verwenden mehrere Datenbanken, um die differentiell exprimierten Gene (DEGs) funktionell zu annotieren und die entsprechende Anreicherungsanalyse durchzuführen, um Einblicke in die zellulären und molekularen Prozesse zu gewinnen, die der Transkriptomantwort zugrunde liegen.
●Post-Sales-Support: Unser Engagement geht über den Projektabschluss hinaus und umfasst einen 3-monatigen Kundendienstzeitraum.Während dieser Zeit bieten wir Projektnachverfolgung, Unterstützung bei der Fehlerbehebung und Frage-und-Antwort-Sitzungen an, um alle Fragen zu den Ergebnissen zu beantworten
Musteranforderungen und Lieferung
| Bibliothek | Sequenzierungsstrategie | Daten empfohlen | Qualitätskontrolle |
| rRNA abgereichert | Illumina PE150 | 16 Gigabyte | Q30≥85 % |
| Größe ausgewählt | Illumina SE50 | 10–20 Millionen Lesevorgänge |
Probenanforderungen:
Nukleotide:
| Konz. (ng/μl) | Menge (μg) | Reinheit | Integrität |
| ≥ 100 | ≥ 1 | OD260/280=1,7-2,5 OD260/230=0,5-2,5 Begrenzte oder keine Protein- oder DNA-Kontamination auf dem Gel sichtbar. | Pflanzen: RIN≥6,5 Tier: RIN≥7,0 5,0≥28S/18S≥1,0; begrenzte oder keine Grundlinienhöhe |
Empfohlene Musterlieferung
Container:
2 ml Zentrifugenröhrchen (Alufolie wird nicht empfohlen)
Probenbeschriftung: Gruppe+Replikation, z. B. A1, A2, A3;B1, B2, B3... ...
Sendung:
1. Trockeneis: Proben müssen in Beutel verpackt und in Trockeneis vergraben werden.
2.RNAstable-Röhrchen: RNA-Proben können in RNA-Stabilisierungsröhrchen (z. B. RNAstable®) getrocknet und bei Raumtemperatur versendet werden.
Service-Workflow

Experimentdesign

Musterlieferung

RNA-Extraktion

Bibliotheksbau

Sequenzierung

Datenanalyse

Kundendienst
Bioinformatik
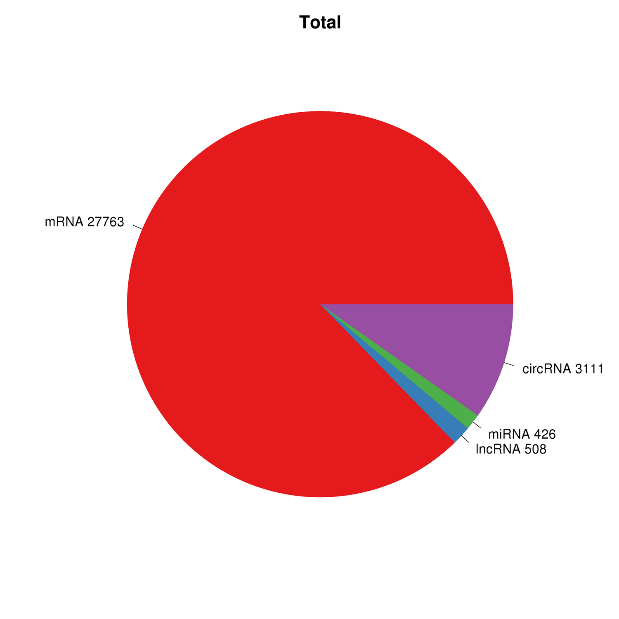
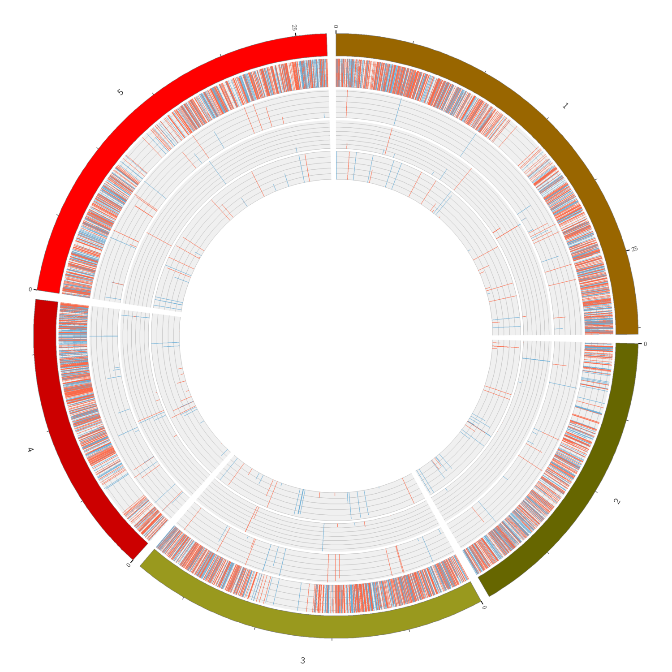
Übersicht über die RNA-Expression
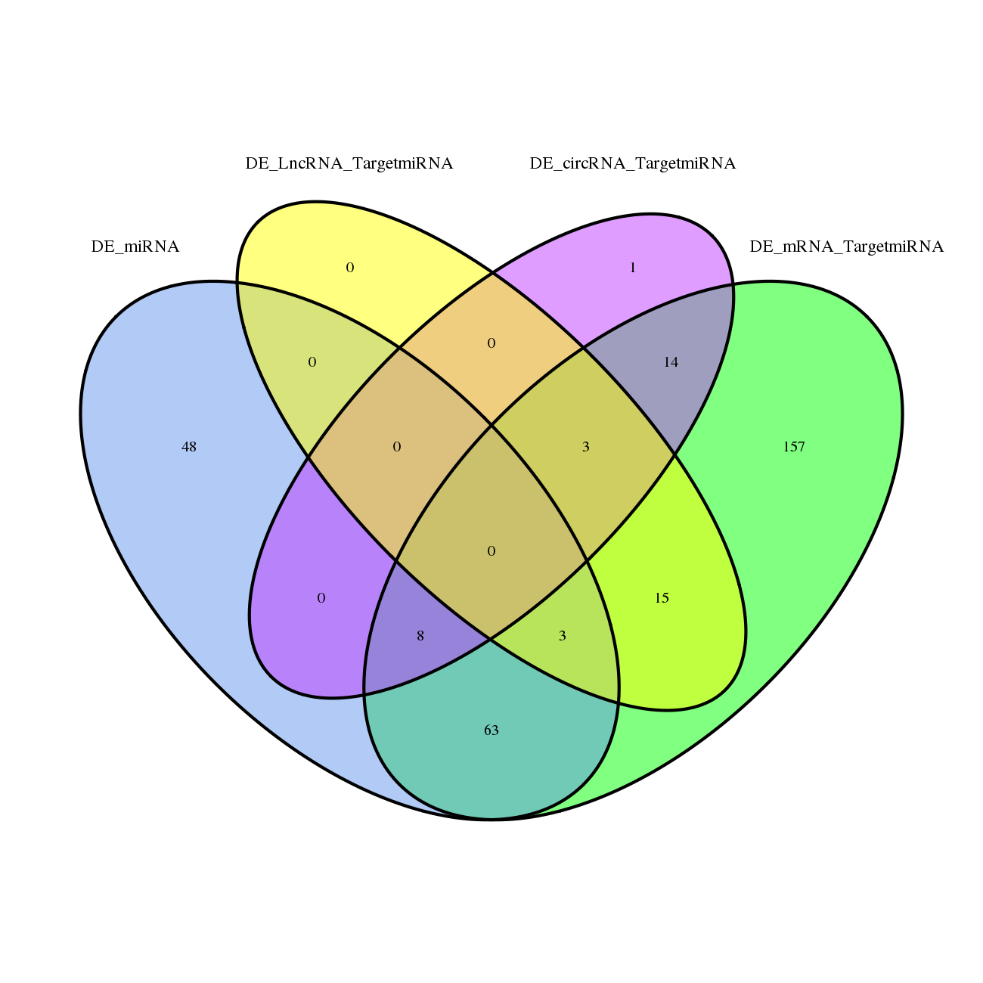
Differenziell exprimierte Gene
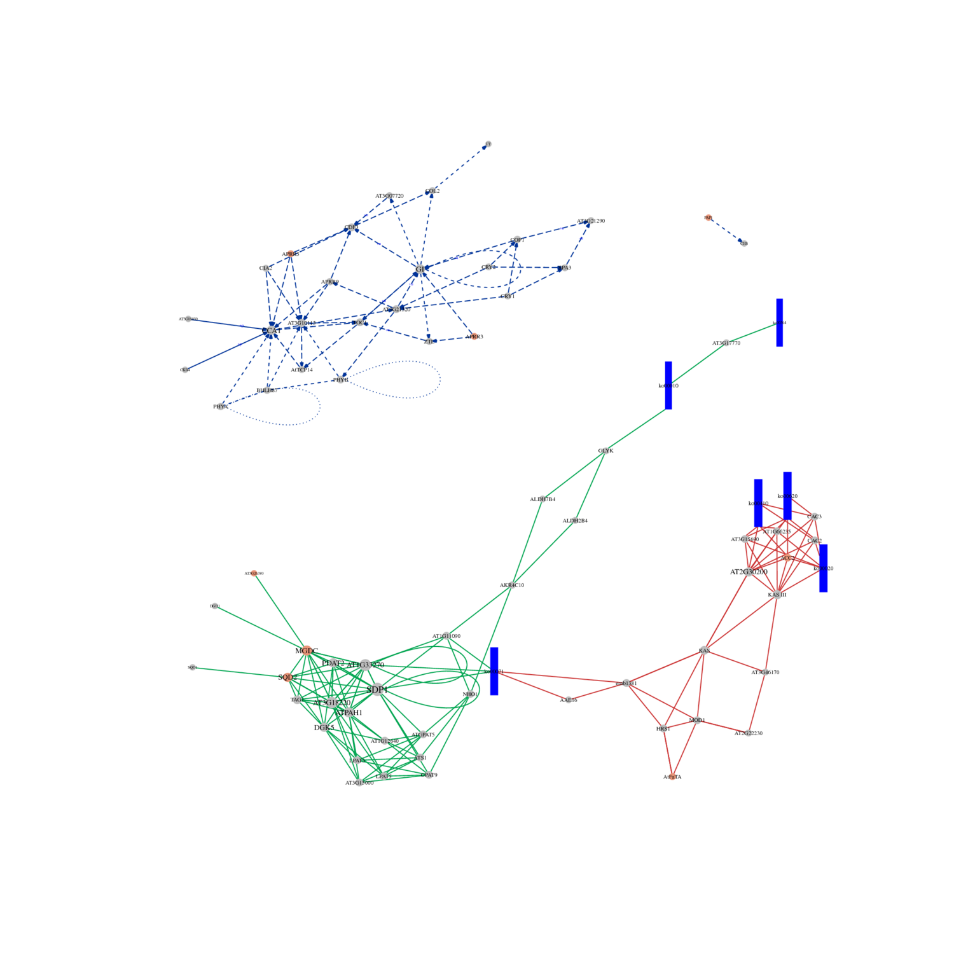
ceRNA-Analyse
Entdecken Sie die Forschungsfortschritte, die durch die Sequenzierungsdienste für das gesamte Transkriptom von BMKGene ermöglicht werden, anhand einer kuratierten Sammlung von Veröffentlichungen.
Dai, Y. et al.(2022) „Umfassende Expressionsprofile von mRNAs, lncRNAs und miRNAs bei der Kashin-Beck-Krankheit, identifiziert durch RNA-Sequenzierung“, Molecular Omics, 18(2), S. 154–166.doi: 10.1039/D1MO00370D.
Liu, N. nan et al.(2022) „Transkriptomanalyse in voller Länge der Kälteresistenz von Apis cerana im Changbai-Gebirge während der Überwinterungszeit.“, Gene, 830, S. 146503–146503.doi: 10.1016/J.GENE.2022.146503.
Wang, XJ et al.(2022) „Multi-Omics Integration-Based Prioritization of Competing Endogenous RNA Regulation Networks in Small Cell Lung Cancer: Molecular Characteristics and Drug Candidates“, Frontiers in Oncology, 12, S.904865. doi: 10.3389/FONC.2022.904865/BIBTEX.
Xu, P. et al.(2022) „Die integrierte Analyse der lncRNA/circRNA-miRNA-mRNA-Expressionsprofile enthüllt neue Einblicke in mögliche Mechanismen als Reaktion auf Wurzelknotennematoden in Erdnüssen“, BMC Genomics, 23(1), S. 1–12.doi: 10.1186/S12864-022-08470-3/FIGURES/7.
Yan, Z. et al.(2022) „Die RNA-Sequenzierung des gesamten Transkriptoms beleuchtet die molekularen Mechanismen, die mit der Aufrechterhaltung der Nacherntequalität von Brokkoli durch rote LED-Bestrahlung verbunden sind“, Postharvest Biology and Technology, 188, S.111878. doi: 10.1016/J.POSTHARVBIO.2022.111878.