
Secuenciación de amplicones 16S/18S/ITS-NGS
Ventajas del servicio
● Identificación rápida y sin aislamiento de la composición microbiana en muestras ambientales
● Alta resolución en componentes poco abundantes en muestras ambientales.
● Último flujo de análisis QIIME2 con diversos análisis en términos de base de datos, anotaciones, OTU/ASV.
● Alto rendimiento, mayor precisión
● Aplicable a diversos estudios de comunidades microbianas.
● BMK posee una amplia experiencia con más de 100.000 muestras al año, que abarcan suelo, agua, gas, lodos, heces, intestinos, piel, caldo de fermentación, insectos, plantas, etc.
● BMKCloud facilitó la interpretación de datos que contiene 45 herramientas de análisis personalizadas.
Especificaciones de servicio
| SecuenciaciónPlataforma | Biblioteca | Rendimiento de datos recomendado | Tiempo estimado de respuesta |
| Plataforma Illumina NovaSeq | PE250 | Etiquetas 50K/100K/300K | 30 dias |
Análisis bioinformáticos.
● Control de calidad de los datos sin procesar
● Agrupación OTU/eliminación de ruido (ASV)
● Anotación OTU
● Diversidad alfa
● Diversidad beta
● Análisis intergrupal
● Análisis de asociación frente a factores experimentales.
● Función de predicción de genes.
Requisitos de muestra y entrega
Requisitos de muestra:
Paraextractos de ADN:
| Tipo de ejemplo | Cantidad | Concentración | Pureza |
| extractos de ADN | > 30 ng | > 1 ng/μl | DO260/280= 1,6-2,5 |
Para muestras ambientales:
| Tipo de ejemplo | Procedimiento de muestreo recomendado |
| Suelo | Cantidad de muestra: aprox.5 gramos;Es necesario eliminar de la superficie los restos de sustancia marchita;Moler trozos grandes y pasar por filtro de 2 mm;Muestras alícuotas en tubo EP o criotubo estéril para reserva. |
| Heces | Cantidad de muestra: aprox.5 gramos;Recoja y haga alícuotas de muestras en un tubo EP o criotubo estéril para reservarlas. |
| Contenido intestinal | Las muestras deben procesarse en condiciones asépticas.Lavar el tejido recogido con PBS;Centrifugar el PBS y recoger el precipitante en tubos EP. |
| Lodo | Cantidad de muestra: aprox.5 gramos;Recoger y tomar alícuotas de la muestra de lodo en un tubo EP o criotubo estéril para reservar |
| Agua corporal | Para muestras con una cantidad limitada de microbios, como agua del grifo, agua de pozo, etc., recolecte al menos 1 litro de agua y páselo a través de un filtro de 0,22 μm para enriquecer los microbios en la membrana.Guarde la membrana en un tubo estéril. |
| Piel | Raspe con cuidado la superficie de la piel con un hisopo de algodón esterilizado o un bisturí quirúrgico y colóquelo en un tubo esterilizado. |
Entrega de muestra recomendada
Congele las muestras en nitrógeno líquido durante 3 a 4 horas y guárdelas en nitrógeno líquido o a -80 grados para una reserva a largo plazo.Se requiere envío de muestras con hielo seco.
Flujo de trabajo del servicio

Entrega de muestra

construcción de biblioteca

Secuenciación

Análisis de los datos

Servicios postventa
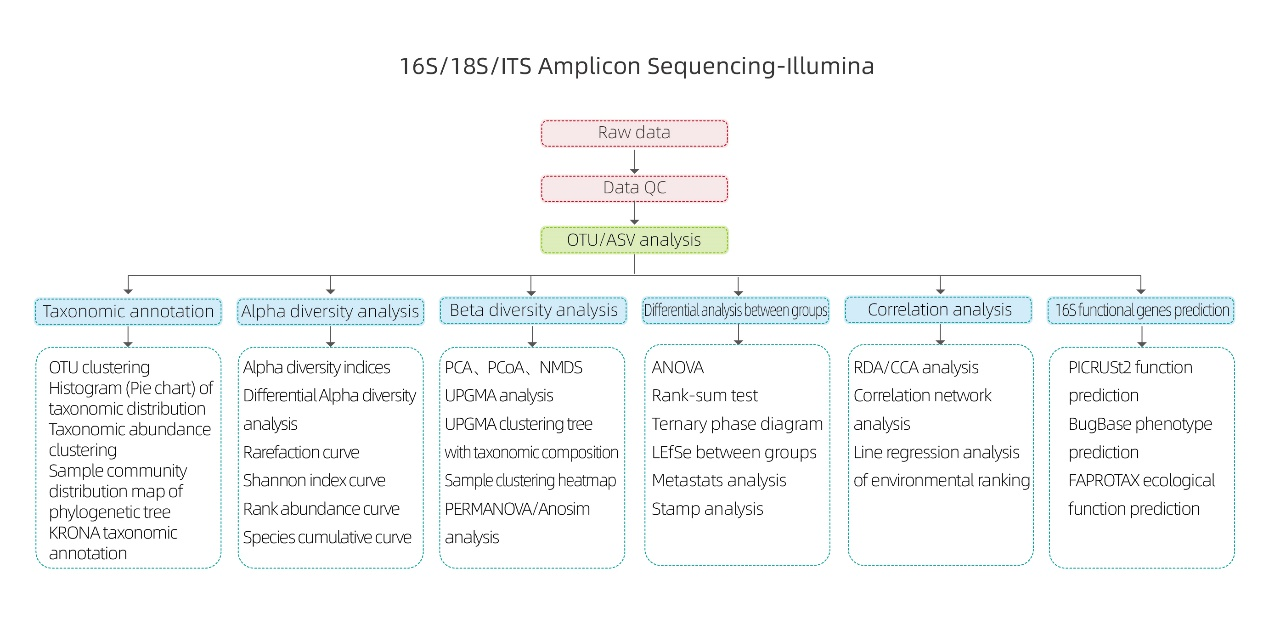
1.Distribución de especies
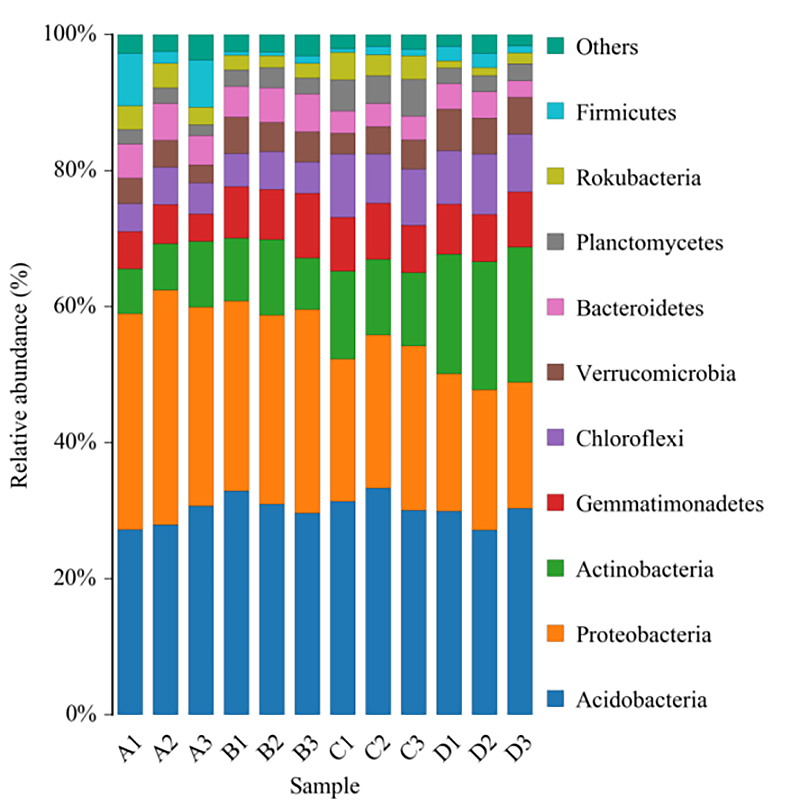
2.Mapa de calor: agrupación de riqueza de especies
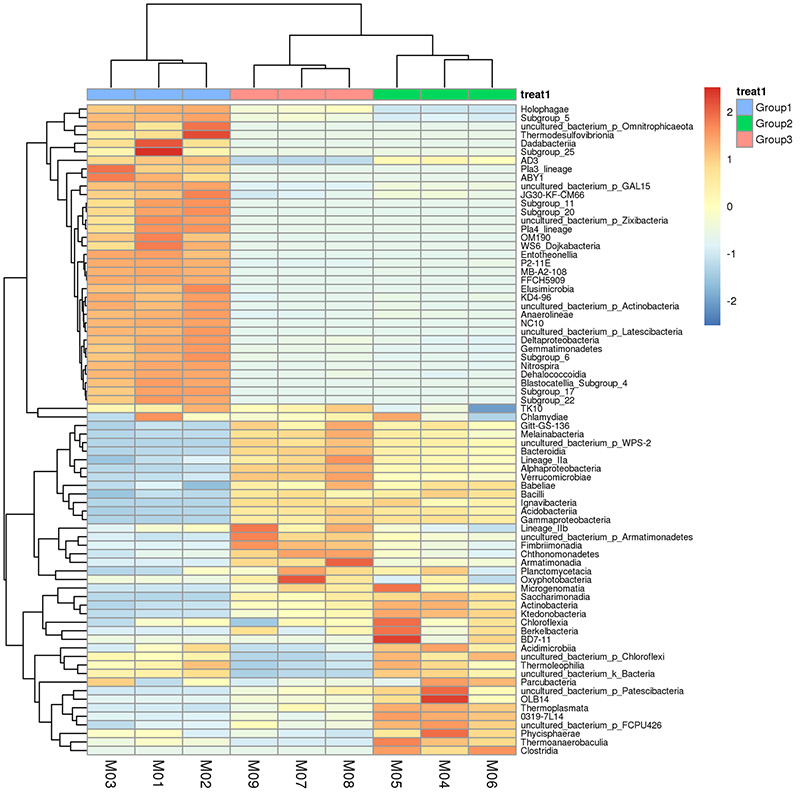
3.Curva de facción rara
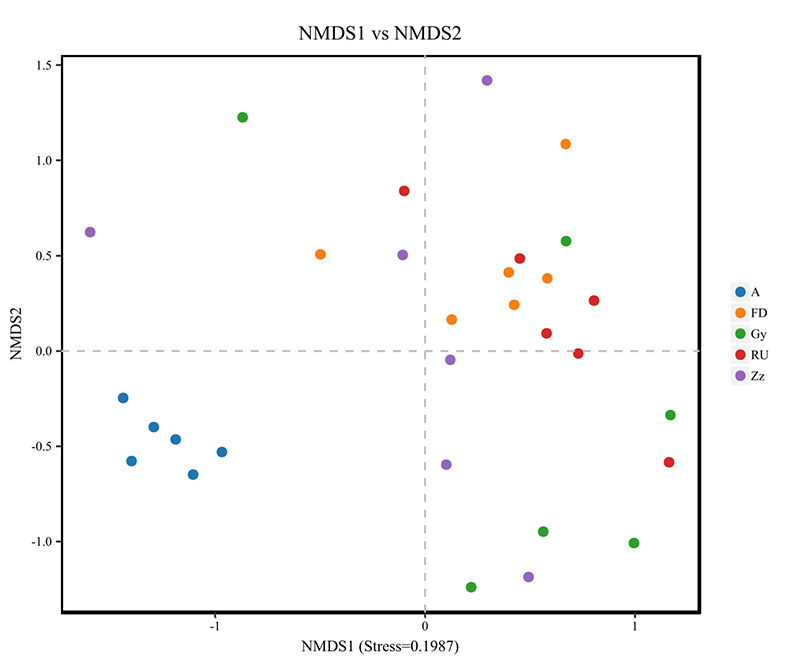
4.Análisis NMDS
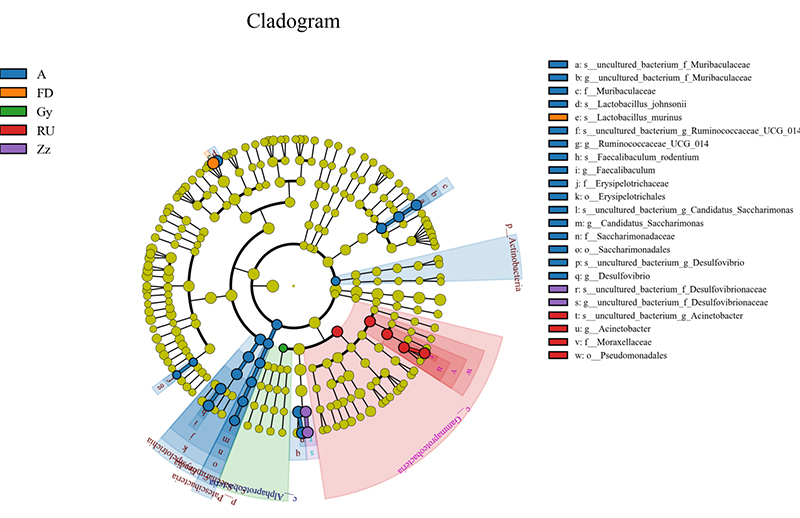
5.Análisis de izquierda
Caso BMK
Las personas obesas con y sin diabetes tipo 2 muestran diferentes capacidades y composiciones funcionales microbianas intestinales
Publicado:Huésped celular y microbio, 2019
Estrategia de secuenciación:
Personas magras sin diabetes (n=633);Obesos no diabéticos (n=494);Diabetes obesa tipo 2 (n = 153);
Región objetivo: 16S rDNA V1-V2
Plataforma: Illumina Miseq (secuenciación de amplicones basada en NGS)
Un subconjunto de extractos de ADN se sometió a secuenciación metagenómica en Illumina Hiseq
Resultados clave
Se diferenciaron con éxito los perfiles microbianos de estas enfermedades metabólicas.
Al comparar las características microbianas generadas por la secuenciación 16S, se encontró que la obesidad se asocia con cambios en la composición microbiana, características individuales, especialmente una disminución significativa de Akkermansia, Faecalibacterium, Oscillibacter, Alistipes, etc. Además, se encontró que la diabetes tipo 2 se asocia con un aumento de Escherichia/shigella. .
Referencia
Thingholm, LB y col."Las personas obesas con y sin diabetes tipo 2 muestran diferente capacidad funcional y composición microbiana intestinal".Huésped celular y microbio26.2 (2019).











