Secuenciación de circRNA-Illumina
Características
● Agotamiento del ARNr seguido de la preparación de la biblioteca direccional, lo que permite secuenciar datos específicos de cada cadena.
● El flujo de trabajo bioinformático permite la predicción y cuantificación de la expresión del ARN circular
Ventajas del servicio
●Bibliotecas de ARN más completas:Utilizamos el agotamiento del ARNr en lugar del agotamiento lineal del ARN en nuestra preparación previa a la biblioteca, lo que garantiza que los datos de secuenciación incluyan no solo el ARNcirc, sino también el ARNm y el ARNlnc, lo que permite el análisis conjunto de estos conjuntos de datos.
●Análisis opcional de redes competitivas de ARN endógeno (ARNce): proporcionando conocimientos más profundos sobre los mecanismos reguladores celulares
●Amplia experiencia: con un historial de procesamiento de más de 20 000 muestras en BMK, que abarca diversos tipos de muestras y proyectos de lncRNA, nuestro equipo aporta una gran experiencia a cada proyecto.
●Control de calidad riguroso: implementamos puntos de control centrales en todas las etapas, desde la preparación de muestras y bibliotecas hasta la secuenciación y la bioinformática.Este seguimiento meticuloso garantiza la entrega de resultados consistentes de alta calidad.
● Soporte posventa: Nuestro compromiso se extiende más allá de la finalización del proyecto con un período de servicio postventa de 3 meses.Durante este tiempo, ofrecemos seguimiento del proyecto, asistencia para la resolución de problemas y sesiones de preguntas y respuestas para abordar cualquier consulta relacionada con los resultados.
Requisitos de muestra y entrega
| Biblioteca | Plataforma | Datos recomendados | Control de calidad de datos |
| Poli A enriquecido | Iluminación PE150 | 16-20 GB | Q30≥85% |
Requisitos de muestra:
Nucleótidos:
| Conc.(ng/μl) | Cantidad (μg) | Pureza | Integridad |
| ≥ 100 | ≥ 0,5 | DO260/280=1,7-2,5 DO260/230=0,5-2,5 En el gel se muestra contaminación limitada o nula de proteínas o ADN. | Para plantas: RIN≥6,5; Para animales: RIN≥7,0; 5,0≥28S/18S≥1,0; elevación basal limitada o nula |
● Plantas:
Raíz, Tallo o Pétalo: 450 mg
Hoja o Semilla: 300 mg
Fruta: 1,2 gramos
● Animales:
Corazón o Intestino: 450 mg
Vísceras o Cerebro: 240 mg
Músculo: 600 mg
Huesos, Cabello o Piel: 1,5g
● Artrópodos:
Insectos: 9g
Crustáceos: 450 mg
● Sangre entera:2 tubos
● Celdas: 106 células
● Suero y Plasma: 6ml
Entrega de muestra recomendada
Envase: tubo de centrífuga de 2 ml (no se recomienda papel de aluminio)
Etiquetado de muestras: Grupo+réplica, por ejemplo, A1, A2, A3;B1, B2, B3... ...
Envío:
1. Hielo seco: las muestras deben empaquetarse en bolsas y enterrarse en hielo seco.
2. Tubos RNAstable: las muestras de ARN se pueden secar en un tubo de estabilización de ARN (por ejemplo, RNAstable®) y enviarse a temperatura ambiente.
Flujo de trabajo del servicio

Diseño de experimentos

Entrega de muestra

extracción de ARN

construcción de biblioteca

Secuenciación

Análisis de los datos

Servicios postventa
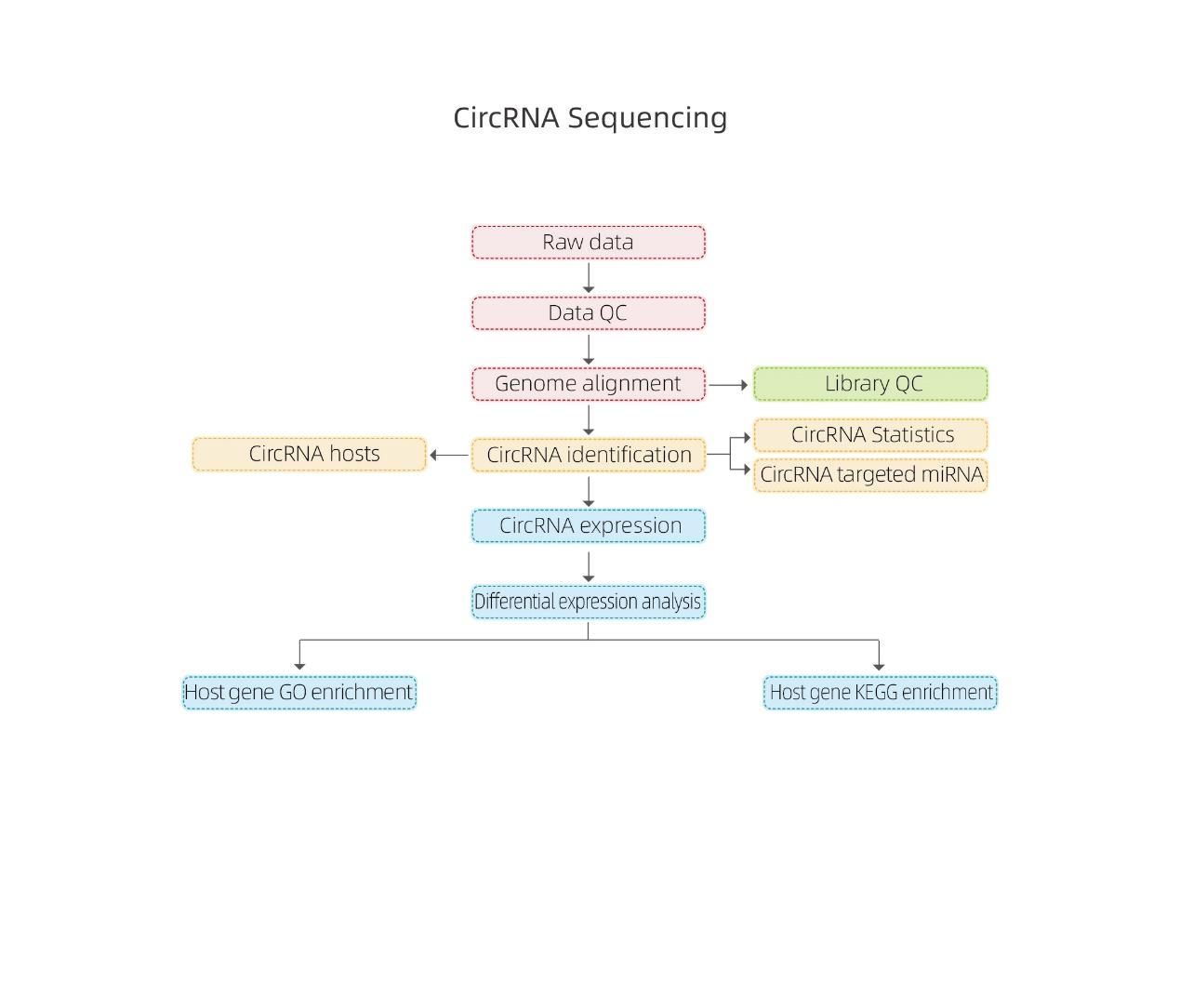
Bioinformática
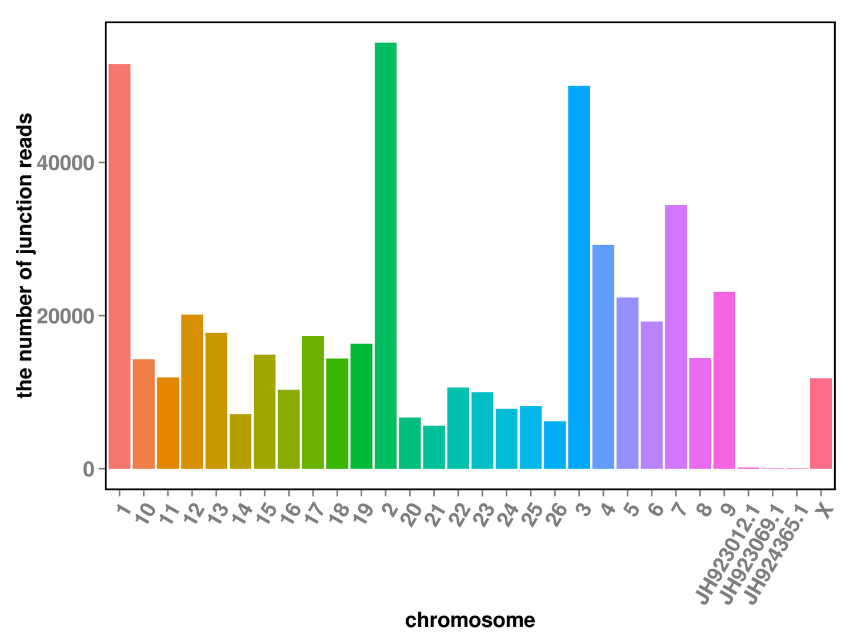
Predicción de circRNA: distribución cromosómica
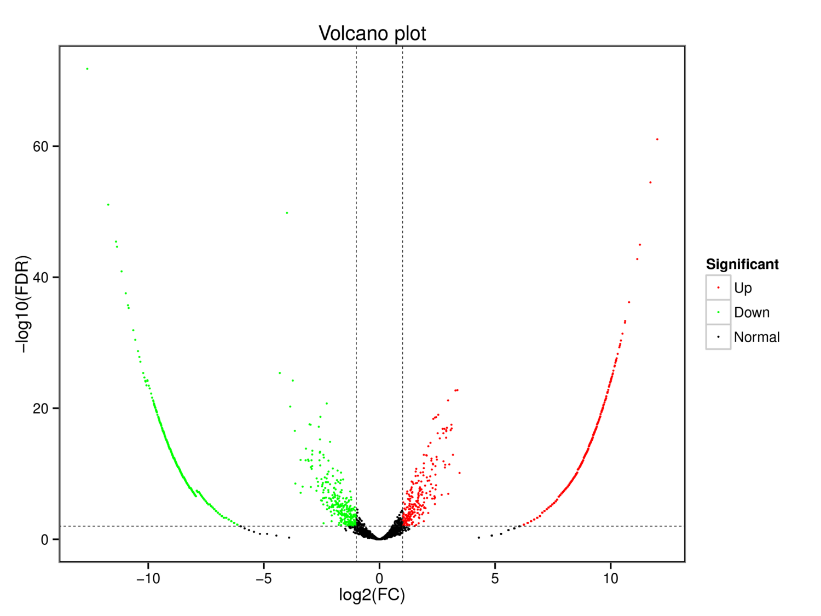
CirRNA expresados diferencialmente: diagrama de volcán
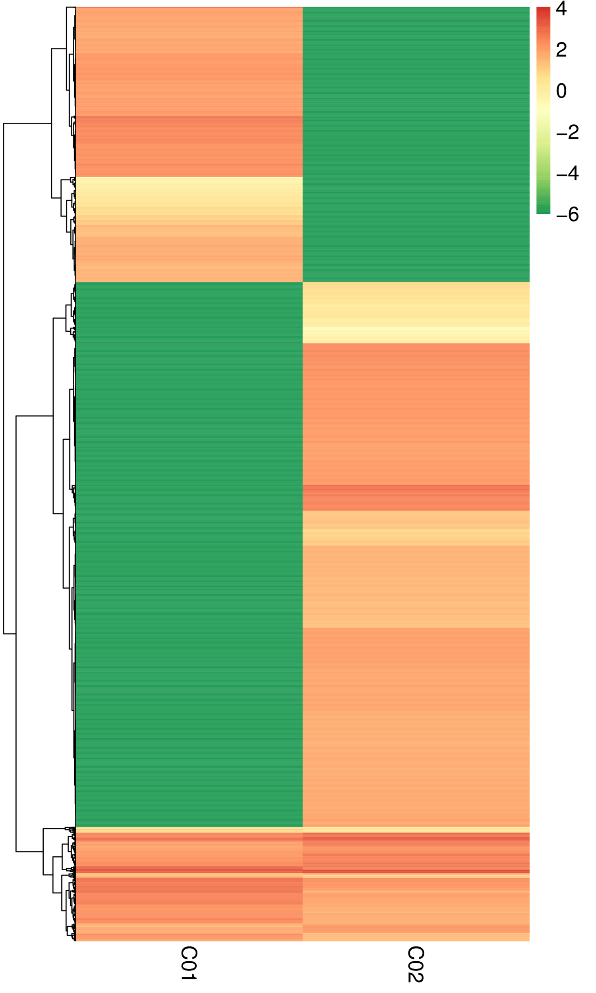
CirRNA expresados diferencialmente: agrupación jerárquica
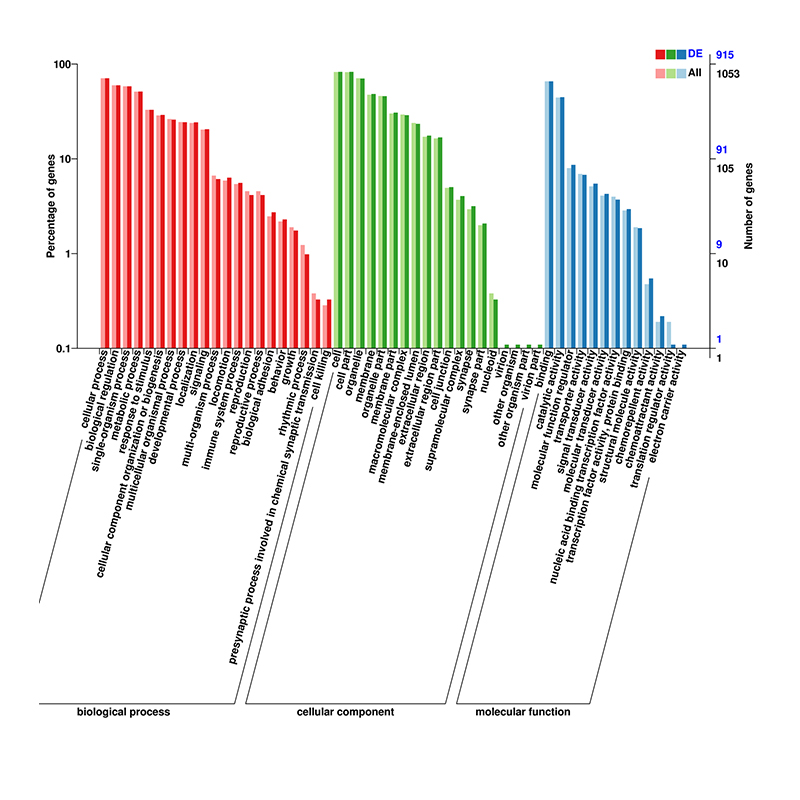
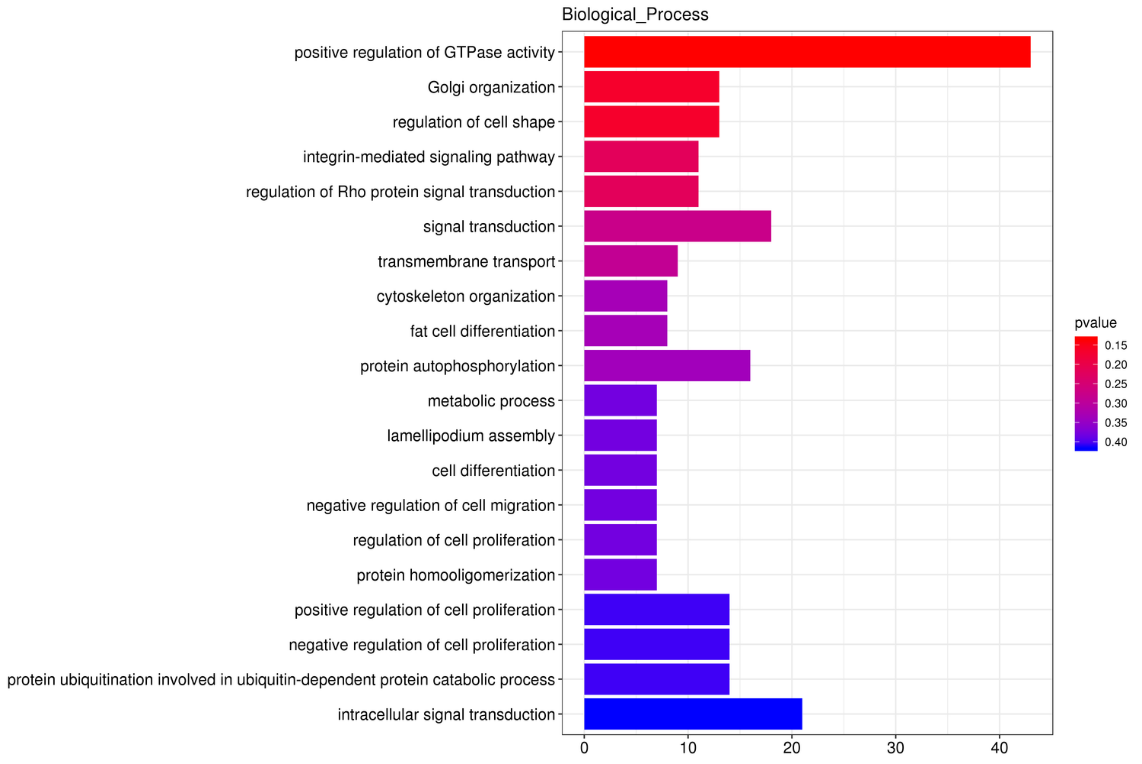
Enriquecimiento funcional de los genes del huésped de circRNA.
Explore los avances en la investigación facilitados por los servicios de secuenciación de circRNA de BMKGene a través de una colección seleccionada de publicaciones.
Wang, X. et al.(2021) 'CPSF4 regula la formación de circRNA y el silenciamiento de genes mediado por microRNA en el carcinoma hepatocelular', Oncogene 2021 40:25, 40(25), págs.doi: 10.1038/s41388-021-01867-6.
Xia, K. y col.(2023) 'El transcriptoma sensible a X oo revela el papel del ARN133 circular en la resistencia a enfermedades mediante la regulación de la expresión de OsARAB en el arroz', Phytopathology Research, 5 (1), págs.doi: 10.1186/S42483-023-00188-8/FIGURAS/6.
Y, H. et al.(2023) 'CPSF3 modula el equilibrio de transcripciones circulares y lineales en el carcinoma hepatocelular'.doi: 10.21203/RS.3.RS-2418311/V1.
Zhang, Y. et al.(2023) 'Evaluación integral de los ARNcirc en la miocardiopatía cirrótica antes y después del trasplante de hígado', Inmunofarmacología internacional, 114, p.109495. doi: 10.1016/J.INTIMP.2022.109495.