Secuenciación de ARNm de eucariotas-Illumina
Ventajas
● Alta experiencia: se han procesado más de 200.000 muestras en BMK que cubren diversos tipos de muestras, incluidos cultivos celulares, tejidos, fluidos corporales, etc. y se han cerrado más de 7.000 proyectos de mRNA-Seq que cubren diversas áreas de investigación.
● Estricto sistema de control de calidad: los puntos centrales de control de calidad en todos los pasos, incluida la preparación de muestras, la preparación de bibliotecas, la secuenciación y la bioinformática, están bajo estrecha supervisión para ofrecer resultados de alta calidad.
● Múltiples bases de datos disponibles para anotación de funciones y estudios de enriquecimiento para cumplir diversos objetivos de investigación.
● Servicios posventa: Servicios posventa válidos por 3 meses una vez finalizado el proyecto, incluido seguimiento de proyectos, resolución de problemas, preguntas y respuestas sobre resultados, etc.
Requisitos de muestra y entrega
| Biblioteca | Estrategia de secuenciación | Datos recomendados | Control de calidad |
| Poli A enriquecido | Iluminación PE150 | 6GB | Q30≥85% |
Requisitos de muestra:
Nucleótidos:
| Conc.(ng/μl) | Cantidad (μg) | Pureza | Integridad |
| ≥ 20 | ≥ 0,5 | DO260/280=1,7-2,5 DO260/230=0,5-2,5 En el gel se muestra contaminación limitada o nula de proteínas o ADN. | Para plantas: RIN≥6,5; Para animales: RIN≥7,0; 5,0≥28S/18S≥1,0; elevación basal limitada o nula |
Tejido: Peso (seco):≥1 gramos
*Para tejido de menos de 5 mg, recomendamos enviar una muestra de tejido congelada instantáneamente (en nitrógeno líquido).
Suspensión celular:Recuento de células = 3×106- 1×107
*Recomendamos enviar lisado celular congelado.En caso de que esa celda cuente con menos de 5×105.
Muestras de sangre:Volumen≥1 ml
Microorganismo:Masa ≥ 1 g
Entrega de muestra recomendada
Envase: tubo de centrífuga de 2 ml (no se recomienda papel de aluminio)
Etiquetado de muestras: Grupo+réplica, por ejemplo, A1, A2, A3;B1, B2, B3... ...
Envío:
- Hielo seco: las muestras deben empaquetarse en bolsas y enterrarse en hielo seco.
- Tubos RNAstable: las muestras de ARN se pueden secar en un tubo de estabilización de ARN (por ejemplo, RNAstable®) y enviarse a temperatura ambiente.
Flujo de trabajo del servicio

Diseño de experimentos

Entrega de muestra

extracción de ARN

construcción de biblioteca

Secuenciación

Análisis de los datos

Servicios postventa
Bioinformática
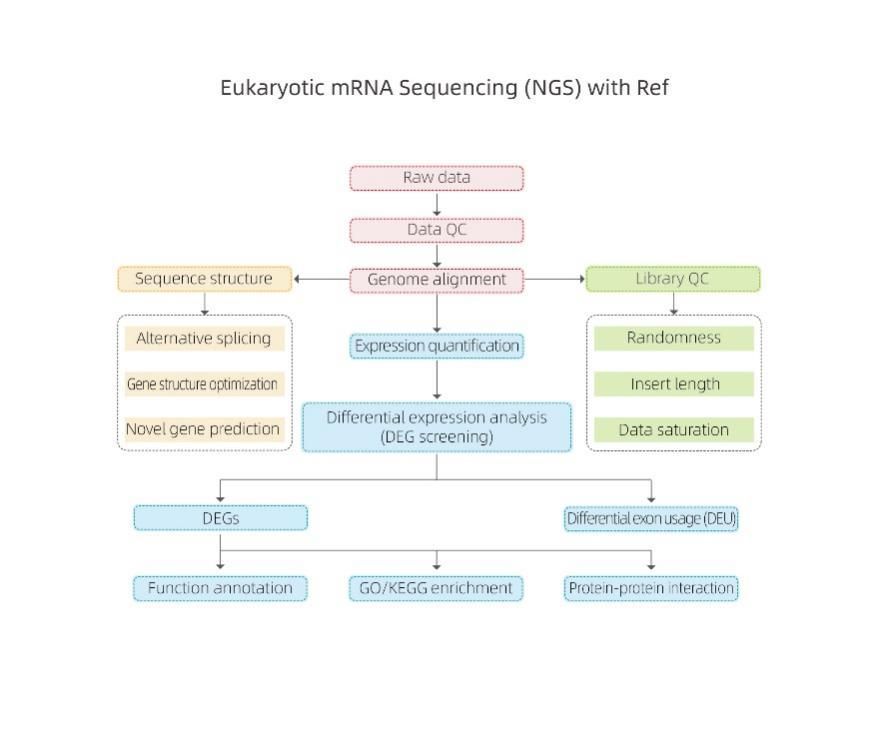
eucariota Flujo de trabajo de análisis de secuenciación de ARNm
Bioinformática
ØControl de calidad de datos sin procesar
ØAlineación del genoma de referencia.
ØAnálisis de la estructura de la transcripción.
ØCuantificación de expresiones
ØAnálisis de expresión diferencial.
ØAnotación y enriquecimiento de funciones.
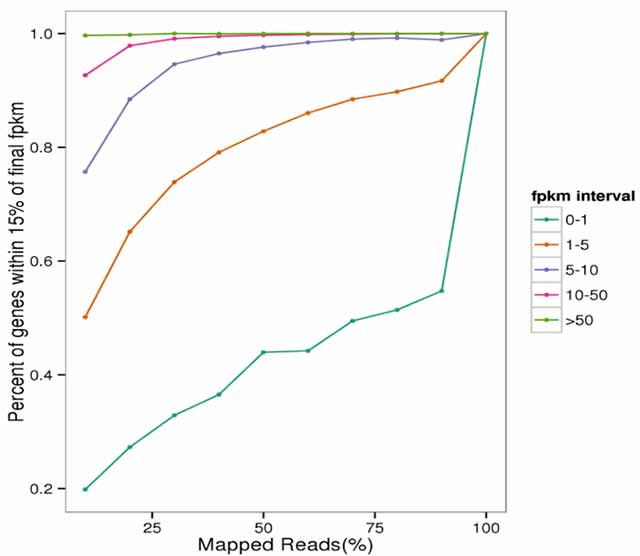
1.Curva de saturación de datos de ARNm
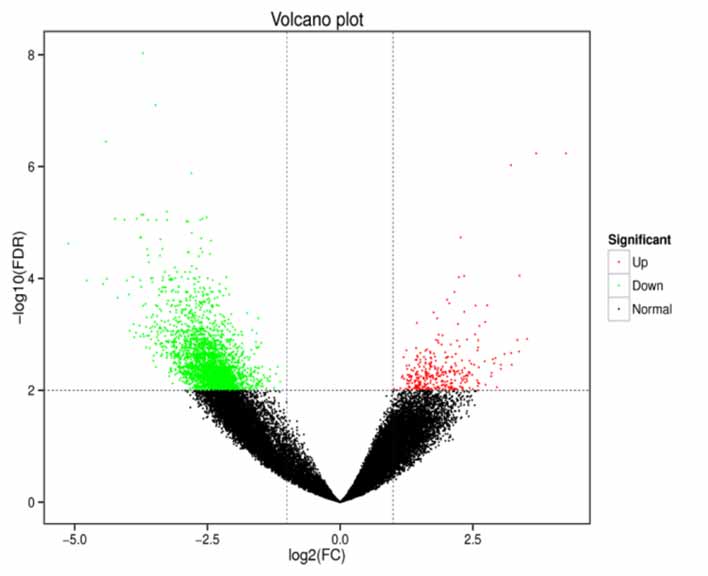
2.Análisis de expresión diferencial-gráfico de volcán.
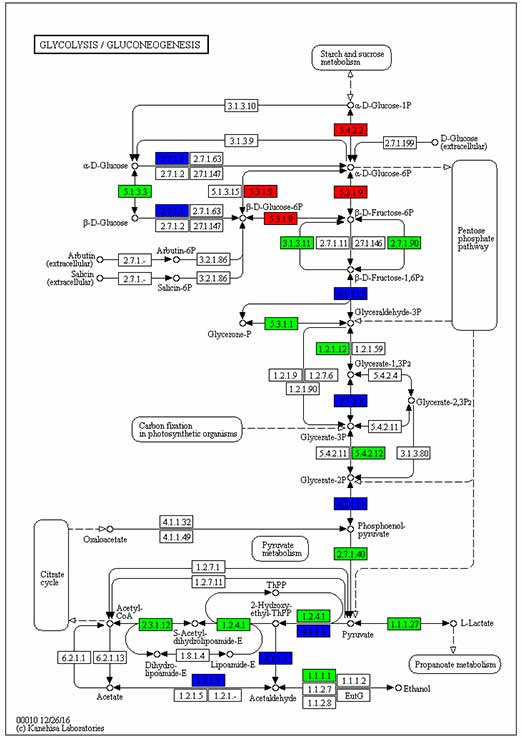
3.Anotación KEGG en DEG
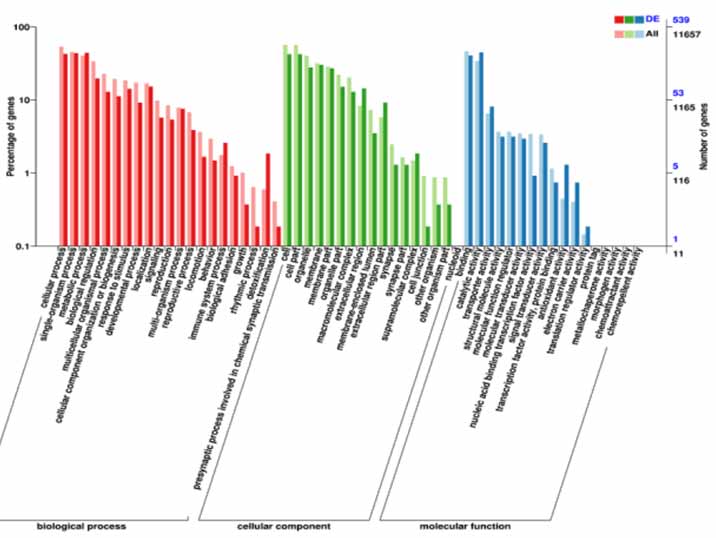
4.Clasificación GO en DEG