TRANSCRIPTOMICA
naturaleza
COMUNICACIONES
La caracterización completa de la transcripción de la mutación SF3B1 en la leucemia linfocítica crónica revela una regulación negativa de los intrones retenidos
Transcripciones completas |Secuenciación de nanoporos|Análisis de isoformas alternativas
Fondo
SSe ha informado ampliamente que las mutaciones omáticas en el factor de empalme SF3B1 se asocian con varios cánceres, incluida la leucemia linfocítica crónica (LLC), el melanoma uveal, el cáncer de mama, etc. Además, los estudios transcriptómicos de lectura corta han revelado patrones de empalme aberrantes inducidos por mutaciones SF3B1.Sin embargo, los estudios sobre estos patrones de empalme alternativos se han limitado durante mucho tiempo al nivel de evento y la falta de conocimiento a nivel de isoforma debido a la limitación de transcripciones ensambladas de lectura corta.Aquí, se introdujo una plataforma de secuenciación de nanoporos para generar transcripciones completas, lo que permitió la investigación sobre isoformas de AS.
Diseño experimental
experimentos
Agrupamiento:1. CLL-SF3B1(WT) 2. CLL-SF3B1(mutación K700E);3. Células B normales
Estrategia de secuenciación:Secuenciación de la biblioteca MinION 2D, secuenciación de la biblioteca PromethION 1D;datos de lectura corta de las mismas muestras
Plataforma de secuenciación:ONT Minión;ONT PromethION;
Análisis Bioinformático
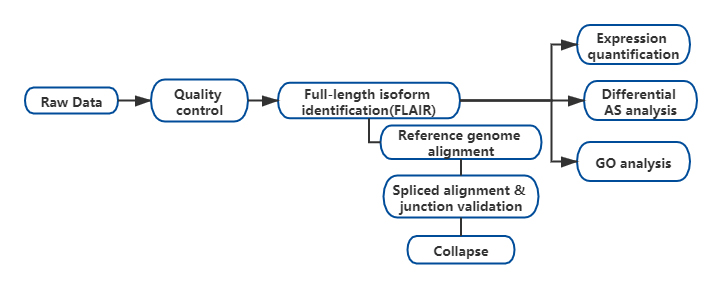
Resultados
ASe generaron un total de 257 millones de lecturas a partir de 6 muestras de CLL y 3 células B.En promedio, el 30,5% de estas lecturas se identificaron como transcripciones completas.
FSe desarrolló un análisis de isoformas alternativas de longitud completa del ARN (FLAIR) para generar un conjunto de isoformas de alta confianza.FLAIR se puede resumir como:
Nanopore lee la alineación: identifica la estructura general de la transcripción basada en el genoma de referencia;
Scorrección de unión de empalme: errores de secuencia correctos (rojo) con sitio de empalme de intrones anotados, intrones de datos de lectura corta o ambos;
Collapse: resume las isoformas representativas basadas en cadenas de unión de empalme (conjunto de primer paso).Seleccione una isodesde de alta confianza según la cantidad de lecturas de respaldo (Umbral: 3).
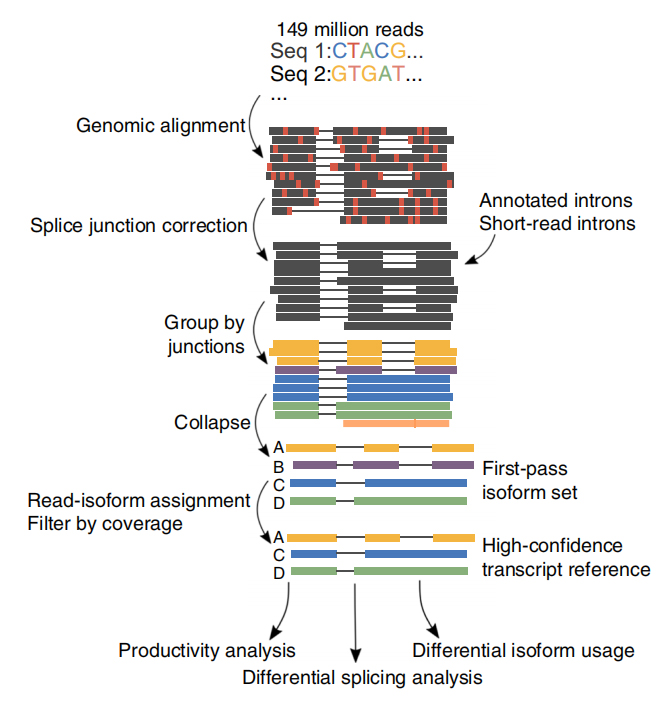
Figura 1. Análisis FLAIR para identificar isoformas completas asociadas con la mutación SF3B1 en la LLC
FLAIR identificó 326.699 isoformas empalmadas de alta confianza, el 90% de las cuales son isoformas novedosas.Se descubrió que la mayoría de estas isoformas no anotadas eran combinaciones novedosas de uniones de empalme conocidas (142,971), mientras que el resto de las isoformas novedosas contenían intrones retenidos (21,700) o exones novedosos (3594).
LLas secuencias de lectura larga permiten la identificación de sitios de empalme alterados por SF3B1-K700E mutante a nivel de isoforma.Se encontró que 35 3'SS alternativos y 10 5'SS alternativos estaban empalmados de manera significativamente diferencial entre SF3B1-K700E y SF3B1-WT.33 de las 35 alteraciones fueron descubiertas recientemente mediante secuencias de lectura larga.En los datos de Nanopore, la distribución de la distancia entre los 3'SS alterados por SF3B1-K700E y los picos de los sitios canónicos es de alrededor de -20 pb, lo que difiere significativamente de una distribución de control, similar a lo que se ha informado en secuencias de lectura corta de CLL.Se analizaron las isoformas del gen ERGIC3, donde se encontró que una nueva isoforma que contiene el sitio de empalme proximal era más abundante en SF3B1-K700E.Tanto el SS 3' proximal como el distal se asociaron con patrones AS diferenciados que generaban múltiples isoformas.
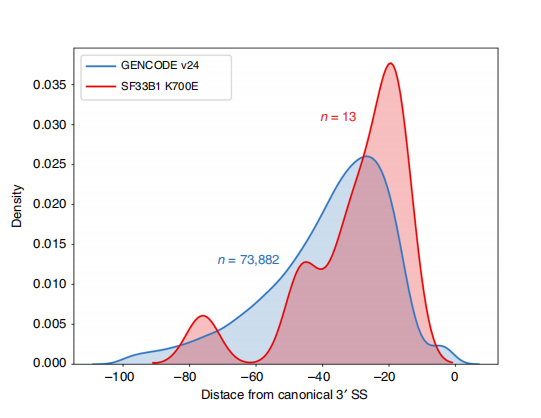
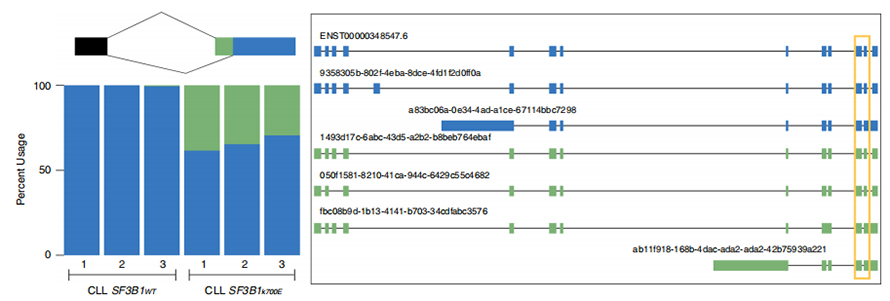
Figura 2. Patrones de empalme 3' alternativos identificados con datos de secuenciación de nanoporos
El análisis del uso de eventos de IR ha estado limitado durante mucho tiempo en análisis basados en lecturas cortas debido a la confianza en la identificación y cuantificación de IR.La expresión de las isoformas de IR en SF3B1-K700E y SF3B1-WT se cuantificó en función de secuencias de nanoporos, lo que revela una regulación negativa global de las isoformas de IR en SF3B1-K700E.
Figura 4. Intensidad agrícola y conectividad de la red en tres sistemas agrícolas (A y B);Análisis forestal aleatorio (C) y relación entre intensidad agrícola y colonización de HMA (D)
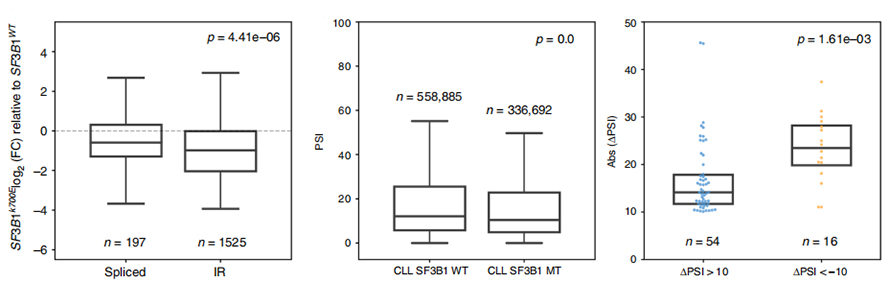
Figura 3. Los eventos de retención de intrones están regulados a la baja con mayor fuerza en CLL SF3B1-K700E
Tecnología
Secuenciación de lectura larga de nanoporos
NLa secuenciación de anoporos es una tecnología de secuenciación de señales eléctricas en tiempo real de una sola molécula.
DEl ADN o ARN bicatenario se unirá a una proteína nanoporosa incrustada en la biopelícula y se desenrollará bajo la dirección de una proteína motora.
DLas hebras de NA/ARN pasan a través de la proteína del canal de nanoporos a cierta velocidad bajo la acción de la diferencia de voltaje.
MLas oléculas generan diferentes señales eléctricas según la estructura química.
RLa detección de secuencias en tiempo real se logra mediante llamadas de base.
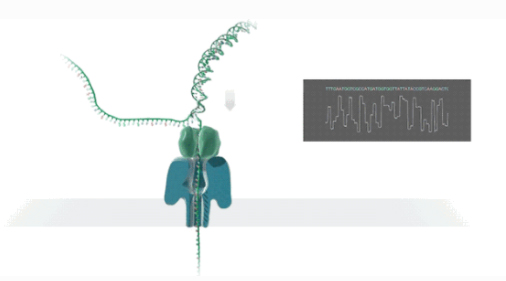
Realización de secuenciación del transcriptoma completo.
√ Saturación de datos
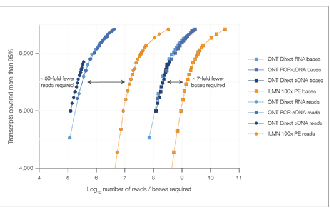
Se requieren siete veces menos lecturas para alcanzar una saturación de datos comparable.
√ Identificación de la estructura de la transcripción
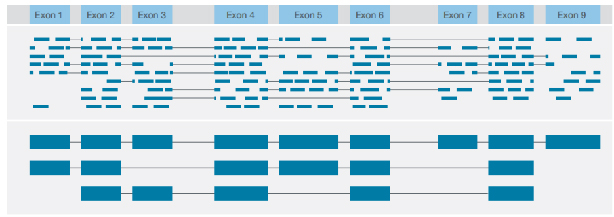
Identificación de diversas variantes estructurales con lectura completa consensuada de cada transcripción
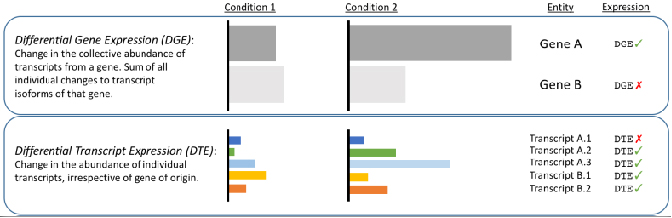
√ Análisis diferencial a nivel de transcripción: revela cambios ocultos en lecturas breves
Referencia
Tang AD, Soulette CM, Baren MJV, et al.La caracterización completa de la transcripción de la mutación SF3B1 en la leucemia linfocítica crónica revela una regulación negativa de los intrones retenidos [J].Comunicaciones de la naturaleza.
Tecnología y aspectos destacados tiene como objetivo compartir las aplicaciones exitosas más recientes de diferentes tecnologías de secuenciación de alto rendimiento en diversos campos de investigación, así como ideas brillantes en diseño experimental y minería de datos.
Hora de publicación: 08-ene-2022

