Secuenciación de bisulfito de representación reducida (RRBS)
Especificaciones de servicio
Plataforma: plataforma NovaSeq, PE150
Tipo de biblioteca: biblioteca de ADN tratada con bisulfito con inserto de 200-400 pb
Estrategia de secuenciación: extremo emparejado 150 pb
Salida de datos recomendada: 10 Gb de datos sin procesar/muestra
Requisitos de muestra
Cantidad de ADN: ≥ 2ug
Concentración de ADN: ≥ 20 ng/μl.
Pureza: OD260/280 = 1,8 a 2,0 sin degradación ni contaminación de ARN
Análisis bioinformático
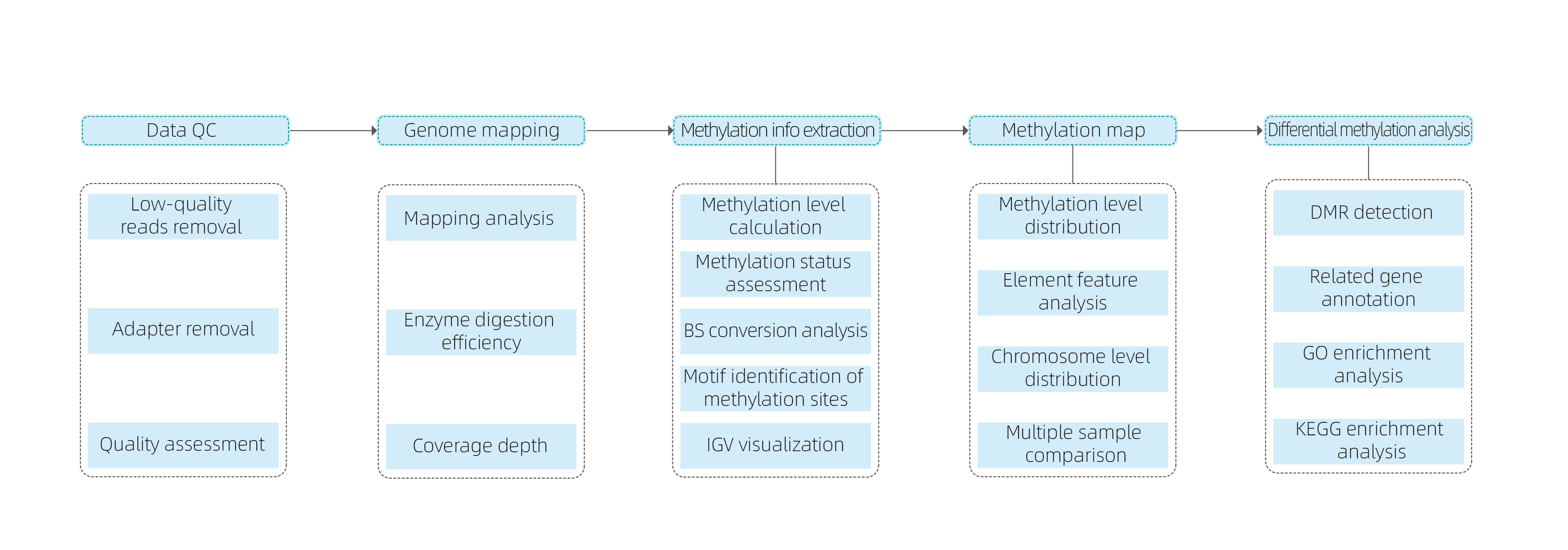
Flujo de trabajo del servicio

Entrega de muestra

construcción de biblioteca

Secuenciación

Análisis de los datos
Servicios postventa
a.Estadísticas de alineación del genoma de referencia.
Estadísticas de lecturas de mapeo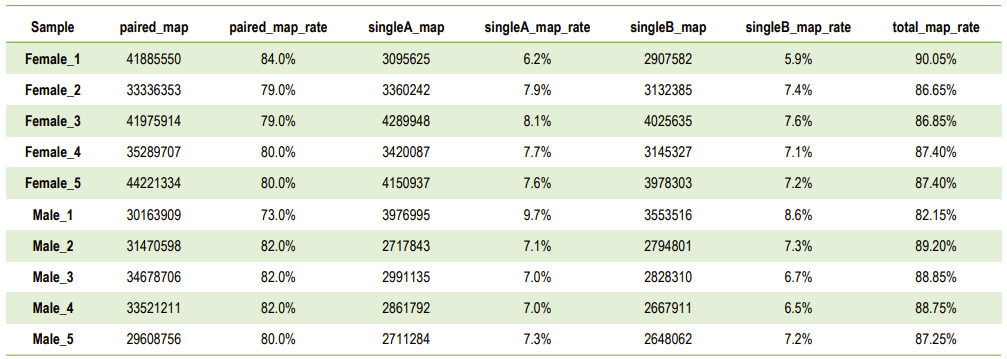
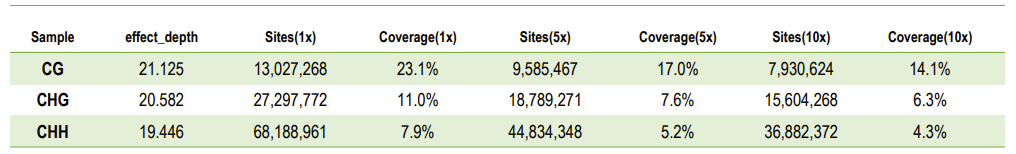
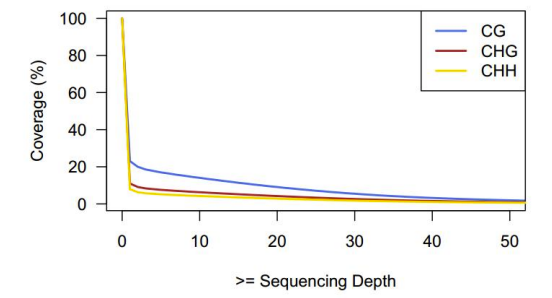
SEstadísticas de profundidad de secuenciación y cobertura del sitio C.
Acobertura acumulada
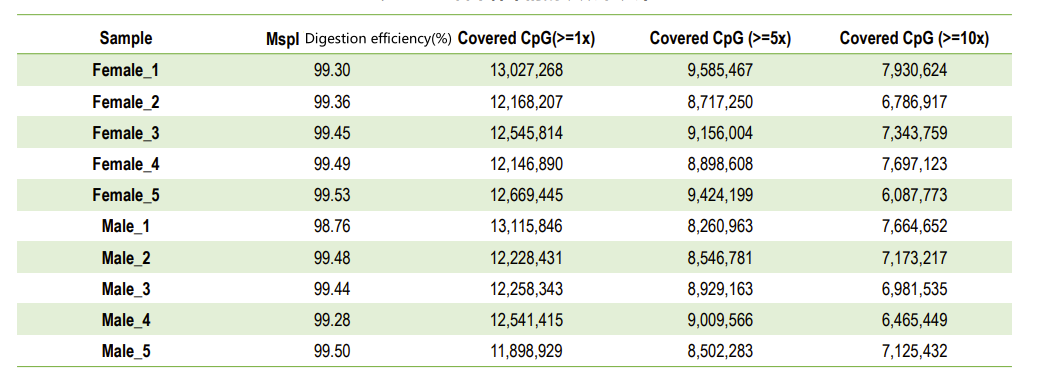
Deficiencia de digestión de cada muestra
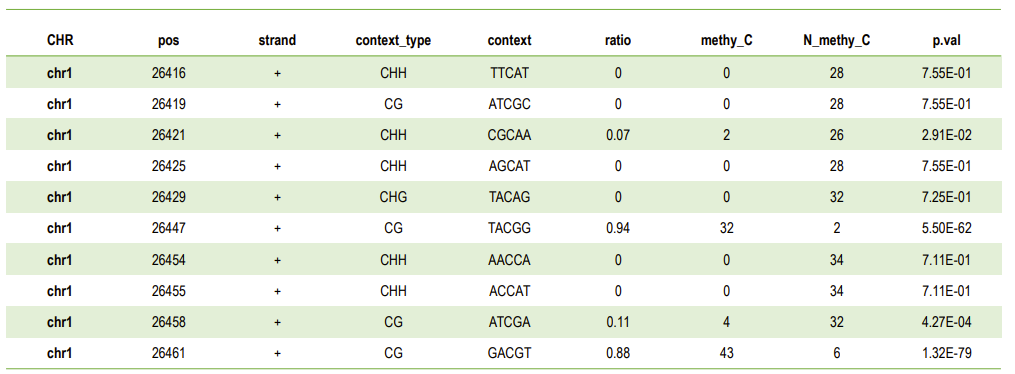
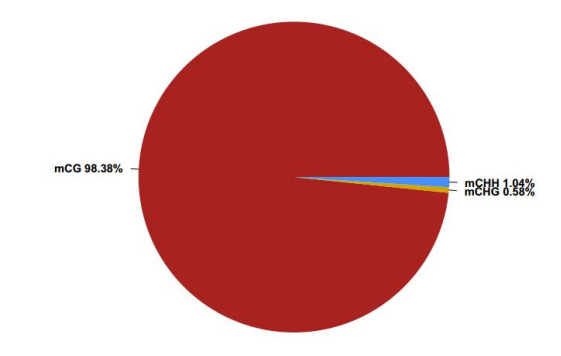
b.Detección del sitio de metilación
Lista de niveles de metilación en el sitio C
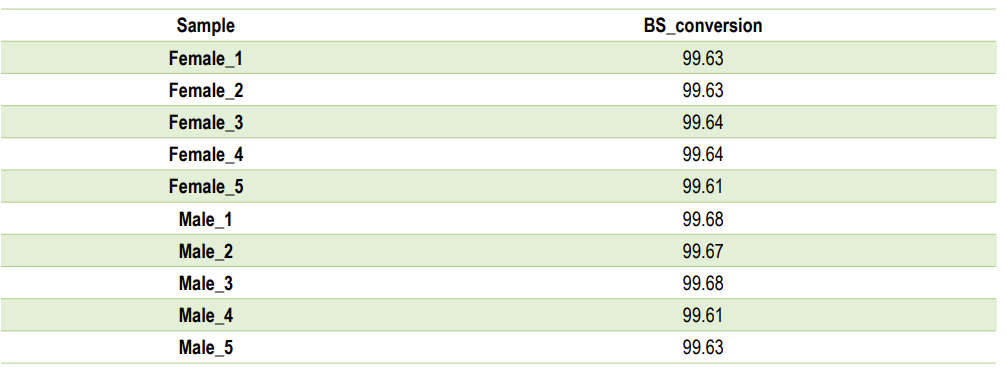
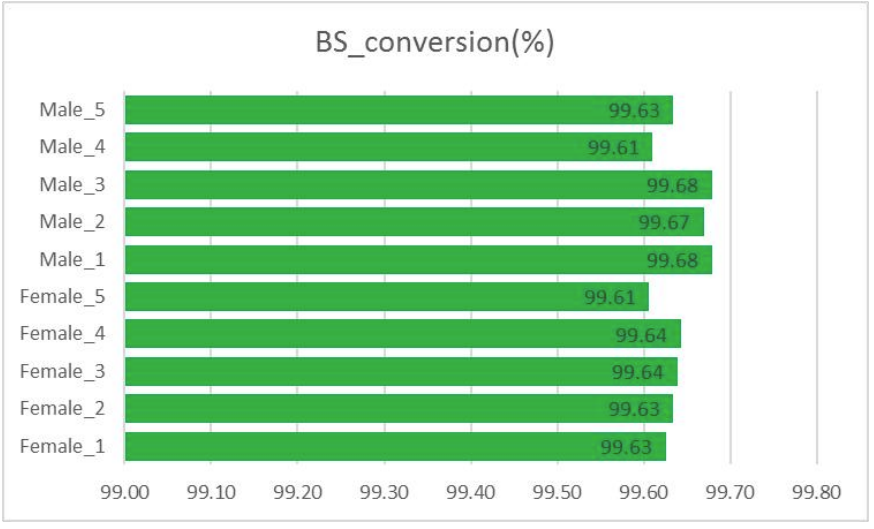
Bconversión S
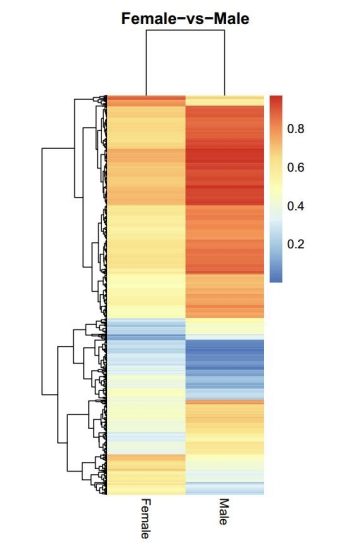
Distribución del nivel de metilación.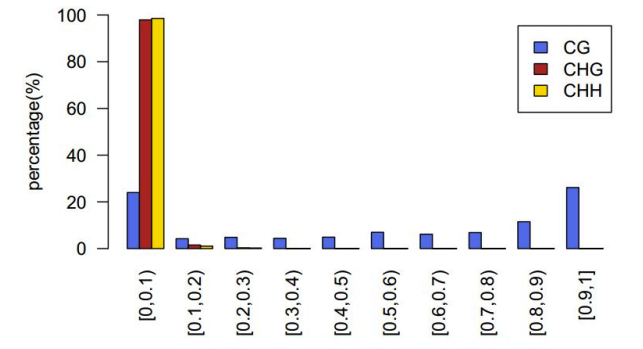
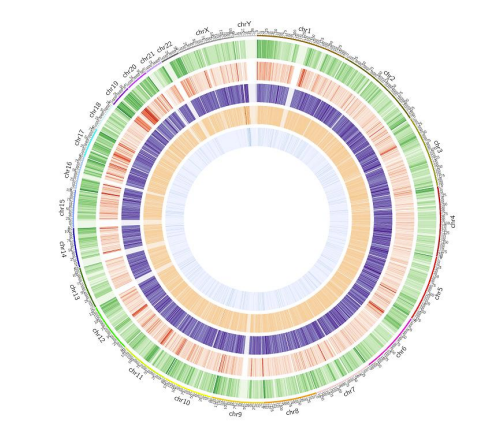
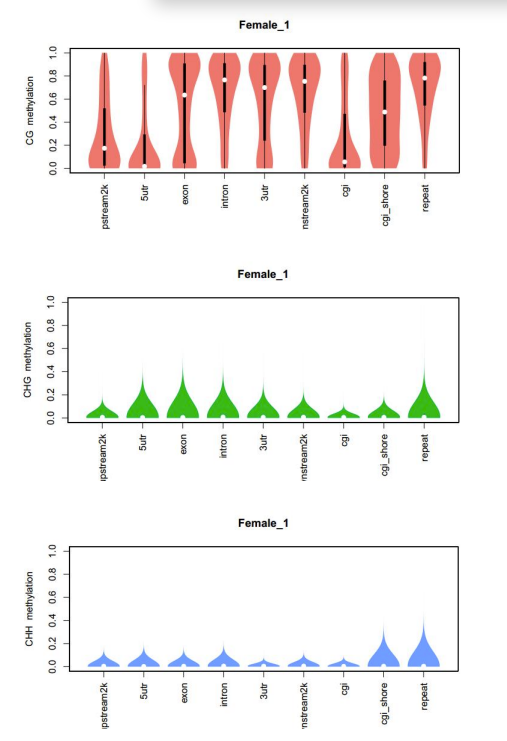
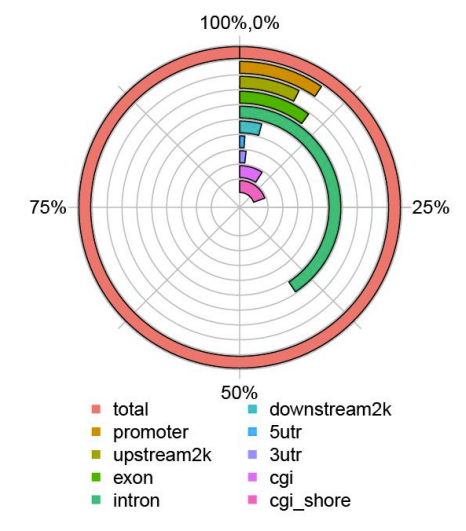
c.Mapa de metilación
Gelemento enoma
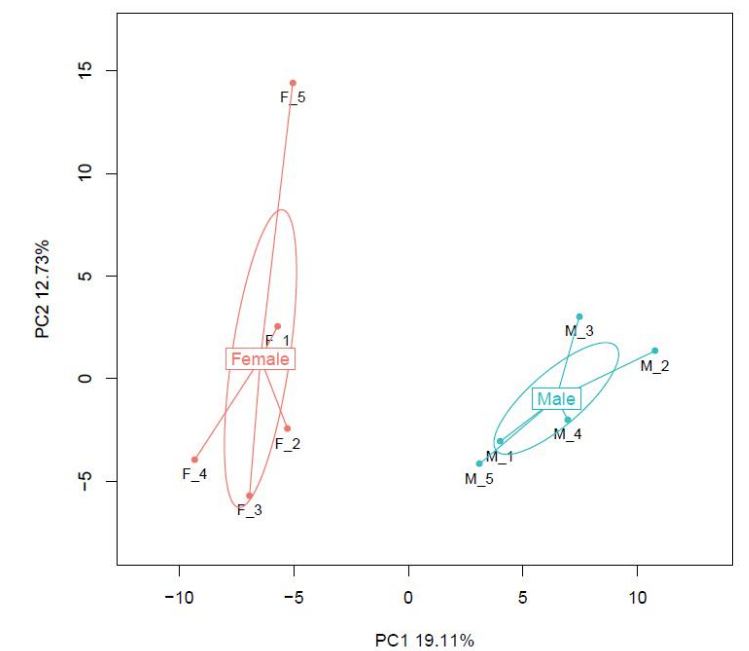
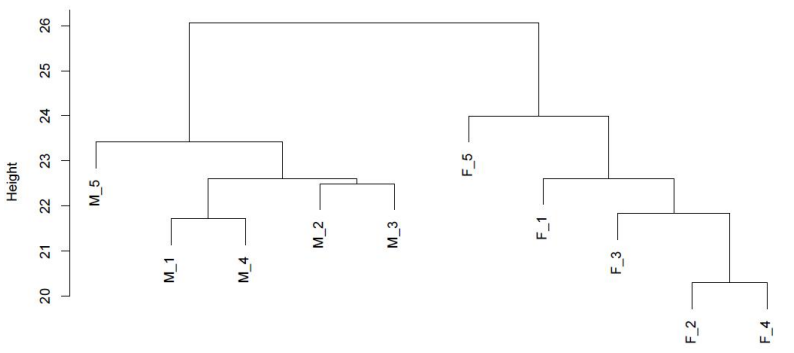
d.Comparación de niveles de metilación entre muestras.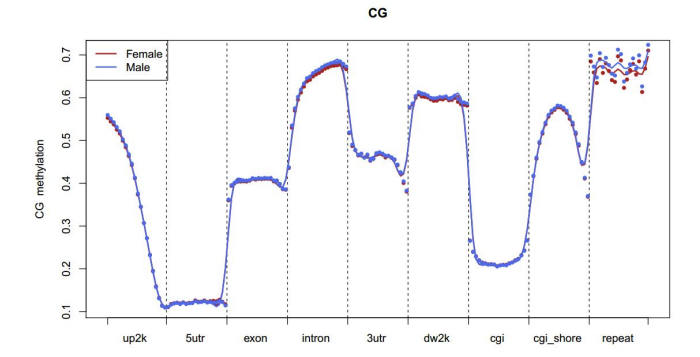
PCorrelación aérea de la metilación de CG.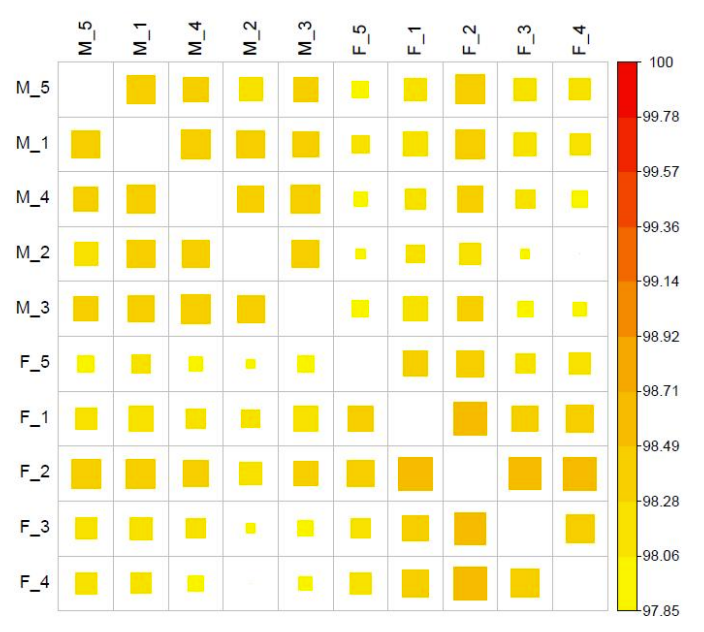
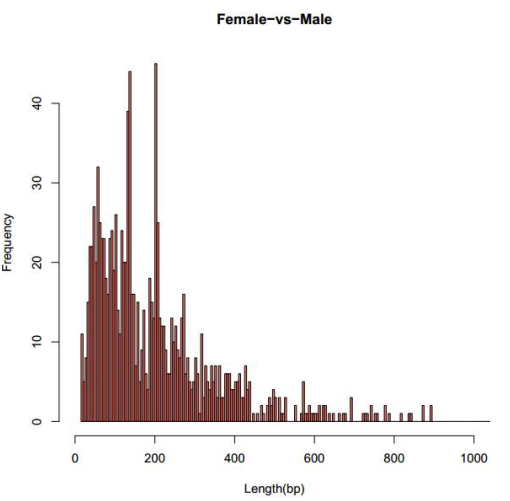
d.Análisis diferencial de metilación.
Sestadísticas de DMR