Secuenciación del transcriptoma completo – Illumina
Características
● Biblioteca dual para secuenciar el transcriptoma completo: agotamiento del ARNr seguido de preparación de la biblioteca PE150 y selección de tamaño seguida de preparación de la biblioteca SE50
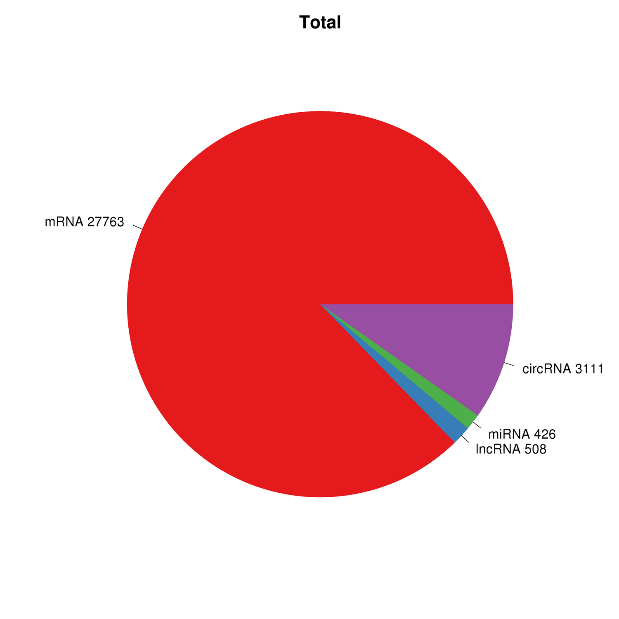
● Análisis bioinformático completo de ARNm, ARNc, ARNcirc y miARN en informes bioinformáticos separados
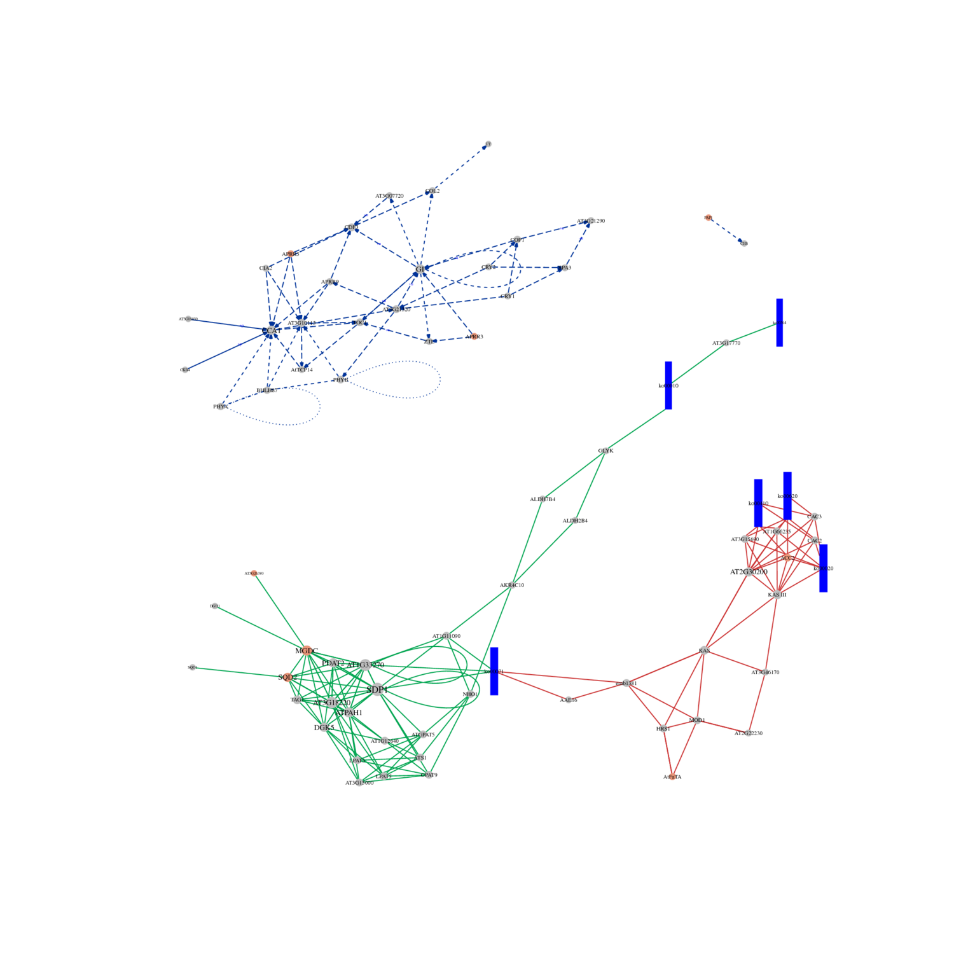
● Análisis conjunto de toda la expresión de ARN en un informe combinado, incluido el análisis de redes de ARNce.
Ventajas del servicio
●Análisis en profundidad de las redes regulatorias: El análisis de la red de ceRNA es posible mediante la secuenciación conjunta de mRNA, lncRNA, circRNA y miRNA y mediante un flujo de trabajo bioinformático exhaustivo.
●Anotación completa: utilizamos múltiples bases de datos para anotar funcionalmente los genes expresados diferencialmente (DEG) y realizar el análisis de enriquecimiento correspondiente, proporcionando información sobre los procesos celulares y moleculares subyacentes a la respuesta del transcriptoma.
●Amplia experiencia: con un historial de cierre exitoso de más de 2000 proyectos de transcriptomas completos en diversos dominios de investigación, nuestro equipo aporta una gran experiencia a cada proyecto.
●Control de calidad riguroso: implementamos puntos de control centrales en todas las etapas, desde la preparación de muestras y bibliotecas hasta la secuenciación y la bioinformática.Este seguimiento meticuloso garantiza la entrega de resultados consistentes de alta calidad.
● Anotación completa: utilizamos múltiples bases de datos para anotar funcionalmente los genes expresados diferencialmente (DEG) y realizar el análisis de enriquecimiento correspondiente, proporcionando información sobre los procesos celulares y moleculares subyacentes a la respuesta del transcriptoma.
●Soporte postventa: Nuestro compromiso se extiende más allá de la finalización del proyecto con un período de servicio postventa de 3 meses.Durante este tiempo, ofrecemos seguimiento del proyecto, asistencia en la resolución de problemas y sesiones de preguntas y respuestas para abordar cualquier consulta relacionada con los resultados.
Requisitos de muestra y entrega
| Biblioteca | Estrategia de secuenciación | Datos recomendados | Control de calidad |
| ARNr agotado | Iluminación PE150 | 16 GB | Q30≥85% |
| Tamaño seleccionado | Iluminación SE50 | 10-20 millones de lecturas |
Requisitos de muestra:
Nucleótidos:
| Conc.(ng/μl) | Cantidad (μg) | Pureza | Integridad |
| ≥ 100 | ≥ 1 | DO260/280=1,7-2,5 DO260/230=0,5-2,5 En el gel se muestra contaminación limitada o nula de proteínas o ADN. | Plantas: RIN≥6,5 Animales: RIN≥7.0 5,0≥28S/18S≥1,0; elevación basal limitada o nula |
Entrega de muestra recomendada
Envase:
Tubo de centrífuga de 2 ml (no se recomienda papel de aluminio)
Etiquetado de muestras: Grupo+réplica, por ejemplo, A1, A2, A3;B1, B2, B3... ...
Envío:
1. Hielo seco: las muestras deben empaquetarse en bolsas y enterrarse en hielo seco.
2.Tubos RNAstable: las muestras de ARN se pueden secar en un tubo de estabilización de ARN (por ejemplo, RNAstable®) y enviarse a temperatura ambiente.
Flujo de trabajo del servicio

Diseño de experimentos

Entrega de muestra

extracción de ARN

construcción de biblioteca

Secuenciación

Análisis de los datos

Servicios postventa
Bioinformática
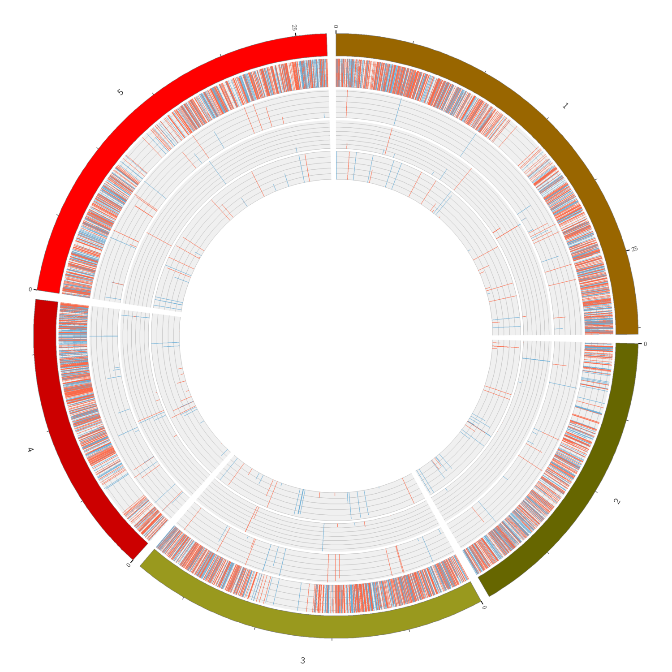
Descripción general de la expresión de ARN
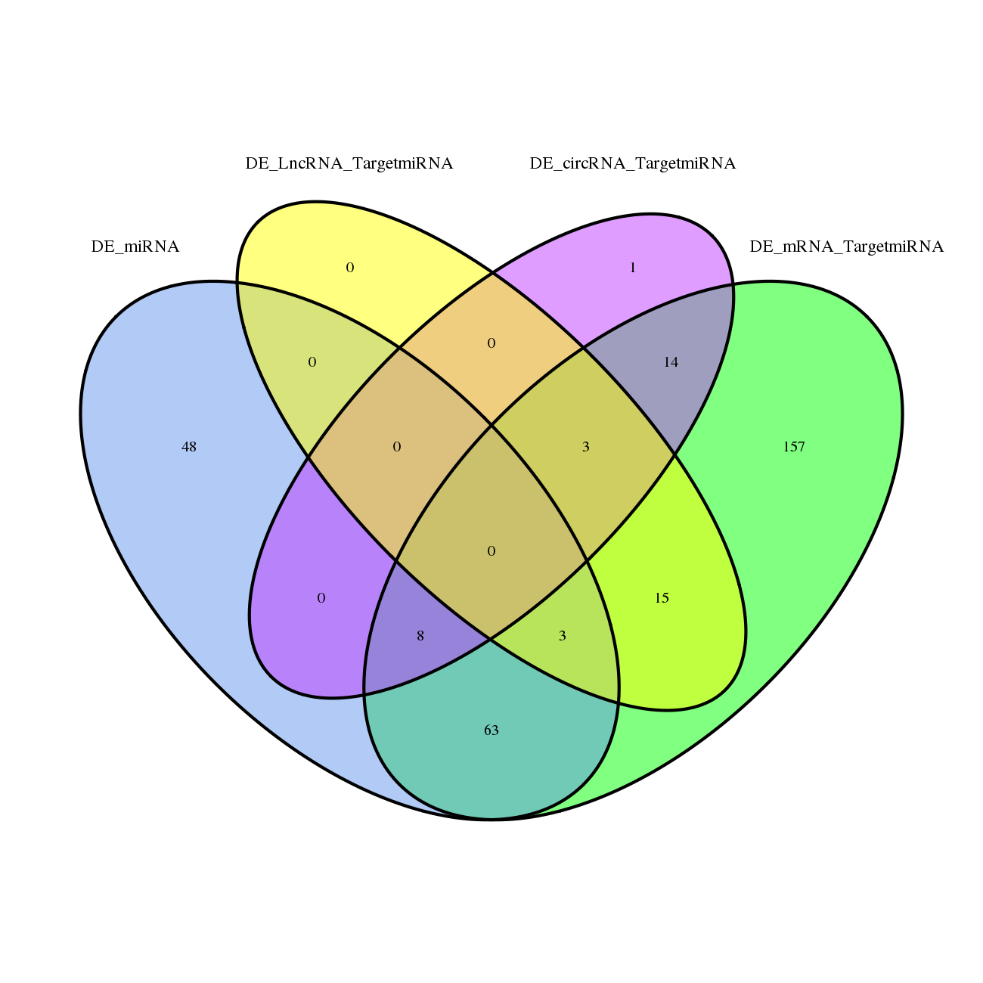
Genes expresados diferencialmente
análisis de ARNce
Explore los avances en la investigación facilitados por los servicios completos de secuenciación del transcriptoma de BMKGene a través de una colección seleccionada de publicaciones.
Dai, Y. et al.(2022) 'Perfiles de expresión completos de ARNm, ARNc y miARN en la enfermedad de Kashin-Beck identificados mediante secuenciación de ARN', Molecular Omics, 18 (2), págs.doi: 10.1039/D1MO00370D.
Liu, N.nan y col.(2022) 'Análisis completo de transcriptomas de la resistencia al frío de Apis cerana en la montaña Changbai durante el período de invernada', Gene, 830, págs.doi: 10.1016/J.GENE.2022.146503.
Wang, XJ y cols.(2022) 'Priorización basada en la integración multiómica de redes de regulación de ARN endógeno en competencia en el cáncer de pulmón de células pequeñas: características moleculares y fármacos candidatos', Fronteras en oncología, 12, pág.904865. doi: 10.3389/FONC.2022.904865/BIBTEX.
Xu, P. y col.(2022) 'El análisis integrado de los perfiles de expresión de lncRNA/circRNA-miRNA-mRNA revela nuevos conocimientos sobre mecanismos potenciales en respuesta a los nematodos agalladores en el maní', BMC Genomics, 23 (1), págs.doi: 10.1186/S12864-022-08470-3/FIGURAS/7.
Yan, Z. y col.(2022) 'La secuenciación de ARN del transcriptoma completo destaca los mecanismos moleculares asociados con el mantenimiento de la calidad poscosecha en brócoli mediante irradiación con LED rojo', Postharvest Biology and Technology, 188, pág.111878. doi: 10.1016/J.POSTHARVBIO.2022.111878.