TRANSKRIPTOMIA
luonto
VIESTINTÄ
SF3B1-mutaation täyspitkä transkription karakterisointi kroonisessa lymfosyyttisessä leukemiassa paljastaa säilyneiden intronien vähenemisen
Täyspitkät transkriptit|Nanopore-sekvensointi|Vaihtoehtoinen isoformianalyysi
Tausta
Somaattisten mutaatioiden silmukointitekijässä SF3B1 on laajalti raportoitu liittyvän erilaisiin syöpiin, mukaan lukien krooninen lymfaattinen leukemia (CLL), uveal melanooma, rintasyöpä jne. Lisäksi lyhytlukuiset transkriptomitutkimukset ovat paljastaneet SF3B1-mutaatioiden aiheuttamia poikkeavia silmukointikuvioita.Näitä vaihtoehtoisia silmukointikuvioita koskevat tutkimukset ovat kuitenkin pitkään rajoittuneet tapahtumatasolle ja isoformitason tiedon puutteeseen lyhytlukuisten koottujen transkriptien rajoitusten vuoksi.Täällä otettiin käyttöön nanohuokosekvensointialusta täyspitkien transkriptien luomiseksi, mikä mahdollisti AS-isoformien tutkimisen.
Kokeellinen suunnittelu
Kokeilut
Ryhmittely:1. CLL-SF3B1(WT) 2. CLL-SF3B1 (K700E-mutaatio);3. Normaalit B-solut
Sekvensointistrategia:MinION 2D -kirjaston sekvensointi, PromethION 1D -kirjaston sekvensointi;lyhyesti luettava data samoista näytteistä
Sekvensointialusta:ONT MinION;ONT PromethION;
Bioinformaattinen analyysi
Tulokset
Ayhteensä 257 miljoonaa lukua tuotettiin 6 CLL-näytteestä ja 3 B-solusta.Keskimäärin 30,5 % näistä lukemista tunnistettiin täyspituisiksi transkripteiksi.
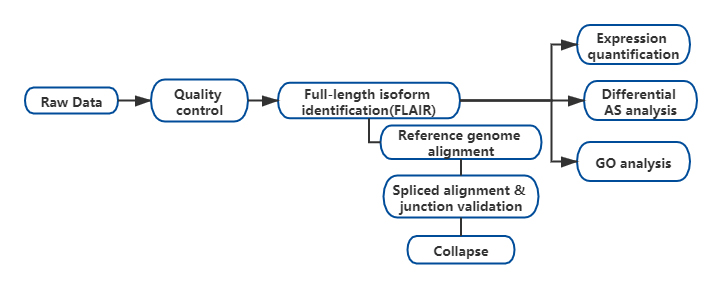
FRNA:n pitkäaikainen vaihtoehtoinen isoformianalyysi (FLAIR) kehitettiin luomaan joukko erittäin luotettavia isoformeja.FLAIR voidaan tiivistää seuraavasti:
Nanopore lukee kohdistuksen: tunnista yleinen transkriptirakenne referenssigenomin perusteella;
Splice-liitoksen korjaus: korjaa sekvenssivirheet (punainen) silmukointikohdalla joko annotoiduista introneista, introneista lyhyen lukutiedon perusteella tai molemmista;
Collapse: yhteenveto edustavista isoformeista perustuen silmukointiliitosketjuihin (ensikierrossarja).Valitse korkean luotettavuuden isofrom tukilukemien lukumäärän perusteella (Kynnys: 3).
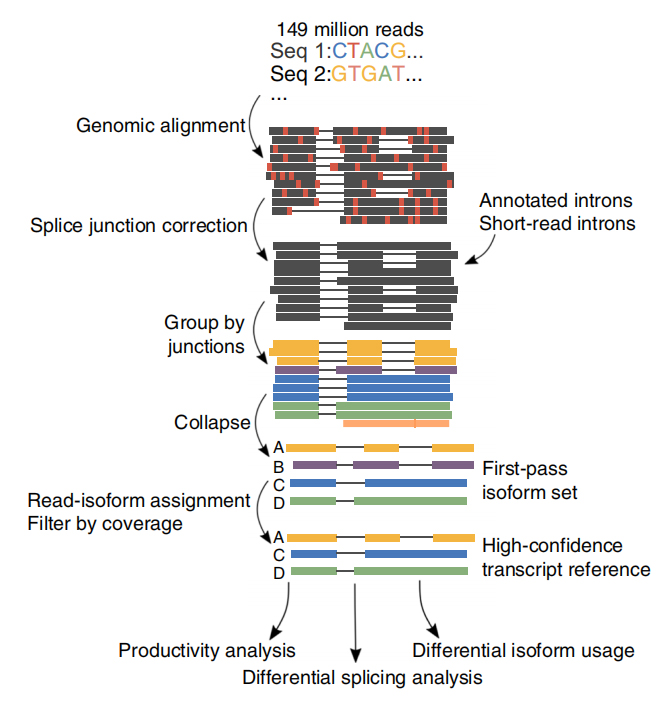
Kuva 1. FLAIR-analyysi SF3B1-mutaatioon liittyvien täysimittaisten isoformien tunnistamiseksi CLL:ssä
FLAIR tunnisti 326 699 erittäin luotettavaa silmukoitua isoformia, joista 90 % on uusia isoformeja.Useimpien näistä huomauttamattomista isoformeista havaittiin olevan uusia yhdistelmiä tunnettuja silmukointiliitoksia (142 971), kun taas loput uudet isomuodot sisälsivät joko säilytetyn introni (21 700) tai uuden eksonin (3594).
Long-read-sekvenssit mahdollistavat mutanttien SF3B1-K700E:n muunnettujen silmukointikohtien tunnistamisen isoformitasolla.35 vaihtoehtoisen 3'SS:n ja 10 vaihtoehtoisen 5'SS:n havaittiin olevan merkittävästi erilainen silmukointi SF3B1-K700E:n ja SF3B1-WT:n välillä.33 muutoksesta 35:stä löydettiin hiljattain pitkään luettujen sekvenssien avulla.Nanopore-tiedoissa etäisyyden jakauma SF3B1-K700E:llä muunnettujen 3'SS:ien ja kanonisten kohtien välillä on noin -20 emäsparia, mikä eroaa merkittävästi kontrollijakaumasta, joka on samanlainen kuin mitä on raportoitu CLL:n lyhytlukusekvensseissä.ERGIC3-geenin isoformeja analysoitiin, jolloin uusi isoformi, joka sisälsi proksimaalisen silmukointikohdan, havaittiin runsaammin SF3B1-K700E:ssä.Sekä proksimaalinen että distaalinen 3'SS liittyivät erottuviin AS-kuvioihin, jotka synnyttivät useita isoformeja.
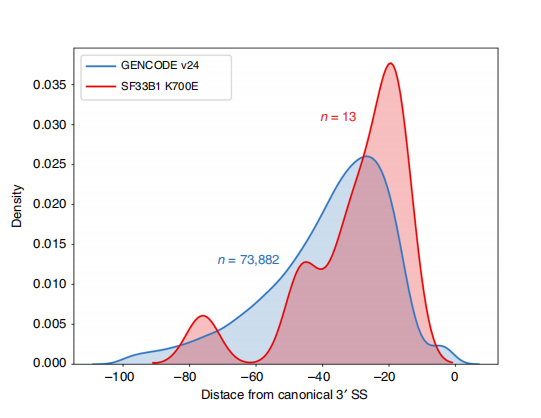
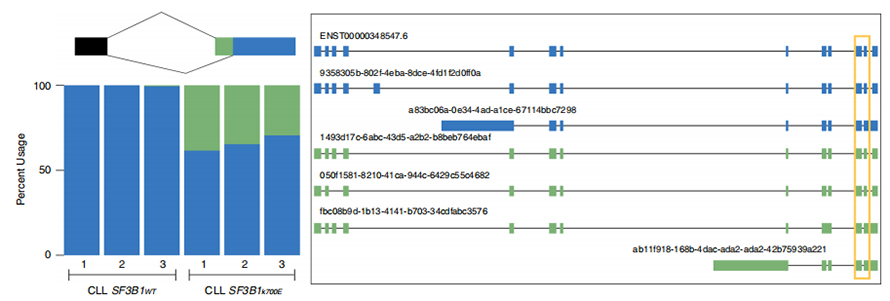
Kuva 2. Vaihtoehtoiset 3′ silmukointikuviot, jotka on tunnistettu nanohuokosekvensointitiedoilla
IR-tapahtumien käyttöanalyysi on ollut pitkään rajoitettu lyhytlukupohjaisessa analyysissä, koska se luottaa IR-tunnistukseen ja kvantifiointiin.IR-isoformien ilmentyminen SF3B1-K700E:ssä ja SF3B1-WT:ssä kvantifioitiin nanohuokosekvenssejen perusteella, mikä paljasti IR-isoformien maailmanlaajuisen alasäätelyn SF3B1-K700E:ssä.
Kuva 4. Maatalouden intensiteetti ja verkkoyhteydet kolmen viljelyjärjestelmän välillä (A ja B);Satunnainen metsäanalyysi (C) ja suhde maatalouden intensiteetin ja AMF-kolonisaation välillä (D)
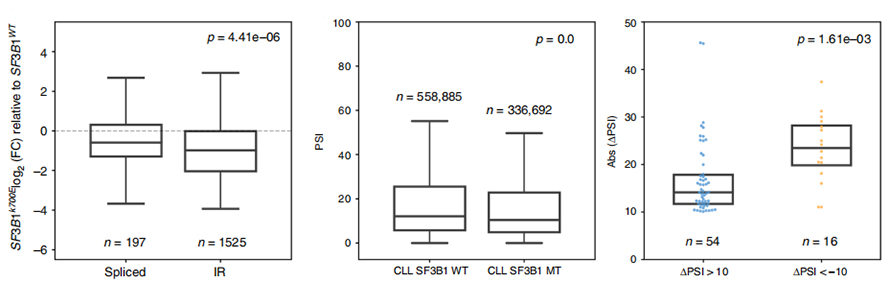
Kuva 3. Intronien vuokraustapahtumat ovat voimakkaammin alennettuja CLL:ssä SF3B1-K700E
Tekniikka
Nanopore Pitkään luettu sekvensointi
Nanopore-sekvensointi on yhden molekyylin reaaliaikainen sähköisen signaalin sekvensointitekniikka.
Dkaksijuosteinen DNA tai RNA sitoutuu nanohuokoiseen proteiiniin, joka on upotettu biofilmiin ja purkautuu moottoriproteiinin johdolla.
DNA/RNA-säikeet kulkevat nanohuokoskanavaproteiinin läpi tietyllä nopeudella jännite-eron vaikutuksesta.
Molekyylit tuottavat erilaisia sähköisiä signaaleja kemiallisen rakenteen mukaan.
Rsekvenssien ea-aikainen tunnistus saavutetaan peruskutsulla.
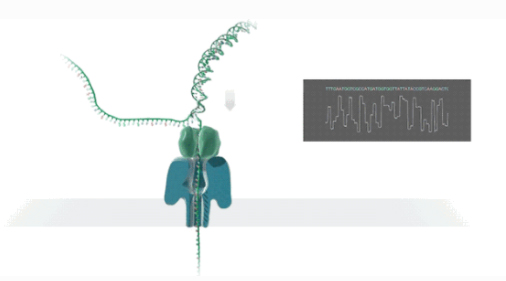
Täyspitkän transkriptisekvenssin suoritus
√ Tietojen kylläisyys
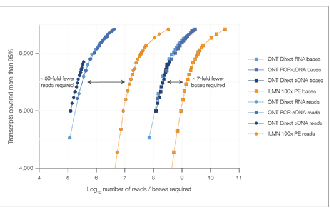
7 kertaa vähemmän lukuja tarvitaan vertailukelpoisen tiedon kyllästymisen saavuttamiseksi.
√ Transkription rakenteen tunnistus
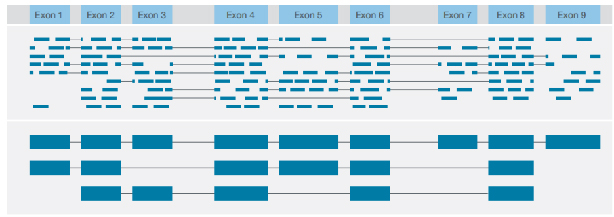
Erilaisten rakenteellisten varianttien tunnistaminen kunkin transkriptin täysimittaisella konsensuksella
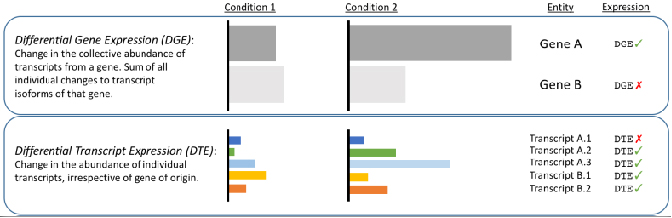
√ Transkriptiotason differentiaalianalyysi - Paljasta lyhyiden lukujen piilottamat muutokset
Viite
Tang AD, Soulette CM, Baren MJV, et ai.SF3B1-mutaation täyspitkä transkription karakterisointi kroonisessa lymfosyyttisessä leukemiassa paljastaa säilyneiden intronien alentumisen [J].Luontoviestintä.
Tekniikka ja kohokohdat Tavoitteena on jakaa erilaisten korkean suorituskyvyn sekvensointiteknologioiden viimeisimpiä onnistuneita sovelluksia eri tutkimusareenoilla sekä loistavia ideoita kokeelliseen suunnitteluun ja tiedon louhintaan.
Postitusaika: 08.01.2022

