MINSKLIKE GENOMIKA
natuer genetika
Langlêzen sequencing identifisearret GGC-werhelle útwreidingen yn NOTCH2NLC assosjearre mei neuronale yntranukleêre ynklúzjesykte
ONT resequencing |Illumina |Folsleine exome-sekwinsje |CRISPR-Cas9 ONT rjochte sequencing |RNA-seq |ONT 5mC methylaasje oprop
Hichtepunten
1.By Linkage-analyze op in grutte NIID-famylje waarden twa keppele regio's identifisearre.
2.ONT-basearre lange-lêzen sequencing en Cas-9 mediated ferriking ONT sequencing ûntdutsen in potinsjele genetyske oarsaak fan NIID, GGC werhelling útwreidings yn 5' UTR fan NOTCH2NLC.Dizze stúdzje rapporteare werhelle útwreidingen yn minsklike spesifike genen foar it earst dy't evoluearre troch segmintele duplikaasjes.
3.RNA-sekwinsje iepenbiere abnormale antisense-transkrippen yn it begjin of binnen GGC-werhelle útwreidingsregio's yn NOTCH2NLC.
Eftergrûn
Neuronal intranuclear inclusion disease (NIID) is in progressive en fatale neurodegenerative sykte, dy't karakterisearre wurdt troch de oanwêzigens fan eosinofiele hyaline yntranukleêre ynklúzjes yn sintrale en perifeare nervous systemen.De heul fariabele klinyske manifestaasjes ferheegje grutte swierrichheden by diagnoaze oant yntroduksje fan hûdbiopsy.Histopatology-basearre metoaden hawwe lykwols noch te lijen fan misdiagnoaze, wat freget om in genetysk begryp fan NIID.
Prestaasjes
Linkage analyze
Short-lêzen sequencing basearre hiele genome sequencing (WGS) en hiele exome sequencing (WES) waard útfierd op in grutte NIID famylje (13 beynfloede en 7 net beynfloede leden).Linkage-analyze op SNP's ekstrahearre út dizze gegevens lieten mar twa keppele regio's sjen: in 3.5 Mb-regio by 1p36.31-p36.22 (maksimum LOD=2.32) en in 58.1 Mb-regio by 1p22.1-q21.3 (maksimum LOD: 4.21) ).Gjin pathogene SNP's of CNV's waarden lykwols identifisearre yn dizze keppele regio's.
GGC repeat útwreidings yn NOTCH2NLC
Nanopore-basearre sequencing waard ferwurke op 13 troffen en 4 net beynfloede leden út 8 famyljes (in oar troffen lid waard sekwinsje troch Pacbio lange lêzen sequencing platfoarm.).Lange-lêzen gegevens iepenbiere sykte assosjearre GGC werhelling útwreidings yn 'e 5' UTR fan NOTCH2NLC gen mapping nei 58.1 Mb keppele regio (figuer 1).Dizze werhellingsútwreidingen waarden ek identifisearre yn alle 40 sporadyske NIID-gefallen hifke troch RP-PCR.
Cas-9 bemiddele doelsekwinsjes op nanopore-platfoarm waard brûkt om hegere lêsdekking te berikken op NOTCH2NLC-werhelling (100 X-1,795 X).Dizze konsensussekwinsjes kamen goed oerien mei eardere befinings oer GGC-werhelle útwreidingen.Boppedat waarden {(GGA)n (GGC)n}n werhellingen identifisearre as in potinsjele genetyske marker foar swakke-dominant fenotype (figuer 2).
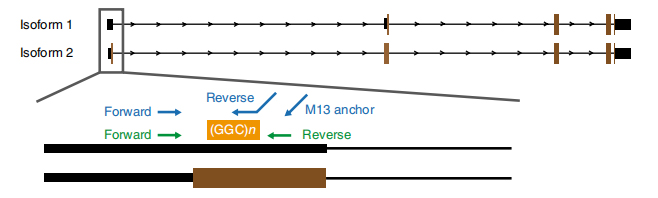
figuer 1. Disease assosjearre werhelling útwreiding identifisearre op exon 1 fan NOTCH2NLC isoforms.
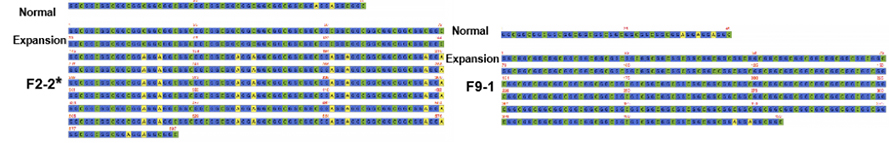
Ofbylding 2. Konsensussekwinsjes fan NPTCH2NLC werhelje yn NIID-pasjinten mei(*) of sûnder swakke-dominant fenotype
NOTCH2NL-genen binne minsklike spesifike genen, dy't leaud wurde dat se in wichtige rol spylje yn minsklike harsensevolúsje en neurologyske sykten.Trije NOTCH2-relatearre genen (NOTCH2NLA, NOTCH2NLB en NOTCH2NLC) mei> 99.1% sekwinsje-identiteit waarden lykwols net oplost oant de lêste minsklike genome-assemblage.Syntezefrije en langlêzen sequencing op nanopore-platfoarm hawwe opmerklike foardielen sjen litten by it oplossen fan regio's fan hege oerienkomst en (GGC) n werhellingen mei 100% GC-ryk.
GGC repeat útwreidings yn NOTCH2NLC
Transcriptome sequencing waard ferwurke op 2 beynfloede en 2 net beynfloede leden.Normalisearre lêsdjipte waard berekkene op sin en antisense stringen yn streamop fan earste eksons fan NOTCH2NL paralogs.Abnormale anty-sintranskripten waarden allinich fûn yn beynfloede gefallen, dy't yn it begjin of yn 'e werhellende útwreidingsregio sitte (Purple peaks yn F1-14 en F1-16 yn figuer 3.).Derneist waarden 54 DEG's identifisearre en waarden allegear ferrike yn GO- en MPO-termen relatearre oan neuronale funksjes.
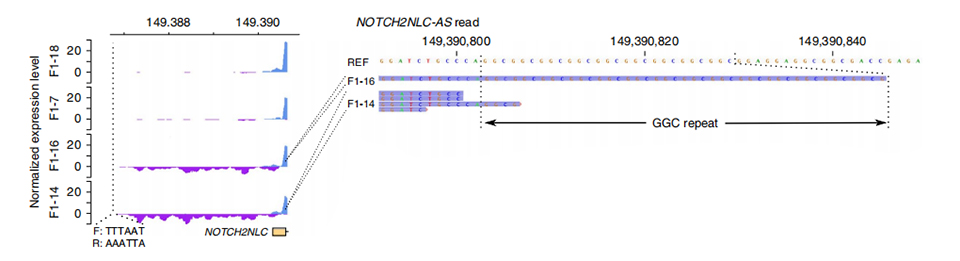
Figure 3. Normalisearre lêsdjipte op streamop fan 'e earste ekson fan NOTCH2NLC yn ûnoantaaste (boppe) en beynfloede (ûnder) gefallen.
Technology
Oxford Nanopore Technologies (ONT)
Nanopore sequencing ûnderskiedt him fan oare sequencing platfoarms, yn dat de nucleotides wurde lêzen direkt sûnder DNA synteze proses.As in inkele strân DNA troch in nano-grutte proteïnepoar (nanopore) giet, generearje ferskate nukleotiden ferskate ionyske streamingen, dy't kinne wurde fêstlein en oerbrocht yn folchoarder fan basen.ONT sequencing platfoarm sels toant gjin skynbere technyske limyt op 'e lingte fan DNA lêzen.Dêrom binne Ultra-lange lêzings (ULR's) beskikber foar genome-assemblage fan hege kwaliteit.Boppedat helpe dizze ekstreem lange lêzings, dy't lang genôch binne om komplekse folchoarderfunksjes as strukturele fariaasje te krúsjen, de beheiningen fan sekwinsje fan koarte lêzen hjir te oerwinnen.

Nanopore sequencing
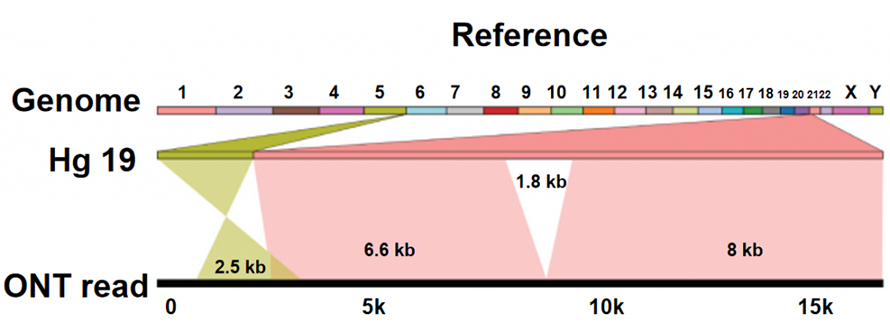
Struktuer fariaasje (SV) identifikaasje
Synthesis-frije sequencing foar it grutste part bewarre bleaun DNA methylation ynformaasje op template.Methylated A, T, C en G generearje ûnderskate ionyske streamingen fan un-methylearre, dy't direkt kinne wurde lêzen troch it platfoarm.Nanopore sequencing machtigje folsleine-genome profilearring fan sawol 5mC as 6mA by single-nucleotide resolúsje.
Referinsje
Jun Sone, et al.al.Langlêzen sequencing identifisearret GGC-werhelle útwreidingen yn NOTCH2NLC assosjearre mei neuronale yntranukleêre ynklúzjesykte.Natuergenetika (2019)
Technyk en hichtepunten stribbet nei it dielen fan de meast resinte súksesfolle tapassing fan ferskate technologyen foar sekwinsjes mei hege trochset yn ferskate ûndersyksarena's, lykas briljante ideeën yn eksperiminteel ûntwerp en data mining.
Post tiid: Jan-06-2022

