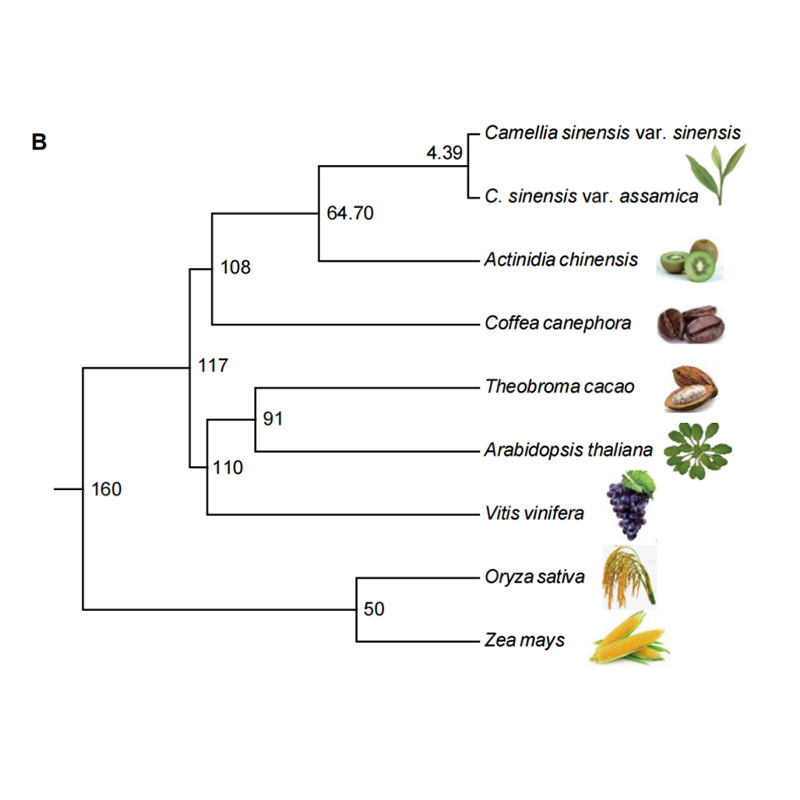
Xenómica comparada
Vantaxes do servizo
● Paquete completo de análises, que contén as oito análises máis habitualmente requiridas
● Alta fiabilidade na análise con interpretación detallada e facilmente comprensible dos resultados
● Figuras listas para publicar ben deseñadas
● Un equipo de bioinformática altamente cualificado cumpre diversas demandas de análise personalizada
● Menor tempo de resposta cunha maior precisión na análise
● Abundante experiencia con máis de 90 casos exitosos cun factor de impacto publicado acumulado superior a 900
Especificacións do servizo
| Tempo estimado de entrega | Número de especies | Análise |
| 30 días hábiles | 6-12 | Agrupación de familias xenéticas Expansión e contracción da familia xenética Construción da árbore filoxenética Estimación do tempo de diverxencia (requírese calibración de fósiles) Tempo de inserción LTR (Para plantas) Duplicación do xenoma completo (para plantas) Presión selectiva Análise de sintonia |
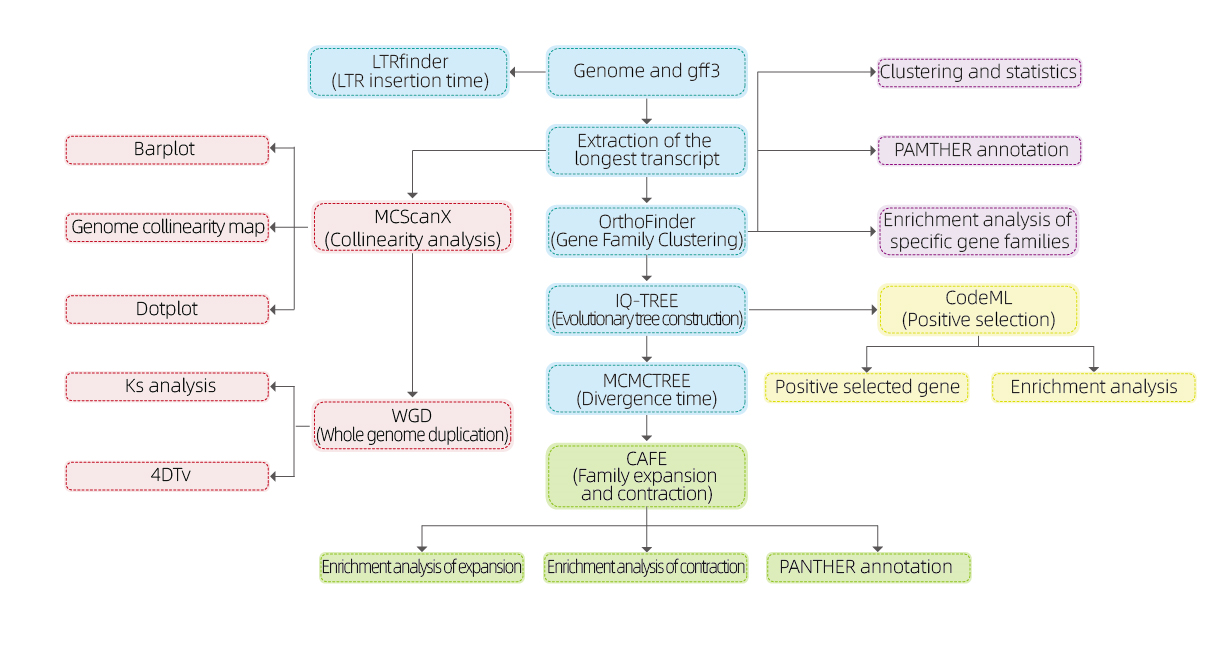
Análise bioinformática
● Familia xenética
● Filoxenética
● Tempo de diverxencia
● Presión selectiva
● Análise de sintonia
Requisitos de mostra e entrega
Requisitos de mostra:
Tecido ou ADN para a secuenciación e ensamblaxe do xenoma
Para Tecido
| Especie | Tecido | Enquisa | PacBio CCS |
| Animal | Tecido visceral | 0,5 ~ 1 g | ≥ 3,5 g |
| Tecido muscular | |||
| ≥ 5,0 g | |||
| ≥ 5,0 ml | |||
| Sangue de mamíferos | |||
| ≥ 0,5 ml | |||
| Sangue de aves/peixes | |||
| Planta | Folla Fresca | 1 ~ 2 g | ≥ 5,0 g |
| Pétalo/Talla | 1 ~ 2 g | ≥ 10,0 g | |
| Raíz/Semente | 1 ~ 2 g | ≥ 20,0 g | |
| Células | Célula cultivada | - | ≥ 1 x 108 |
Datos
Ficheiros de secuencia do xenoma (.fasta) e ficheiros de anotación (.gff3) de especies estreitamente relacionadas
Fluxo de traballo do servizo

Deseño de experimentos

Entrega da mostra

Construción da biblioteca

Secuenciación

Análise de datos

Servizos posvenda
*Os resultados de demostración que se mostran aquí son todos de xenomas publicados con Biomarker Technologies
1. Estimación do tempo de inserción de LTR: a figura mostra unha distribución bimodal única nos tempos de inserción de LTR-RT no xenoma do centeo de Weining, en comparación con outras especies.O pico máis recente apareceu hai uns 0,5 millóns de anos.
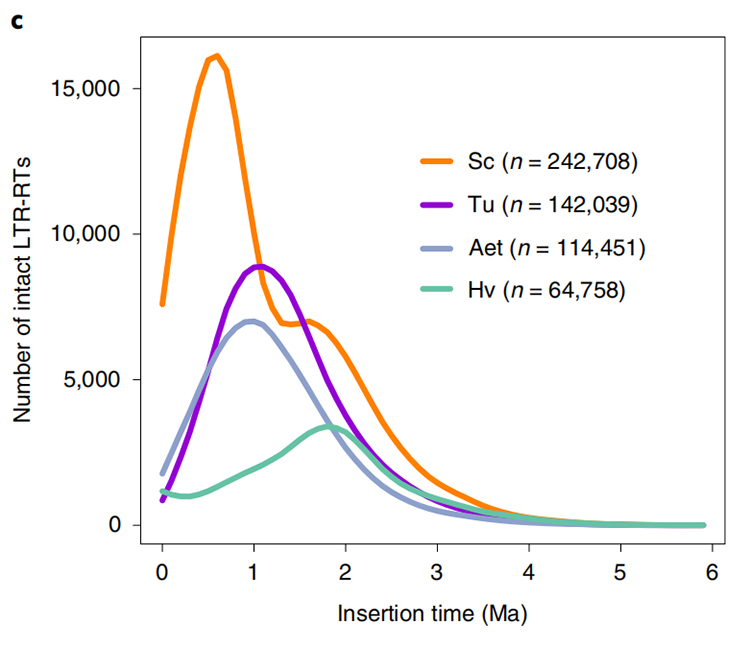
Li Guang et al.,Xenética da Natureza, 2021
2. Filoxenia e análise da familia de xenes en chayote (Sechium edule): Ao analizar o chayote e as outras 13 especies relacionadas na familia xenética, descubriuse que o chayote estaba máis estreitamente relacionado coa cabaciña de serpe (Trichosanthes anguina).Chayote derivado da cabaciña de serpe en aproximadamente 27-45 Mya e observouse duplicación do xenoma completo (WGD) en chayote en 25±4 Mya, que é o terceiro evento WGD en cucuibitaceae.
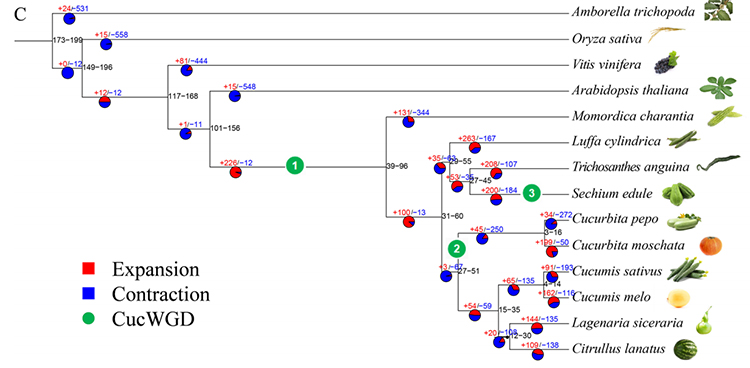
Fu A et al.,Investigación en Horticultura, 2021
3.Análise da sintonia: atopáronse algúns xenes relacionados coas fitohormonas no desenvolvemento da froita no chayote, a cabaciña e a cabaciña.A correlación entre chayote e cabaza é lixeiramente maior que a entre chayote e cabaza de serpe.
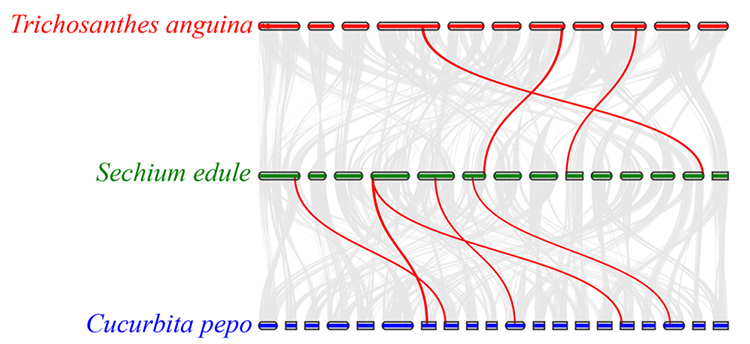
Fu A et al.,Investigación en Horticultura, 2021
4.Análise da familia de xenes: o enriquecemento de KEGG na expansión e contracción da familia de xenes nos xenomas de G.thurberi e G.davidsonii mostrou que se expandían os xenes relacionados coa biosíntese de esteroides e coa biosíntese de brassinosteroides.
Yang Z et al.,BMC Bioloxía, 2021
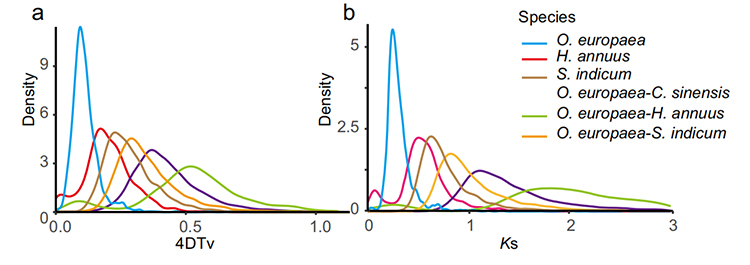
5.Análise de duplicación do xenoma completo: a análise da distribución 4DTV e Ks mostrou o evento de duplicación do xenoma completo.Os picos de intraespecie mostraron eventos de duplicación.Os picos de entre especies mostraron eventos de especiación.A análise indicou que en comparación coas outras tres especies estreitamente relacionadas, O. europaea pasou por unha duplicación xenética a gran escala máis recentemente.

Rao G et al.,Investigación en Horticultura, 2021
Caso BMK
Rosa sen espiña: coñecementos xenómicos relacionados coa adaptación á humidade
Publicado: National Science Review, 2021
Estratexia de secuenciación:
'O de BasyeSen espiñas' (R.Wichurainan) xenoma:
Aprox.93 X PacBio + aprox.90 X Nanopore + 267 X Illumina
Resultados clave
1. O xenoma de R.wichuraiana de alta calidade foi construído utilizando técnicas de secuenciación de lectura longa, que producen un conxunto de 530,07 Mb (o tamaño estimado do xenoma foi de aproximadamente 525,9 Mb por citometría de fluxo e 525,5 por estudo do xenoma; a heterocigosidade foi de aproximadamente 1,03%).A puntuación estimada de BUSCO foi do 93,9%.Comparando con "Old blush" (haploOB), a calidade e integridade deste xenoma confirmáronse mediante a precisión dunha base única e o índice de ensamblaxe LTR (LAI=20,03).O xenoma de R.wichuraiana contén 32.674 xenes codificantes de proteínas.
2.A análise conxunta multiómica, consistente en xenómica comparativa, transcriptómica e análise QTL da poboación xenética, revelou a especiación crucial entre R. wichuraiana e Rosa chinensis.Ademais, é probable que a variación da expresión dos xenes relacionados en QTL estea asociada co patrón de espinas do tronco.

A análise xenómica comparativa entre Basye;s Thornless e Rosa chinensis, incluíndo análise de sintonia, agrupación de xenes, análise de expansión e contracción, revelou un gran número de variacións relacionadas con trazos cruciais das rosas.A expansión única na familia de xenes NAC e FAR1/FRS era moi probable que se asociase coa resistencia á mancha negra.
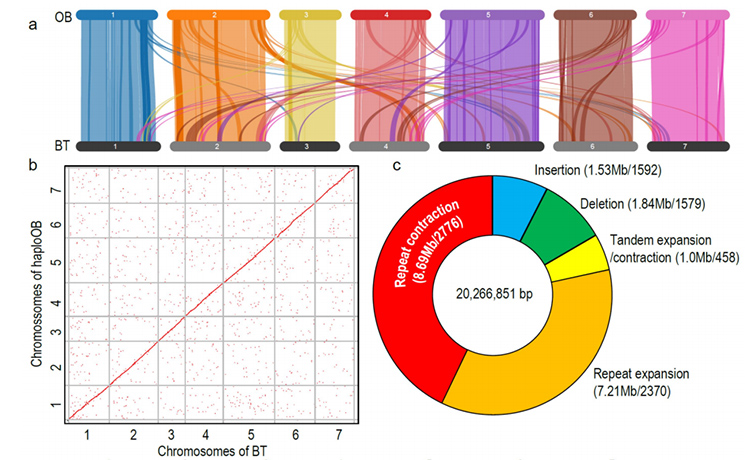

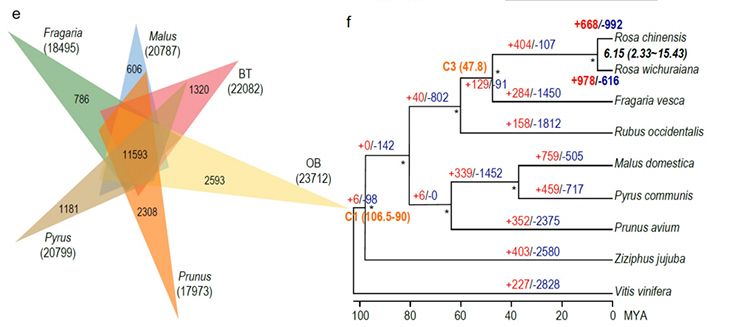
Análise xenómica comparativa entre xenomas BT e haploOB.
Zhong, M., et al."Rosa sen espina: coñecementos xenómicos relacionados coa adaptación á humidade"National Science Review, 2021;, nwab092.