Összehasonlító genomika
Szolgáltatás előnyei
● Átfogó elemzési csomag, amely a nyolc leggyakrabban szükséges elemzést tartalmazza
● Nagyfokú elemzési megbízhatóság az eredmények részletes és könnyen érthető értelmezésével
● Jól megtervezett, publikálásra kész figurák
● A magasan képzett bioinformatikai csapat sokrétű, személyre szabott elemzési igényt elégít ki
● Rövidebb átfutási idő nagyobb elemzési pontossággal
● Bőséges tapasztalat több mint 90 sikeres esettel, 900 feletti összesített közzétett impaktfaktorral
Szolgáltatási specifikációk
| Becsült átfutási idő | A fajok száma | Elemzések |
| 30 munkanap | 6-12 | Géncsalád klaszterezés Géncsalád bővülése és összehúzódása Filogenetikus faépítés Eltérési idő becslése (Fosszilis kalibráció szükséges) LTR beillesztési idő (növényekhez) Teljes genom megkettőzése (növényekhez) Szelektív nyomás Szinténanalízis |
Bioinformatikai elemzések
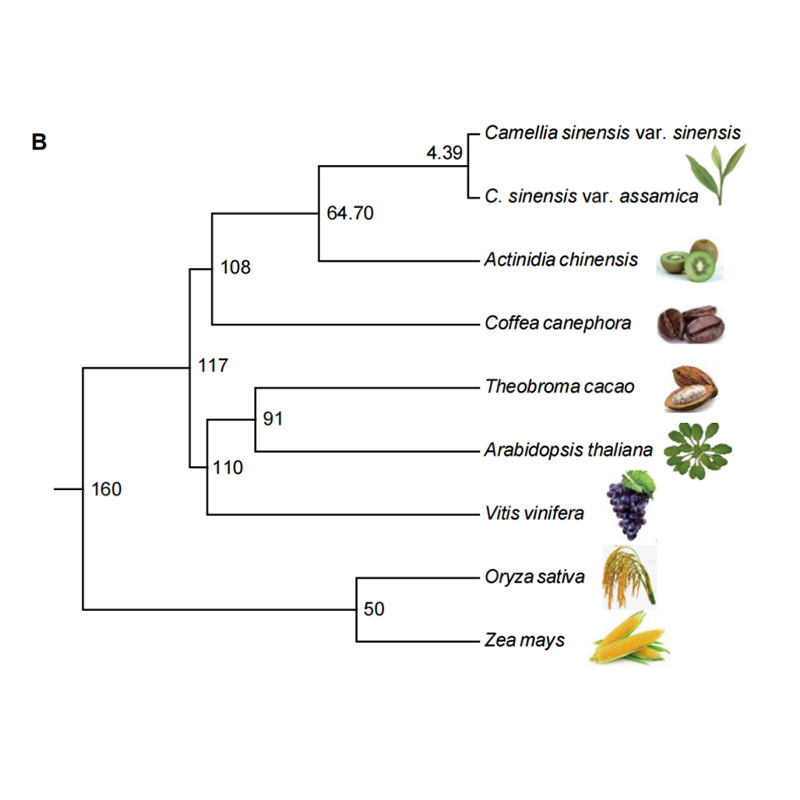
● Géncsalád
● Filogenetika
● Eltérési idő
● Szelektív nyomás
● Szinténanalízis
Mintakövetelmények és szállítás
Mintakövetelmények:
Szövet vagy DNS a genom szekvenálásához és összeállításához
A Tissue számára
| Faj | Szövet | Felmérés | PacBio CCS |
| Állat | Viscerális szövet | 0,5-1 g | ≥ 3,5 g |
| Izomszövet | |||
| ≥ 5,0 g | |||
| ≥ 5,0 ml | |||
| Emlős vér | |||
| ≥ 0,5 ml | |||
| Baromfi/hal vér | |||
| Növény | Friss Levél | 1-2 g | ≥ 5,0 g |
| Szirom/szár | 1-2 g | ≥ 10,0 g | |
| Gyökér/Mag | 1-2 g | ≥ 20,0 g | |
| Sejtek | Tenyésztett sejt | - | ≥ 1 x 108 |
Adat
A közeli rokon fajok genomszekvencia-fájljai (.fasta) és annotációs fájlok (.gff3).
Szerviz munkafolyamat

Kísérleti tervezés

Mintaszállítás

Könyvtárépítés

Sorrendezés

Adatelemzés

Értékesítés utáni szolgáltatások
*Az itt látható demo eredmények mind a Biomarker Technologies által közzétett genomokból származnak
1.LTR beépülési idő becslése: Az ábra egyedi bimodális eloszlást mutat az LTR-RT-k beépülési idejének Weining rozs genomjában, összehasonlítva más fajokkal.A legutóbbi csúcs körülbelül 0,5 millió évvel ezelőtt jelent meg.
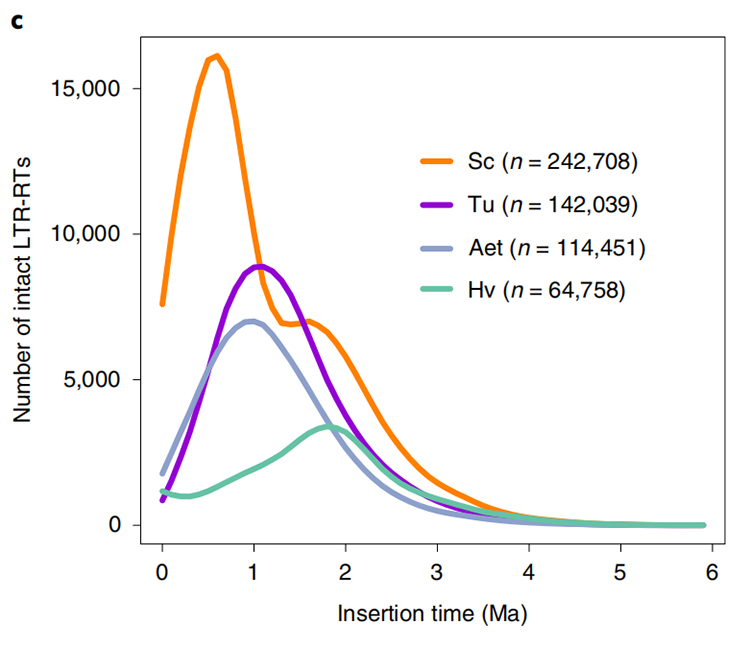
Li Guang és társai,Természetgenetika, 2021
2. A chayote (Sechium edule) filogenetikai és géncsalád-analízise: A chayote és a géncsalád másik 13 rokon faja elemzésével megállapították, hogy a Chayote a legszorosabb rokonságban áll a kígyótökkel (Trichosanthes anguina).A kígyótökből származó chayote-t körülbelül 27-45 Mya-ban, a teljes genom megkettőződését (WGD) pedig 25±4 Mya-ban figyelték meg, ami a harmadik WGD esemény a cucuibitaceae-ban.
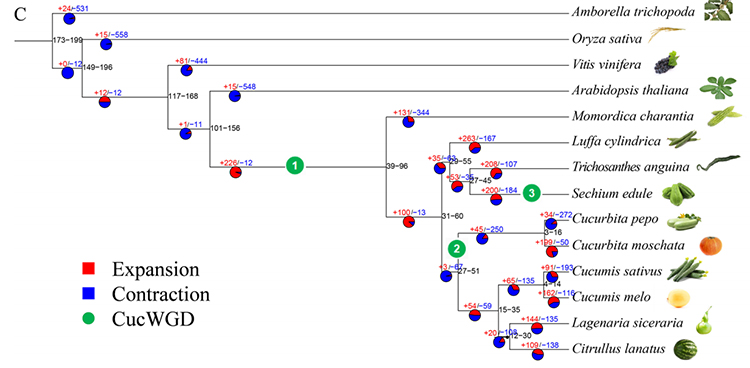
Fu A et al.,Kertészeti Kutatás, 2021
3. Szintén elemzés: A gyümölcsfejlődésben a fitohormonokhoz kapcsolódó géneket találtak a chayote-ban, a kígyótökben és a tökben.A chayote és a squash közötti összefüggés valamivel magasabb, mint a chayote és a kígyótök között.
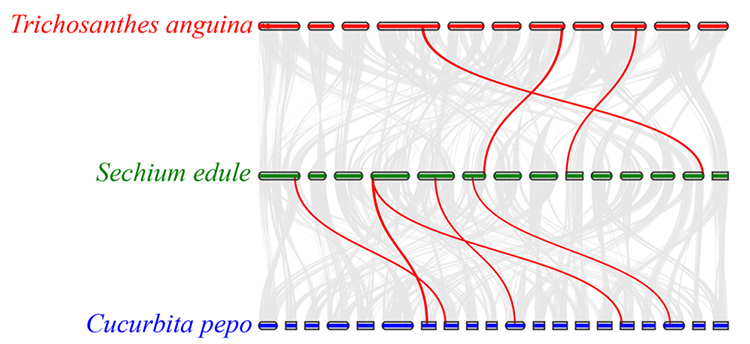
Fu A et al.,Kertészeti Kutatás, 2021
4. Géncsalád analízis: KEGG dúsítás a géncsalád expanzióján és összehúzódásán G.thurberi és G.davidsonii genomban kimutatta, hogy a szteroid bioszintézissel és a brassinoszteroid bioszintézissel kapcsolatos gének bővültek.
Yang Z et al.,BMC biológia, 2021
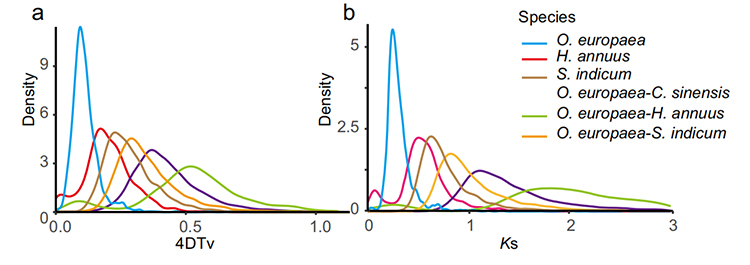
5. Teljes genom duplikációs analízis: A 4DTV és a Ks eloszlás elemzése teljes genom duplikációs eseményt mutatott ki.A fajon belüli csúcsok duplikációs eseményeket mutattak.A fajok közötti csúcsok faji eseményeket mutattak.Az elemzés azt mutatta, hogy a másik három közeli rokon fajhoz képest az O. europaea újabban nagy léptékű génduplikáción ment keresztül.

Rao G és társai,Kertészeti Kutatás, 2021
BMK tok
Rózsa tüskék nélkül: genomikai betekintés a nedvességhez való alkalmazkodáshoz
Közzétett: Országos Tudományos Szemle, 2021
Szekvenálási stratégia:
– BasyéTövistelen' (R.Wichurainan) genom:
kb.93 X PacBio + kb.90 X Nanopore + 267 X Illumina
Főbb eredmények
1. A jó minőségű R.wichuraiana genomot régóta olvasott szekvenálási technikákkal állítottuk elő, amelyek 530,07 Mb-os összeállítást eredményeznek (a becsült genomméret körülbelül 525,9 Mb áramlási citometriával és 525,5 Mb genomfelvétellel. A heterozigótaság körülbelül 1,03%).A BUSCO becsült pontszáma 93,9% volt.Az „Old blush”-hez (haploOB) összehasonlítva ennek a genomnak a minőségét és teljességét a bázis egybázisú pontossága és az LTR összeállítási index (LAI=20,03) igazolta.Az R.wichuraiana genomja 32 674 fehérjét kódoló gént tartalmaz.
2. Az összehasonlító genomikából, transzkriptomikából és a genetikai populáció QTL analíziséből álló multi-omics közös elemzés feltárta a R. wichuraiana és a Rosa chinensis közötti döntő fajlagosságot.Valószínűleg a QTL-ben a rokon gének expressziós variációja is összefüggésbe hozható a szár tüskék mintázatával.

A Basye's Thornless és Rosa chinensis összehasonlító genomikai elemzése, beleértve a szinténelemzést, a géncsalád klaszterét, az expanziós és összehúzódási analízist, nagyszámú variációt tárt fel, amelyek a rózsák döntő tulajdonságaihoz kapcsolódnak.A NAC és a FAR1/FRS géncsalád egyedülálló bővülése nagy valószínűséggel összefüggésbe hozható a fekete foltokkal szembeni rezisztenciával.
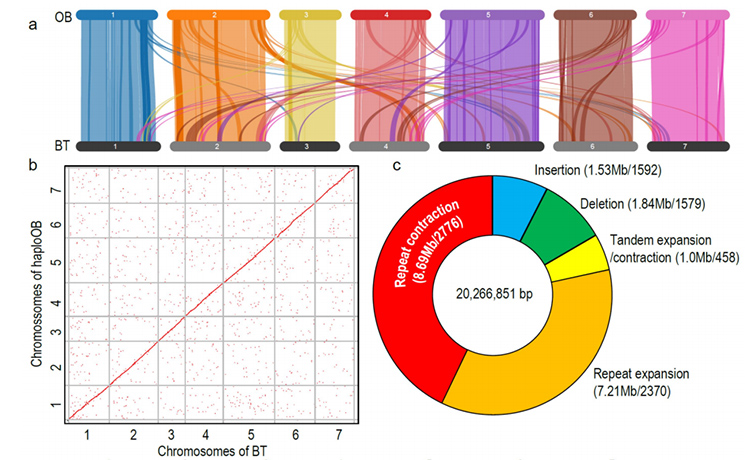

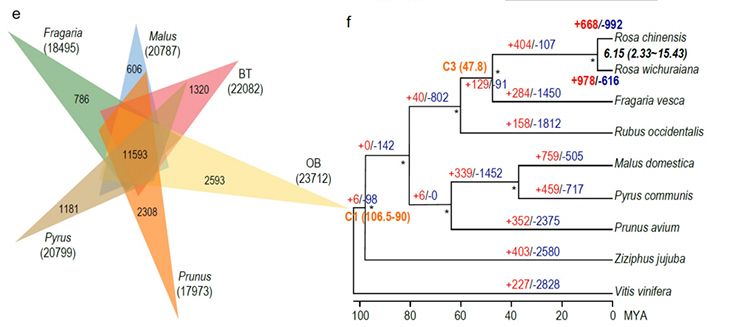
Összehasonlító genomikai elemzés a BT és a haploOB genomok között.
Zhong, M. és mtsai.„Rózsa tüskék nélkül: genomikai betekintés a nedvességhez való alkalmazkodáshoz”Országos Tudományos Szemle, 2021;, nwab092.