Teljes hosszúságú mRNS szekvenálás - nanopórus
Szolgáltatás előnyei
● Alacsony sorrendi torzítás
● Teljes hosszúságú cDNS-molekulák feltárása
● Kevesebb adat szükséges ugyanannyi átirat lefedéséhez
● Génenként több izoforma azonosítása
● Kifejezés mennyiségi meghatározása izoforma szinten
Szolgáltatási specifikációk
| Könyvtár | Felület | Javasolt adatmennyiség (Gb) | Minőség ellenőrzés |
| cDNS-PCR (poli-A-val dúsított) | Nanopore PromethION P48 | 6 Gb/minta (fajtól függően) | Teljes hossz aránya: 70% Átlagos minőségi pontszám: Q10
|
Bioinformatikai elemzések
●Nyers adatfeldolgozás
● Átirat azonosítása
● Alternatív toldás
● Az expresszió mennyiségi meghatározása génszinten és izoforma szinten
● Differenciális kifejezés elemzés
● Funkciójegyzetek és -dúsítás (DEG-ek és DET-ek)
Mintakövetelmények és szállítás
Mintakövetelmények:
Nukleotidok:
| Konc. (ng/μl) | Mennyiség (μg) | Tisztaság | Sértetlenség |
| ≥ 100 | ≥ 0,6 | OD260/280=1,7-2,5 OD260/230=0,5-2,5 A gélen korlátozott fehérje- vagy DNS-szennyeződés látható, vagy nincs. | Növényeknél: RIN≥7,0; Állatok esetében: RIN≥7,5; 5,0≥28S/18S≥1,0; korlátozott vagy nincs alapvonali emelkedés |
Szövet: Súly (száraz): ≥1 g
*5 mg-nál kisebb szövetek esetén gyorsfagyasztott (folyékony nitrogénben) szövetminta küldését javasoljuk.
Sejtszuszpenzió: Sejtszám = 3×106- 1×107
* Javasoljuk fagyasztott sejtlizátum szállítását.Abban az esetben, ha az adott sejtszám kisebb, mint 5×105, folyékony nitrogénben gyorsfagyasztás javasolt, ami mikroextrakciónál előnyösebb.
Vérminták: térfogat ≥1 ml
Javasolt mintaszállítás
Tartály: 2 ml-es centrifugacső (ón fólia nem ajánlott)
Mintacímkézés: Csoport+másolat pl. A1, A2, A3;B1, B2, B3......
Szállítás: 2、Szárazjég: A mintákat zsákokba kell csomagolni, és szárazjégbe kell eltemetni.
- RNS-stabil csövek: Az RNS-minták RNS-stabilizáló csőben (pl. RNAstable®) száríthatók és szobahőmérsékleten szállíthatók.
Szerviz munkafolyamat
Nukleotidok:

Mintaszállítás

Könyvtárépítés

Sorrendezés

Adatelemzés

Értékesítés utáni szolgáltatások
Szerviz munkafolyamat
Szövet:

Kísérleti tervezés

Mintaszállítás

RNS extrakció

Könyvtárépítés

Sorrendezés

Adatelemzés

Értékesítés utáni szolgáltatások
1.Differenciális expressziós elemzés - Vulkán plot
A differenciális expressziós analízis feldolgozható mind génszinten a differenciálisan expresszált gének (DEG) azonosítására, mind izoforma szinten a differenciális azonosítás érdekében
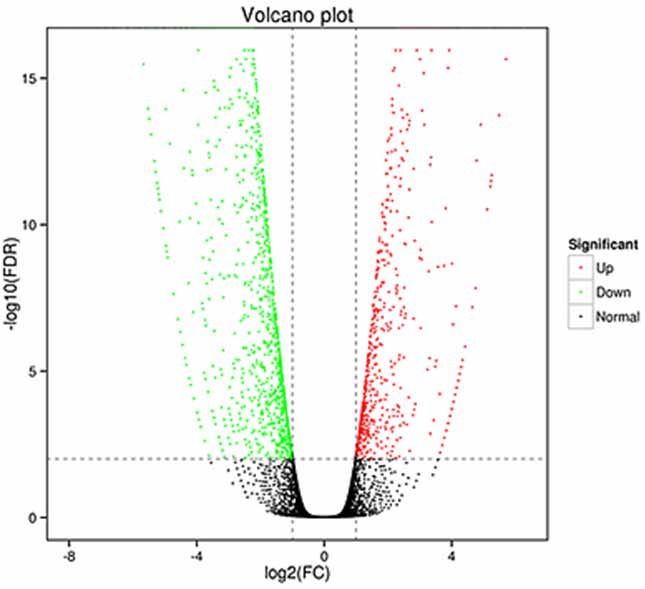
kifejezett átiratok (DET)
2.Hierarchikus klaszterezési hőtérkép
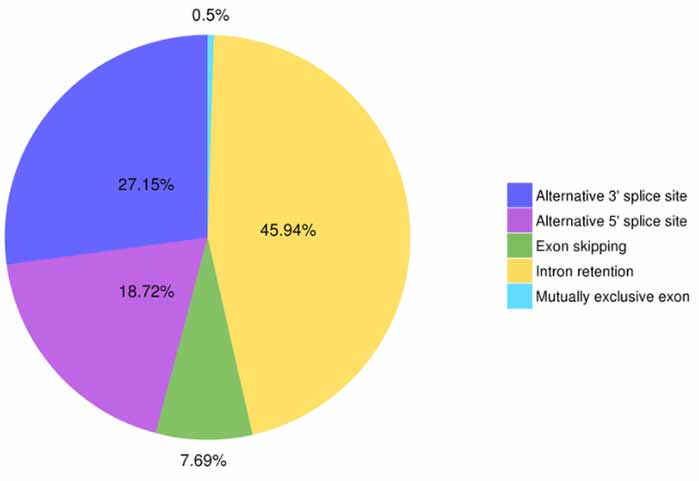
3. Alternatív splicing azonosítás és osztályozás
Az Astalavista ötféle alternatív splicing eseményt tud előre jelezni.
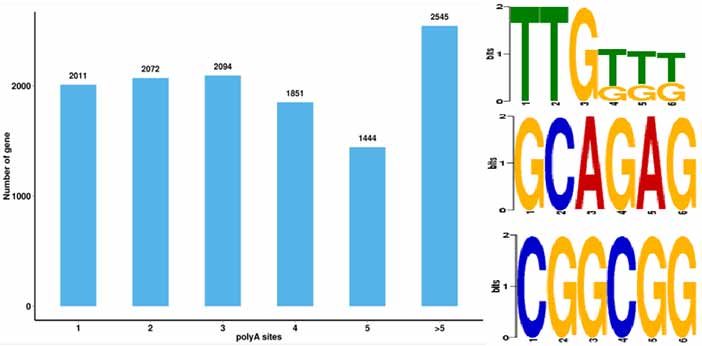
4.Alternatív poli-adenilációs (APA) események azonosítása és motívuma a poli-A-tól 50 bp-vel feljebb
BMK tok
Alternatív splicing azonosítás és izoforma szintű kvantifikáció nanopórusos teljes hosszúságú transzkriptom szekvenálással
Közzétett:Nature Communications, 2020
Szekvenálási stratégia:
Csoportosítás: 1. CLL-SF3B1(WT);2. CLL-SF3B1 (K700E mutáció);3. Normál B-sejtek
Szekvenálási stratégia: MinION 2D könyvtár szekvenálás, PromethION 1D könyvtár szekvenálás;röviden leolvasható adatok ugyanazon mintákból
Szekvenáló platform: Nanopore MinION;Nanopore PromethION;
Főbb eredmények
1.Izoform-szintű alternatív splicing azonosítás
A hosszan olvasott szekvenciák lehetővé teszik a mutáns SF3B1 azonosításátK700E-változott illesztési helyek izoforma szinten.35 alternatív 3′SS és 10 alternatív 5′SS szignifikánsan eltérő splicingot találtak az SF3B1 közöttK700Eés SF3B1WT.A 35 elváltozás közül 33-at újonnan fedeztek fel régóta olvasott szekvenciák.
2.Izoform szintű Alternatív Splicing kvantifikáció
Intronretenciós (IR) izoformák kifejeződése SF3B1-benK700Eés SF3B1WTnanopórusos szekvenciák alapján számszerűsítették, ami az SF3B1 IR izoformáinak globális leszabályozását tárta fel.K700E.
Referencia
Tang AD, Soulette CM, Baren MJV és mások.Az SF3B1 mutáció teljes hosszúságú transzkriptum jellemzése krónikus limfocitás leukémiában a megtartott intronok csökkenését mutatja [J].Nature Communications.