Analisis Asosiasi Seluruh Genom
1. Keuntungan Layanan
● Kasus Proyek Berlimpah: sejak didirikan pada tahun 2009, BMKGENE telah menyelesaikan ratusan proyek spesies dalam penelitian GWAS populasi, membantu peneliti menerbitkan lebih dari 100 artikel, dan faktor dampak kumulatif mencapai 500.
● Analis profesional.
● Siklus analisis yang singkat.
● Penambangan data yang akurat.
2. Spesifikasi Layanan
| Jenis | Skala Populasi | Strategi pengurutan dan kedalaman |
| SLAF-GWAS | Nomor sampel ≥200 | Ukuran genom <400M, dengan ref-genom, WGS direkomendasikan |
| Ukuran genom ≤ 1G, Tag 100K dan 10X | ||
| 1G ≤ Ukuran genom ≤ 2G, Tag 200K dan 10X | ||
| Ukuran genom> 2G, Tag 300K dan 10X | ||
| WGS-GWAS | Nomor sampel ≥200 | 10X untuk setiap sampel |
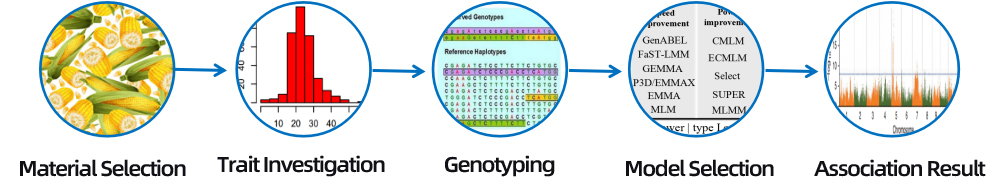
3. Pemilihan Bahan



Varietas, subspesies, ras lokal/bank gen/keluarga campuran/sumber daya alam yang berbeda
Varietas yang berbeda, subspesies, ras lokal
Keluarga saudara tiri/keluarga saudara kandung/sumber daya liar
4. Analisis Bioinformasi
● Analisis asosiasi genom secara luas
● Analisis dan penyaringan SNP yang signifikan
● Anotasi fungsional gen kandidat
5. Alur Kerja Pelayanan

Desain percobaan

Pengiriman sampel

Ekstraksi RNA

Pembangunan perpustakaan

Pengurutan

Analisis data

Layanan purna jual
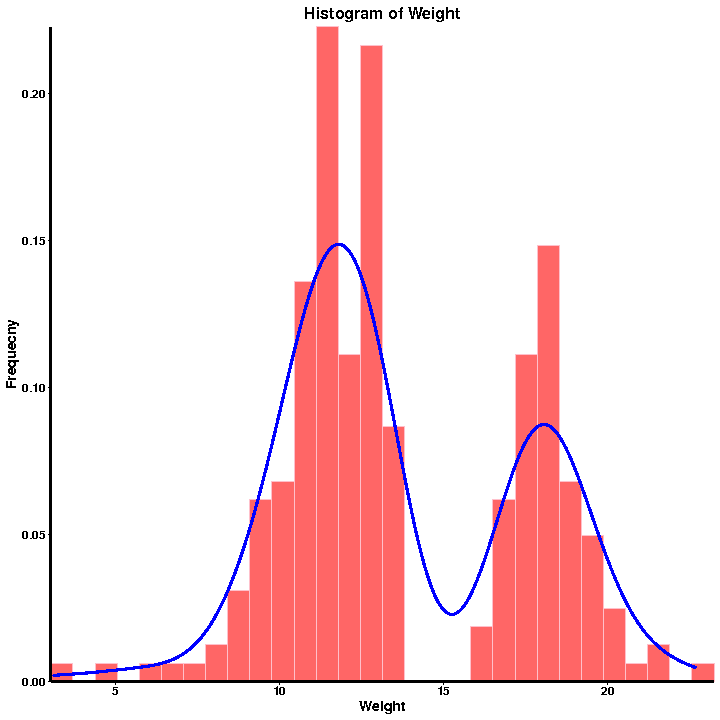
A.QC fenotipe
Histogram distribusi frekuensi
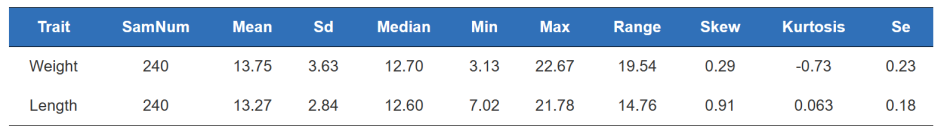
Statistik fenotip
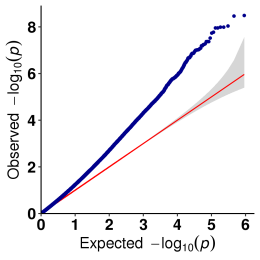
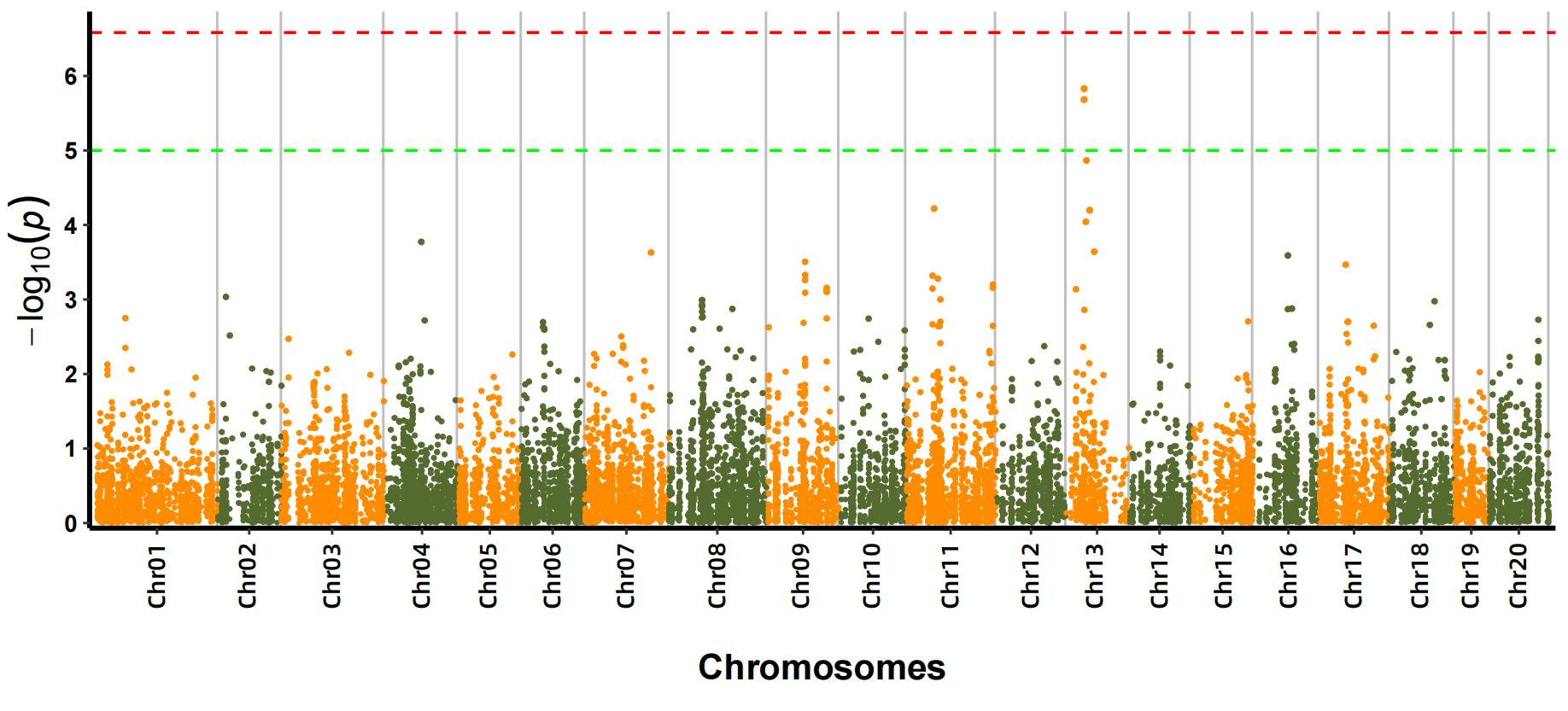
B.Analisis asosiasi (Model: GEMMA, FaST-LMM, EMMAX)
Plot QQ
Plot Manhattan
| Tahun | Jurnal | IF | Judul |
| 2022 | NC | 17.69 | Dasar genom giga-kromosom dan giga-genom pohon peoni Paeonia ostii |
| 2015 | NP | 7.43 | Jejak domestikasi menjadi penanda wilayah genom yang memiliki kepentingan agronomi pada kedelai |
| 2018 | MP | 9.32 | Pengurutan ulang seluruh genom dari koleksi aksesi rapeseed di seluruh dunia mengungkapkan dasar genetik dari perbedaan ekotipe mereka |
| 2022 | HR | 7.29 | Analisis asosiasi genom memberikan wawasan molekuler tentang variasi alami ukuran biji semangka |