Sequenziamento dell'mRNA eucariotico-Illumina
Vantaggi
● Alta esperienza: oltre 200.000 campioni sono stati elaborati in BMK coprendo diversi tipi di campioni, tra cui colture cellulari, tessuti, fluidi corporei, ecc. e oltre 7.000 progetti mRNA-Seq chiusi che coprono varie aree di ricerca.
● Rigoroso sistema di controllo della qualità: i principali punti di controllo della qualità in tutte le fasi, tra cui la preparazione dei campioni, la preparazione delle librerie, il sequenziamento e la bioinformatica, sono sotto stretto monitoraggio al fine di fornire risultati di alta qualità.
● Diversi database disponibili per l'annotazione delle funzioni e gli studi di arricchimento per soddisfare diversi obiettivi di ricerca.
● Servizi post-vendita: servizi post-vendita validi per 3 mesi dal completamento del progetto, inclusi follow-up dei progetti, risoluzione dei problemi, domande e risposte sui risultati, ecc.
Requisiti e consegna del campione
| Biblioteca | Strategia di sequenziamento | Dati consigliati | Controllo di qualità |
| Poli A arricchito | Illumina PE150 | 6 GB | Q30≥85% |
Requisiti del campione:
Nucleotidi:
| Concentrazione (ng/μl) | Quantità (μg) | Purezza | Integrità |
| ≥ 20 | ≥ 0,5 | DE260/280=1,7-2,5 DE260/230=0,5-2,5 Contaminazione limitata o assente di proteine o DNA mostrata sul gel. | Per gli impianti: RIN≥6,5; Per gli animali: RIN≥7,0; 5,0≥28S/18S≥1,0; elevazione della linea di base limitata o assente |
Tessuto: Peso (asciutto):≥1 g
*Per tessuti di peso inferiore a 5 mg, si consiglia di inviare campioni di tessuto congelati istantaneamente (in azoto liquido).
Sospensione cellulare:Conteggio delle cellule = 3×106-1×107
*Si consiglia di spedire il lisato cellulare congelato.Nel caso in cui la cella conteggi inferiore a 5×105.
Campioni di sangue:Volume≥1ml
Microrganismo:Massa ≥ 1 g
Consegna del campione consigliata
Contenitore: provetta da centrifuga da 2 ml (la carta stagnola non è consigliata)
Etichettatura del campione: Gruppo+replica, ad esempio A1, A2, A3;B1, B2, B3... ...
Spedizione:
- Ghiaccio secco: i campioni devono essere confezionati in sacchetti e sepolti nel ghiaccio secco.
- Provette RNAstable: i campioni di RNA possono essere essiccati in provette di stabilizzazione dell'RNA (ad esempio RNAstable®) e spediti a temperatura ambiente.
Flusso di lavoro del servizio

Progettazione dell'esperimento

Consegna del campione

Estrazione dell'RNA

Costruzione della biblioteca

Sequenziamento

Analisi dei dati

Servizi post-vendita
Bioinformatica
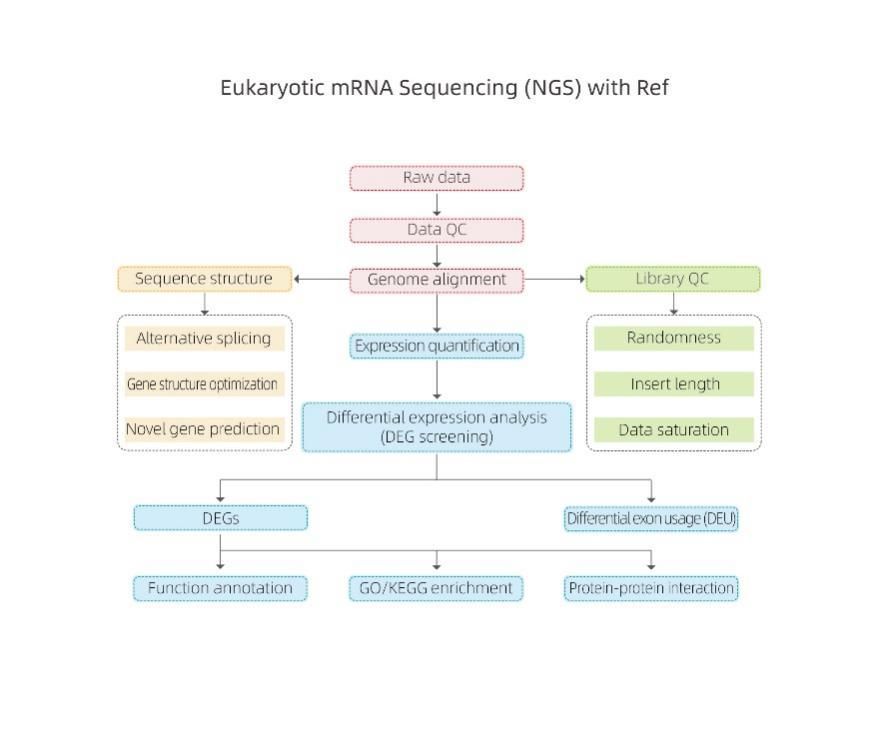
Eucariotico Flusso di lavoro dell'analisi del sequenziamento dell'mRNA
Bioinformatica
ØControllo della qualità dei dati grezzi
ØAllineamento del genoma di riferimento
ØAnalisi della struttura della trascrizione
ØQuantificazione dell'espressione
ØAnalisi dell'espressione differenziale
ØAnnotazione e arricchimento di funzioni
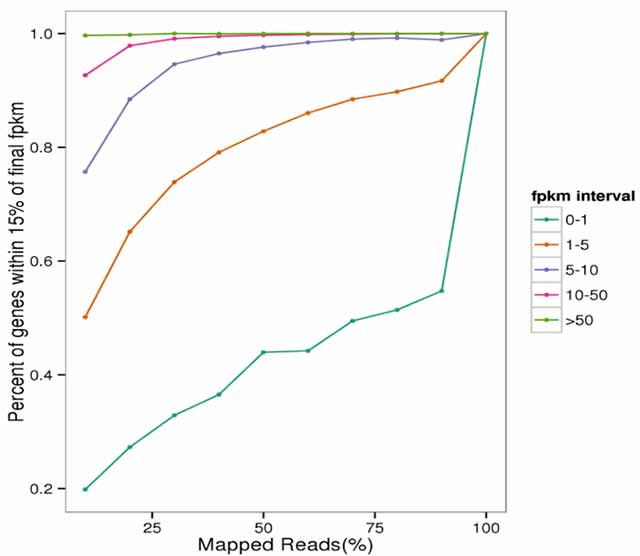
1.Curva di saturazione dei dati dell'mRNA
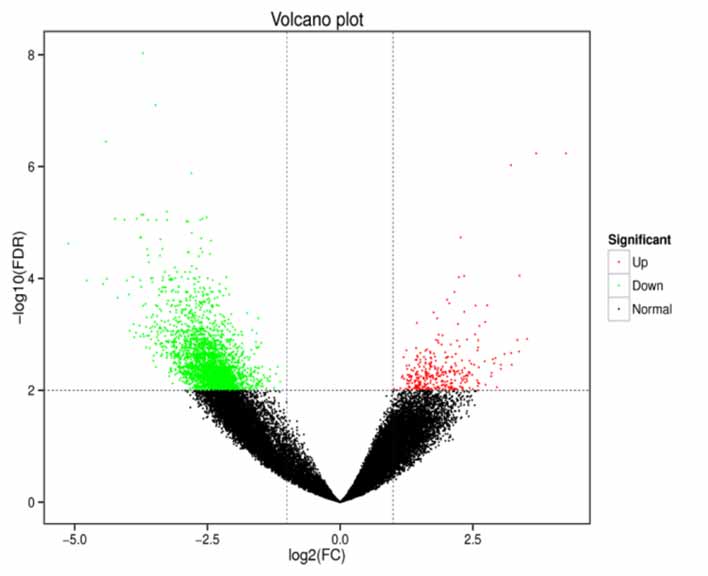
2.Analisi dell'espressione differenziale-grafico del vulcano
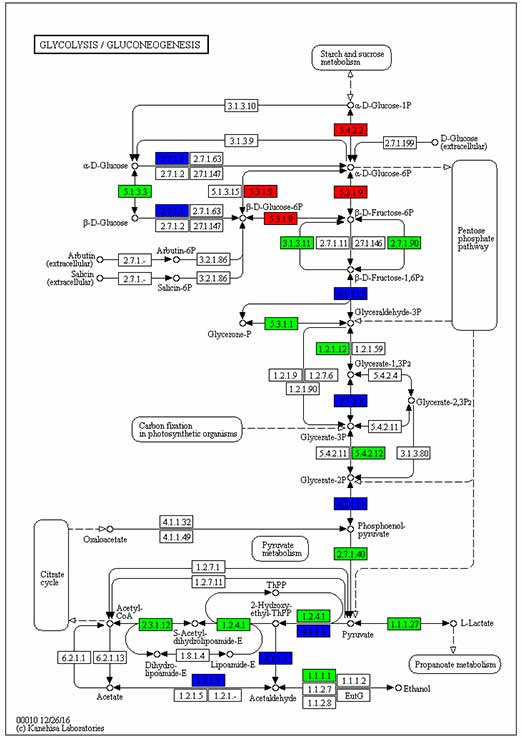
3.Annotazione KEGG sui DEG
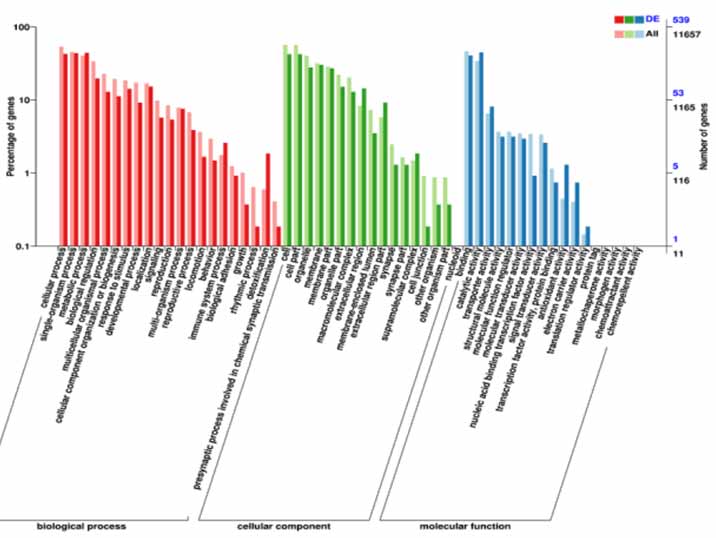
4.Classificazione GO sui DEG