Nanoporo per sequenziamento dell'mRNA a lunghezza intera
Vantaggi del servizio
● Basso bias di sequenza
● Rivelazione di molecole di cDNA a lunghezza intera
● Meno dati richiesti per coprire lo stesso numero di trascrizioni
● Identificazione di più isoforme per gene
● Quantificazione dell'espressione a livello di isoforma
Specifiche del servizio
| Biblioteca | piattaforma | Resa dati consigliata (Gb) | Controllo di qualità |
| cDNA-PCR (arricchito con Poli-A) | Nanoporo PromethION P48 | 6 Gb/campione (a seconda della specie) | Rapporto a tutta lunghezza>70% Punteggio di qualità medio: Q10
|
Analisi bioinformatiche
●Elaborazione dei dati grezzi
● Identificazione della trascrizione
● Giunzioni alternative
● Quantificazione dell'espressione a livello genico e isoforme
● Analisi dell'espressione differenziale
● Annotazione e arricchimento delle funzioni (DEG e DET)
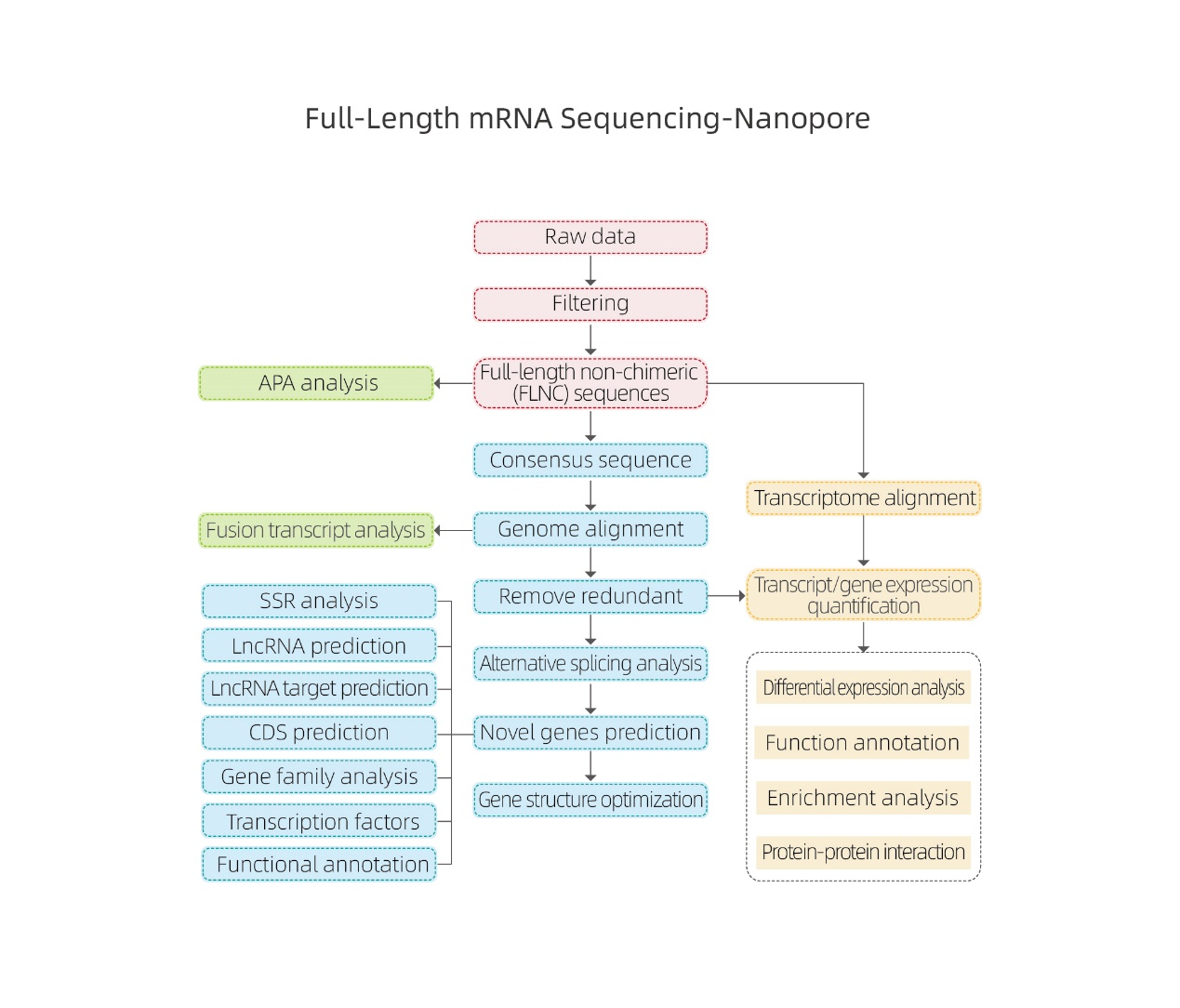
Requisiti e consegna del campione
Requisiti del campione:
Nucleotidi:
| Concentrazione (ng/μl) | Quantità (μg) | Purezza | Integrità |
| ≥ 100 | ≥ 0,6 | DE260/280=1,7-2,5 DE260/230=0,5-2,5 Contaminazione limitata o assente di proteine o DNA mostrata sul gel. | Per gli impianti: RIN≥7,0; Per gli animali: RIN≥7,5; 5,0≥28S/18S≥1,0; elevazione della linea di base limitata o assente |
Tessuto: Peso (secco): ≥1 g
*Per tessuti di peso inferiore a 5 mg, si consiglia di inviare campioni di tessuto congelati istantaneamente (in azoto liquido).
Sospensione cellulare: conta cellulare = 3×106-1×107
*Si consiglia di spedire il lisato cellulare congelato.Nel caso in cui la cella conteggi inferiore a 5×105, si consiglia il congelamento rapido in azoto liquido, preferibile per la microestrazione.
Campioni di sangue: volume≥1 ml
Consegna del campione consigliata
Contenitore: provetta da centrifuga da 2 ml (la carta stagnola non è consigliata)
Etichettatura del campione: Gruppo+replica, ad esempio A1, A2, A3;B1, B2, B3... ...
Spedizione: 2、Ghiaccio secco: i campioni devono essere imballati in sacchetti e sepolti in ghiaccio secco.
- Provette RNAstable: i campioni di RNA possono essere essiccati in provette di stabilizzazione dell'RNA (ad esempio RNAstable®) e spediti a temperatura ambiente.
Flusso di lavoro del servizio
Nucleotidi:

Consegna del campione

Costruzione della biblioteca

Sequenziamento

Analisi dei dati

Servizi post-vendita
Flusso di lavoro del servizio
Tessuto:

Progettazione dell'esperimento

Consegna del campione

Estrazione dell'RNA

Costruzione della biblioteca

Sequenziamento

Analisi dei dati

Servizi post-vendita
1.Analisi dell'espressione differenziale -Vulcano plot
L'analisi dell'espressione differenziale può essere elaborata sia a livello genico per identificare i geni espressi in modo differenziale (DEG) sia a livello di isoforma per identificare in modo differenziale
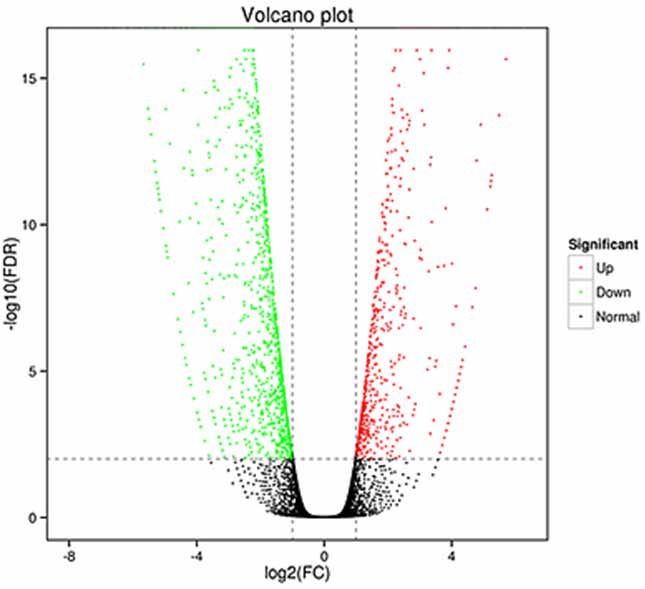
trascrizioni espresse (DET)
2.Mappa termica del clustering gerarchico
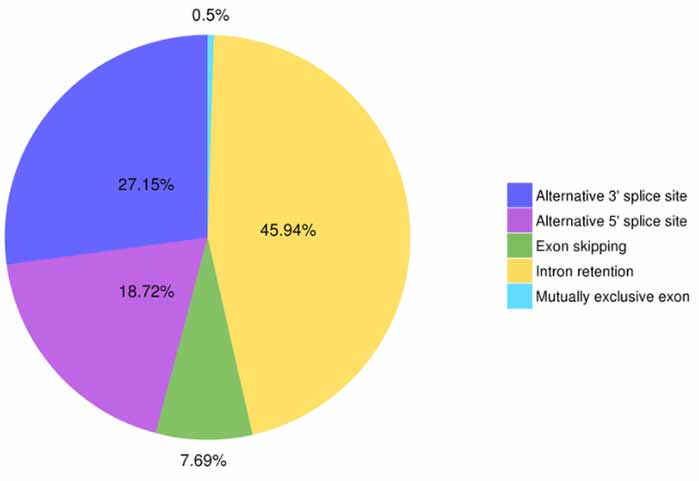
3.Identificazione e classificazione degli splicing alternativi
Astalavista può prevedere cinque tipi di eventi di splicing alternativo.
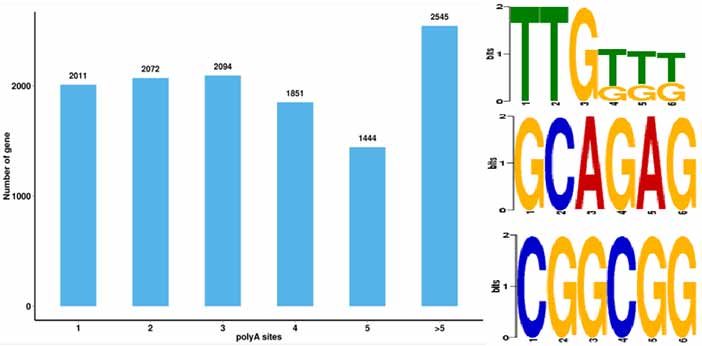
4.Identificazione di eventi alternativi di poli-adenilazione (APA) e motivo a 50 bp a monte del poli-A
Caso BMK
Identificazione dello splicing alternativo e quantificazione a livello di isoforma mediante sequenziamento del trascrittoma a lunghezza intera di nanopori
Pubblicato:Comunicazioni sulla natura, 2020
Strategia di sequenziamento:
Raggruppamento: 1. CLL-SF3B1(WT);2. CLL-SF3B1 (mutazione K700E);3. Cellule B normali
Strategia di sequenziamento: sequenziamento della libreria 2D MinION, sequenziamento della libreria 1D PromethION;dati di lettura breve dagli stessi campioni
Piattaforma di sequenziamento: Nanopore MinION;PromethION di nanopori;
Risultati chiave
1. Identificazione dello splicing alternativo a livello di isoforma
Le sequenze a lunga lettura consentono l'identificazione del mutante SF3B1K700E-siti di giunzione alterati a livello di isoforma.È stato scoperto che 35 3′SS alternativi e 10 5′SS alternativi erano significativamente differenziati tra SF3B1K700Ee SF3B1WT.33 delle 35 alterazioni sono state scoperte di recente da sequenze di lettura prolungata.
2.Quantificazione dello splicing alternativo a livello di isoforma
Espressione delle isoforme di ritenzione degli introni (IR) in SF3B1K700Ee SF3B1WTsono stati quantificati sulla base di sequenze di nanopori, rivelando una down-regolazione globale delle isoforme IR in SF3B1K700E.
Riferimento
Tang AD, Soulette CM, Baren MJV, e al.La caratterizzazione della trascrizione completa della mutazione SF3B1 nella leucemia linfocitica cronica rivela una sottoregolazione degli introni trattenuti[J].Comunicazioni sulla natura.