Sequenziamento dell'intero esoma umano
Vantaggi del servizio
● Regione codificante delle proteine mirata: catturando e sequenziando la regione codificante delle proteine, hWES viene utilizzato per rivelare varianti correlate alla struttura delle proteine;
● Alta precisione: con un'elevata profondità di sequenziamento, hWES facilita il rilevamento di varianti comuni e varianti rare con frequenze inferiori all'1%;
● Conveniente: hWES produce circa l'85% delle mutazioni di malattie umane dall'1% del genoma umano;
● Cinque rigorose procedure di controllo qualità che coprono l'intero processo con Q30>85% garantito.
Specifiche del campione
| piattaforma
| Biblioteca
| Strategia di cattura degli esoni
| Consigliare la strategia di sequenziamento
|
|
Piattaforma Illumina NovaSeq
| PE150 | Agilent SureSelect Human All Exon V6 Pannello IDT xGen Exome Hyb V2 | 5 GB 10 GB |
Requisiti del campione
| Tipo di campione
| Quantità(Qubit®)
| Volume
| Concentrazione
| Purezza(NanoDrop™) |
|
DNA genomico
| ≥ 300ng | ≥ 15 µl | ≥ 20 ng/μL | DE260/280=1,8-2,0 nessun degrado, nessuna contaminazione
|
Profondità di sequenziamento consigliata
Per disturbi mendeliani/malattie rare: profondità di sequenziamento effettiva superiore a 50×
Per campioni tumorali: profondità di sequenziamento effettiva superiore a 100×
Bioinformatica


Flusso di lavoro del servizio

Consegna del campione

Estrazione del DNA

Costruzione della biblioteca

Sequenziamento

Analisi dei dati

Consegna dei dati

Servizi post-vendita
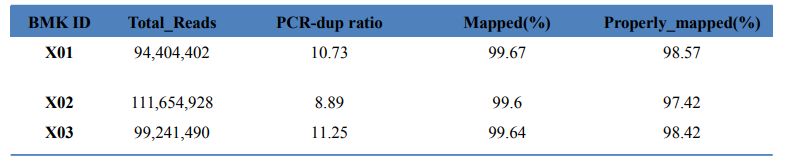
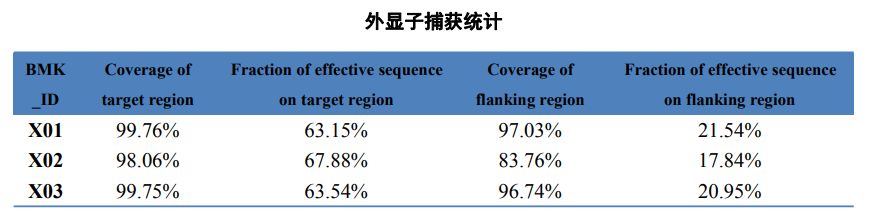
1.Statistiche di allineamento
Tabella 1 Statistiche dei risultati della mappa
Tabella 2 Statistiche sulla cattura dell'esoma
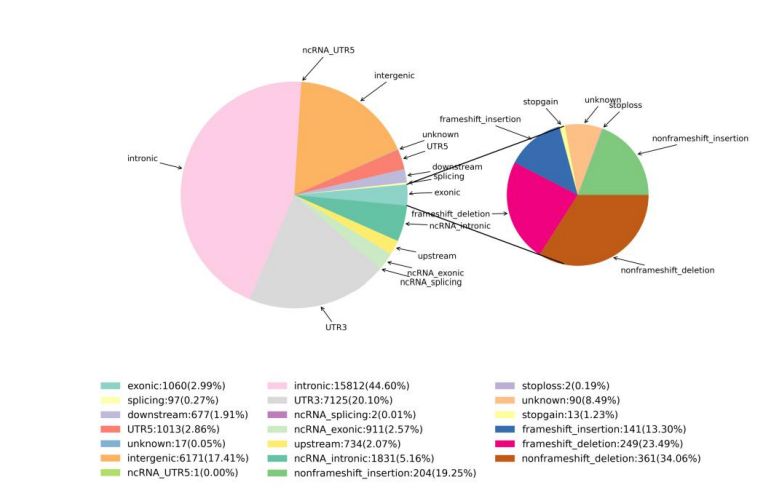
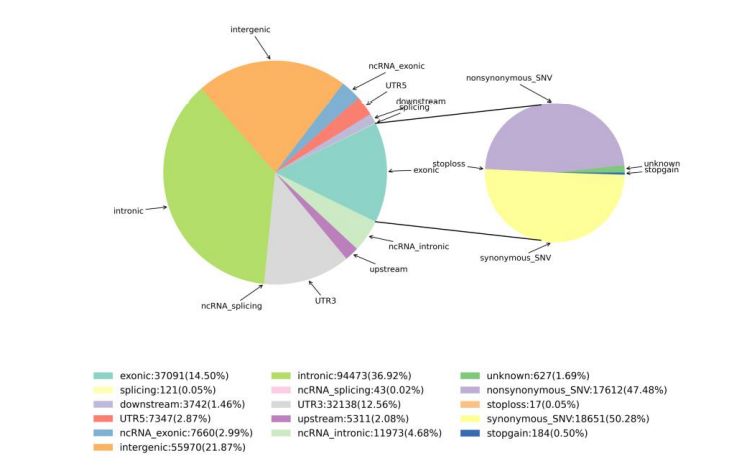
2.Rilevamento delle variazioni
Figura 1 Statistiche di SNV e InDel
3.Analisi avanzata
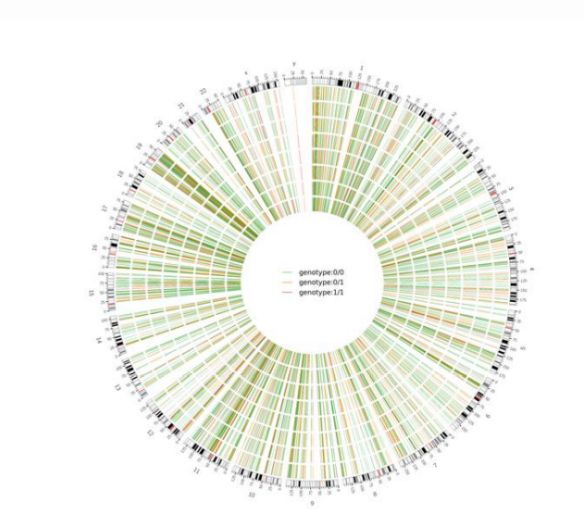
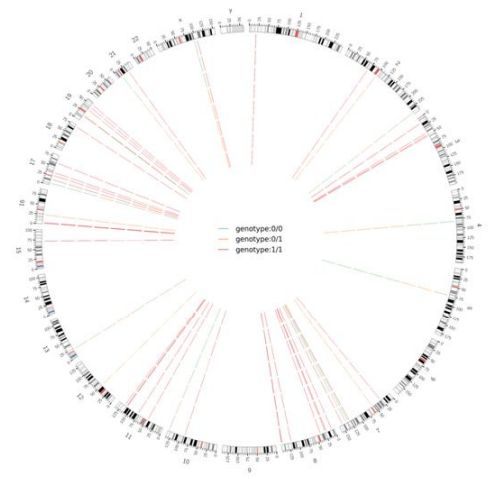
Figura 2 Grafico Circos di SNV e InDel dannosi a livello genomico
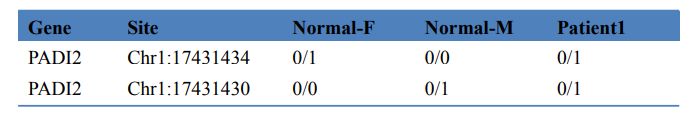
Tabella 3 Screening dei geni che causano malattie