
רצף הגנום של צמח/חיה דה נובו
יתרונות השירות

פיתוח פלטפורמות רצף וביואינפורמטיקה בדה נובוהרכבת הגנום
(Amarasinghe SL וחב',ביולוגיה של הגנום, 2020)
● בניית גנומים חדשים ושיפור גנומי ייחוס קיימים עבור מינים בעלי עניין.
● דיוק גבוה יותר, המשכיות ושלמות בהרכבה
● בניית משאב בסיסי למחקר בפולימורפיזם של רצף, QTLs, עריכת גנים, רבייה וכו'.
● מצויד בספקטרום מלא של פלטפורמות רצף מהדור השלישי: פתרון להרכבת גנום חד פעמית
● אסטרטגיות רצף והרכבה גמישות הממלאות גנומים מגוונים עם תכונות שונות
● צוות ביואינפורמטיקה מיומן ביותר עם ניסיון רב במכלולי גנום מורכבים, לרבות פוליפלואידים, גנומים ענקיים וכו'.
● מעל 100 מקרים מוצלחים עם מקדם השפעה מצטבר של למעלה מ-900
● זמן אספקה מהיר כמו 3 חודשים להרכבת גנום ברמת הכרומוזום.
● תמיכה טכנית מוצקה עם סדרה של פטנטים וזכויות יוצרים בתוכנה הן בצד הניסיוני והן בביואינפורמטיקה.
מפרט שירות
|
תוֹכֶן
|
פּלַטפוֹרמָה
|
אורך קריאה
|
כיסוי
|
| סקר הגנום
| Illumina NovaSeq
| PE150
| ≥ 50X
|
| רצף הגנום
| PacBio Revio
| קריאת HiFi של 15 kb
| ≥ 30X
|
| היי-סי
| Illumina NovaSeq
| PE150
| ≥100X
|
זרימת עבודה

דרישות לדוגמא ומשלוח
דרישות לדוגמה:
| מִין | רִקמָה | עבור PacBio | עבור ננופור |
| בעלי חיים | איברים קרביים (כבד, טחול וכו') | ≥ 1.0 גרם | ≥ 3.5 גרם |
| שְׁרִיר | ≥ 1.5 גרם | ≥ 5.0 גרם | |
| דם של יונקים | ≥ 1.5 מ"ל | ≥ 5.0 מ"ל | |
| דם של דגים או ציפורים | ≥ 0.2 מ"ל | ≥ 0.5 מ"ל | |
| צמחים | עלים טריים | ≥ 1.5 גרם | ≥ 5.0 גרם |
| עלה כותרת או גזע | ≥ 3.5 גרם | ≥ 10.0 גרם | |
| שורשים או זרעים | ≥ 7.0 גרם | ≥ 20.0 גרם | |
| תאים | תרבית תאים | ≥ 3×107 | ≥ 1×108 |
משלוח לדוגמא מומלץ
מיכל: שפופרת צנטריפוגה 2 מ"ל (לא מומלץ נייר כסף)
עבור רוב הדגימות, אנו ממליצים לא לשמר באתנול.
תיוג לדוגמה: דגימות צריכות להיות מסומנות בבירור וזהות לטופס המידע המוגש לדוגמא.
משלוח: קרח יבש: יש לארוז תחילה את הדגימות בשקיות ולקבור אותן בקרח יבש.
זרימת עבודה בשירות

עיצוב ניסוי

משלוח לדוגמא

מיצוי DNA

בניית ספרייה

רצף

ניתוח נתונים

שירותים לאחר המכירה
*תוצאות ההדגמה המוצגות כאן הן כולן מגנומים שפורסמו עם Biomarker Technologies
1.Circos על הרכבה גנום ברמת הכרומוזום שלG. rotundifoliumעל ידי פלטפורמת רצף Nanopore
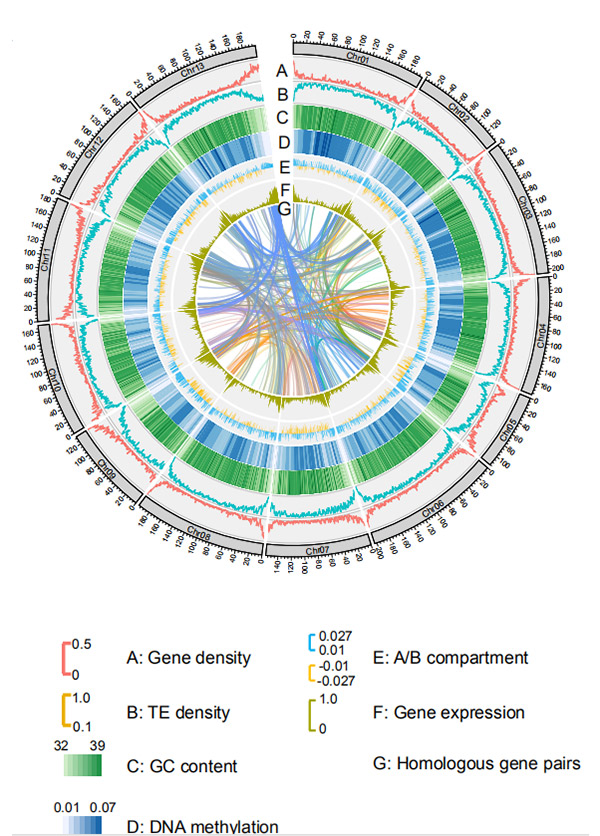
Wang M וחב',ביולוגיה מולקולרית ואבולוציה, 2021
2. סטטיסטיקה של הרכבה והערה של גנום שיפון ווינינג
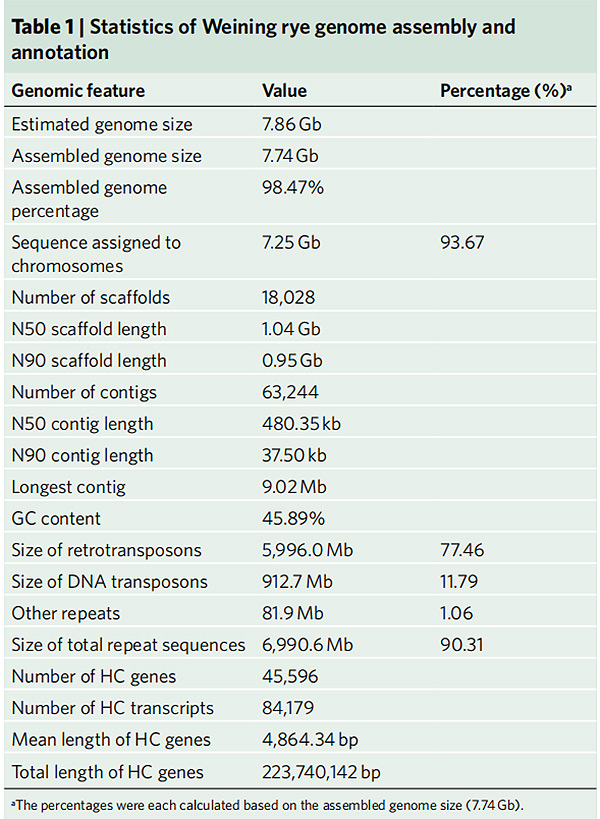
Li G וחב',גנטיקה של הטבע, 2021
3. חיזוי גנים שלSechium eduleגנום, הנגזר משלוש שיטות חיזוי:דה נובוחיזוי, חיזוי מבוסס הומולוגיה וחיזוי מבוסס נתונים RNA-Seq
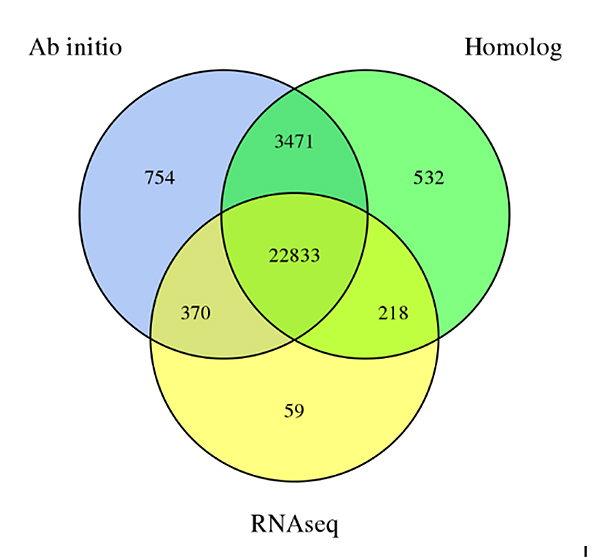
Fu A וחב',מחקר גננות, 2021
4.זיהוי של חזרות ארוכות שלמות בשלושה גנומים מכותנה
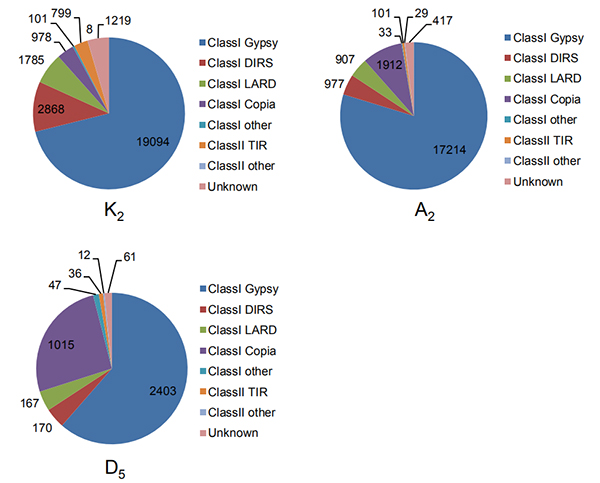
Wang M וחב',ביולוגיה מולקולרית ואבולוציה, 2021
5.Hi-C מפת חום שלC. acuminataגנום המראה אינטראקציות כול-אחר-כל-גנומיות.עוצמת אינטראקציות Hi-C היא פרופורציונלית למרחק הליניארי בין קונטיגים.קו ישר נקי על מפת חום זו מצביע על עיגון מדויק ביותר של קונטיגים בכרומוזומים.(יחס עיגון קונטיג: 96.03%)
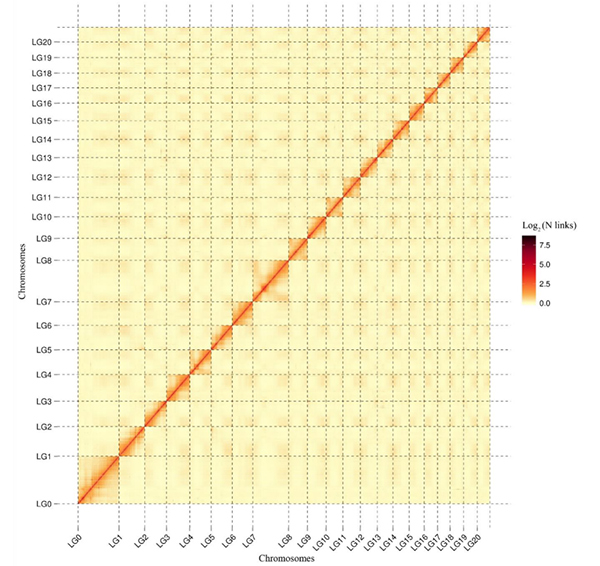
kang M et al.,תקשורת טבע,2021
תיק BMK
מכלול גנום איכותי מדגיש מאפיינים גנומיים של שיפון וגנים חשובים מבחינה אגרונומית
יצא לאור: גנטיקה של הטבע, 2021
אסטרטגיית רצף:
הרכבת גנום: מצב PacBio CLR עם ספרייה של 20 קילו-בייטים (497 ג'יגה-בייט, כ-63×)
תיקון רצף: NGS עם ספריית DNA של 270 bp (430 Gb, בערך 54×) על פלטפורמת Illumina
עיגון קונטיגים: ספריית Hi-C (560 ג'יגה-בייט, כ-71×) על פלטפורמת Illumina
מפה אופטית: (779.55 Gb, כ-99×) ב-Bionano Irys
תוצאות מפתח
1. פורסמה מכלול של גנום שיפון ווינינג עם גודל הגנום הכולל של 7.74 Gb (98.74% מגודל הגנום המשוער לפי ציטומטריית זרימה).פיגום N50 של מכלול זה השיג 1.04 Gb.93.67% מהקונטיגים עוגנו בהצלחה ב-7 פסאודו-כרומוזומים.הרכבה זו הוערכה על ידי מפת הצמדה, LAI ו-BUSCO, מה שהביא לציונים גבוהים בכל ההערכות.
2. מחקרים נוספים על גנומיקה השוואתית, מפת קישור גנטית, מחקרי תעתיק בוצעו על בסיס הגנום הזה.נחשפו סדרה של תכונות גנומיות הקשורות לתכונות, כולל כפילויות של גנים ברחבי הגנום והשפעתם על גנים של ביוסינתזה של עמילן;ארגון פיזי של לוקוסים מורכבים של פרולמין, תכונות ביטוי גנים העומדות בבסיס תכונת הכותרת המוקדמת ואזורים כרומוזומליים משוערים הקשורים לביות בשיפון.
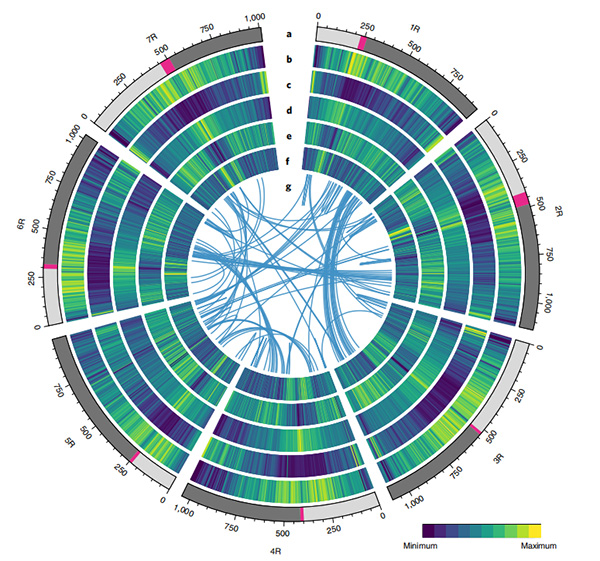 דיאגרמת סירקוס על תכונות גנומיות של גנום שיפון ווינינג | 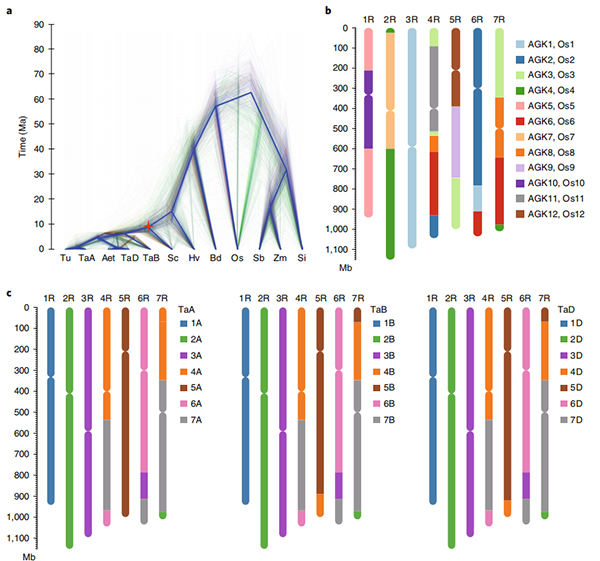 ניתוחים אבולוציוניים וכרומוזומים של גנום השיפון |
Li, G., Wang, L., Yang, J.et al.מכלול גנום איכותי מדגיש מאפיינים גנומיים של שיפון וגנים חשובים מבחינה אגרונומית.נאט ג'נט 53,574–584 (2021).
https://doi.org/10.1038/s41588-021-00808-z










