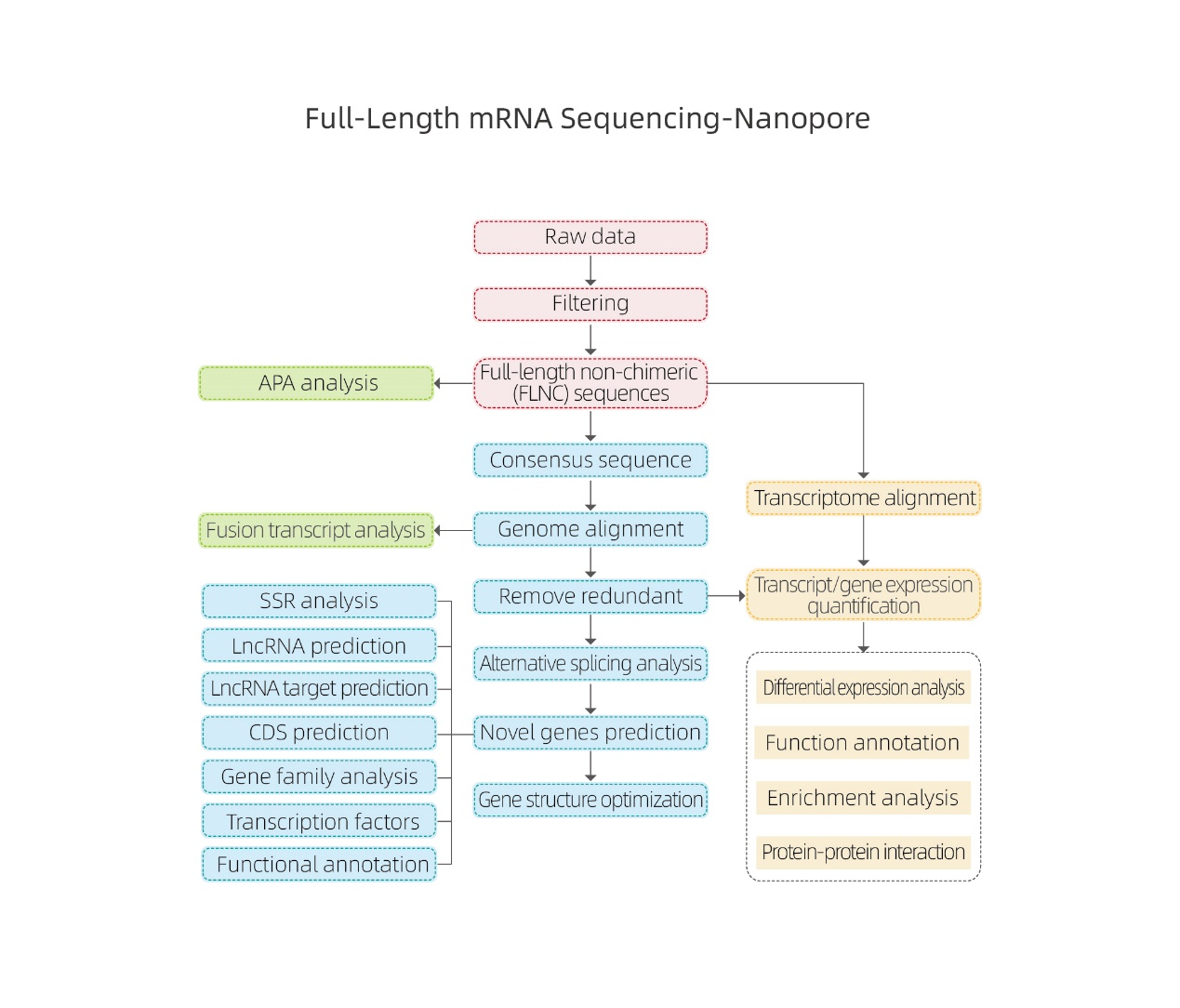
အရှည်အပြည့် mRNA Sequencing-Nanopore
ဝန်ဆောင်မှု အားသာချက်များ
● ဆက်တိုက်ဘက်လိုက်မှု နည်းပါးသည်။
● အစအဆုံး cDNA မော်လီကျူးများကို ဖော်ထုတ်ခြင်း။
● တူညီသော စာသားမှတ်တမ်းများကို ကာမိရန် ဒေတာနည်းပါးသည်။
● မျိုးဗီဇတစ်ခုစီတွင် များစွာသော isoforms များကို ဖော်ထုတ်ခြင်း။
● isoform အဆင့်တွင် ဖော်ပြမှု ပမာဏ
ဝန်ဆောင်မှုသတ်မှတ်ချက်များ
| စာကြည့်တိုက် | ပလပ်ဖောင်း | အကြံပြုထားသော ဒေတာအထွက်နှုန်း (Gb) | အရည်အသွေးထိန်းချုပ်မှု |
| cDNA-PCR (Poly-A ကြွယ်ဝသော) | Nanopore PromethION P48 | 6 Gb/နမူနာ (မျိုးစိတ်ပေါ်မူတည်၍) | ကိုယ်လုံးအချိုး 70% ပျမ်းမျှအရည်အသွေးရမှတ်- Q10
|
Bioinformatics ပိုင်းခြားစိတ်ဖြာချက်များ
●ကုန်ကြမ်းဒေတာ စီမံဆောင်ရွက်ပေးခြင်း။
● စာသားမှတ်တမ်း အထောက်အထား
● အစားထိုး ပေါင်းစည်းခြင်း။
● မျိုးရိုးဗီဇအဆင့်နှင့် isoform အဆင့်တွင် ဖော်ပြမှုပမာဏ
● ကွဲပြားသောအသုံးအနှုန်းခွဲခြမ်းစိတ်ဖြာခြင်း။
● လုပ်ဆောင်ချက် မှတ်ချက်များနှင့် ကြွယ်ဝမှု (DEGs နှင့် DETs)
နမူနာလိုအပ်ချက်များနှင့် ပေးပို့ခြင်း။
နမူနာလိုအပ်ချက်များ-
Nucleotides-
| Conc.(ng/μl) | ပမာဏ (μg) | သန့်ရှင်းစင်ကြယ်ခြင်း။ | သမာဓိ |
| ≥ 100 | ≥ 0.6 | OD260/280=1.7-2.5 OD260/230=0.5-2.5 ဂျယ်တွင်ပြသထားသော ပရိုတင်း သို့မဟုတ် DNA ညစ်ညမ်းမှု အကန့်အသတ် သို့မဟုတ် မရှိပါ။ | အပင်များအတွက် RIN≥7.0; တိရစ္ဆာန်များအတွက် RIN≥7.5; 5.0≥28S/18S≥1.0; အကန့်အသတ် သို့မဟုတ် အခြေခံအဆင့်မြင့်ခြင်း |
တစ်ရှူး- အလေးချိန် (အခြောက်) ≥1 ဂရမ်
* 5 မီလီဂရမ်ထက်သေးငယ်သောတစ်ရှူးအတွက်၊ ဖလက်ရှ်အေးခဲထားသော (နိုက်ထရိုဂျင်အရည်တွင်) တစ်ရှူးနမူနာကို ပေးပို့ရန် အကြံပြုအပ်ပါသည်။
ဆဲလ်ဆိုင်းငံ့မှု- ဆဲလ်အရေအတွက် = 3×106- 1×107
* အေးခဲနေသောဆဲလ် lysate ကိုတင်ပို့ရန်ကျွန်ုပ်တို့အကြံပြုပါသည်။ဆဲလ်သည် 5×10 ထက်ငယ်ပါက ရေတွက်ပါ။5နိုက်ထရိုဂျင်အရည်တွင် အေးခဲထားသည့် flash ကို မိုက်ခရိုထုတ်ယူမှုအတွက် ပိုကောင်းသည်ဟု အကြံပြုထားသည်။
သွေးနမူနာများ- Volume≥1 ml
နမူနာပေးပို့မှု အကြံပြုထားသည်။
ကွန်တိန်နာ- 2 ml centrifuge tube (Tin foil ကို မထောက်ခံပါ)
နမူနာတံဆိပ်ကပ်ခြင်း- အုပ်စု+ဥပမာ A1၊ A2၊ A3၊B1၊ B2၊ B3 ......
ပို့ဆောင်မှု- ၂၊ ရေခဲခြောက်- နမူနာများကို အိတ်များတွင်ထုပ်ပိုးပြီး ရေခဲခြောက်ထဲတွင် မြှုပ်နှံရန် လိုအပ်သည်။
- RNAstable tubes- RNA နမူနာများကို RNA stabilization tube (ဥပမာ RNAstable®) တွင် အခြောက်ခံပြီး အခန်းအပူချိန်တွင် တင်ပို့နိုင်ပါသည်။
Service Work Flow
Nucleotides-

နမူနာပေးပို့ခြင်း။

စာကြည့်တိုက်တည်ဆောက်ရေး

Sequencing

ဒေတာခွဲခြမ်းစိတ်ဖြာ

ရောင်းချပြီးနောက်ဝန်ဆောင်မှုများ
Service Work Flow
တစ်ရှူး-

စမ်းသပ်ထားတာ

နမူနာပေးပို့ခြင်း။

RNA ထုတ်ယူခြင်း။

စာကြည့်တိုက်တည်ဆောက်ရေး

Sequencing

ဒေတာခွဲခြမ်းစိတ်ဖြာ

ရောင်းချပြီးနောက်ဝန်ဆောင်မှုများ
1.Differential expression analysis -Volcano plot
ကွဲပြားသောဖော်ပြသောဗီဇများ (DEGs) ကိုခွဲခြားသတ်မှတ်ရန်နှင့် ကွဲပြားစွာခွဲခြားသတ်မှတ်ရန် isoform အဆင့်တွင် မျိုးရိုးဗီဇအဆင့်နှစ်မျိုးစလုံးတွင် လုပ်ဆောင်နိုင်သည်
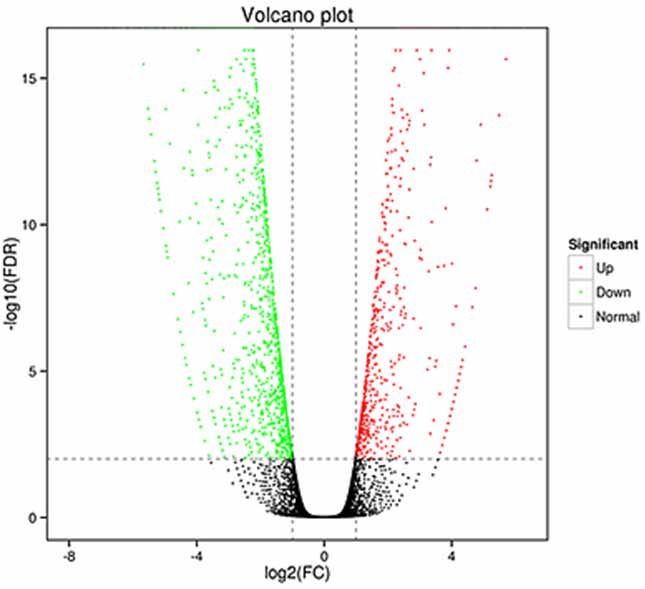
ဖော်ပြထားသော စာသားများ (DETs)
2.အထက်အောက် အစုလိုက်အပြုံလိုက် အပူပြမြေပုံ
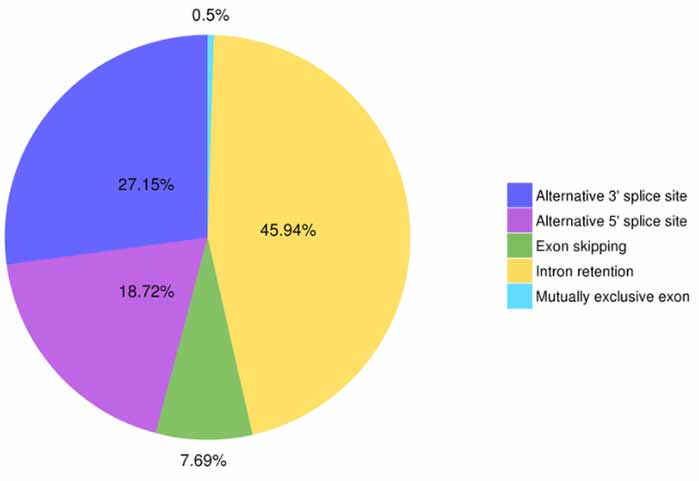
3.Alternative splicing ခွဲခြားသတ်မှတ်ခြင်းနှင့် အမျိုးအစားခွဲခြင်း။
Astalavista မှ အစားထိုးခွဲခြင်းဖြစ်ရပ်ငါးမျိုးအား ခန့်မှန်းနိုင်ပါသည်။
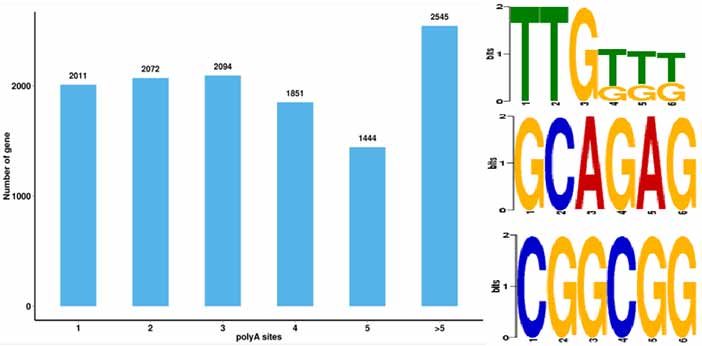
4.အစားထိုး poly-adenylation (APA) ဖြစ်ရပ်များကို ခွဲခြားသတ်မှတ်ခြင်းနှင့် poly-A ၏ 50 bp အထက်တွင်ရှိသော Motif
BMK ဖြစ်ရပ်မှန်
nanopore အစအဆုံး transcriptome sequencing အားဖြင့် အစားထိုး ခွဲခြားသတ်မှတ်ခြင်းနှင့် isoform အဆင့် quantification
ထုတ်ဝေသည်-သဘာဝဆက်သွယ်ရေး၊ 2020
ဆင့်ကဲနည်းဗျူဟာ-
အုပ်စုဖွဲ့ခြင်း- 1. CLL-SF3B1(WT);2. CLL-SF3B1(K700E ပြောင်းလဲမှု);3. ပုံမှန် B-ဆဲလ်များ
Sequencing နည်းဗျူဟာ- MinION 2D စာကြည့်တိုက် စီစစ်ခြင်း၊ PromethION 1D စာကြည့်တိုက် စီစစ်ခြင်း၊တူညီသောနမူနာများမှ အတိုချုံးဖတ်ခြင်းဒေတာ
Sequencing platform- Nanopore MinION;Nanopore PromethION;
အဓိကရလဒ်များ
1.Isoform-level Alternative Splicing Identification
ရှည်လျားစွာဖတ်ထားသော ဆင့်ပွားများသည် မျိုးပြောင်း SF3B1 ကို ဖော်ထုတ်နိုင်စေပါသည်။K700E- isoform အဆင့်တွင် ပြောင်းလဲထားသော splice sites များ။35 အစားထိုး 3′SSs နှင့် 10 အစားထိုး 5′SS များကို SF3B1 အကြား သိသိသာသာ ကွဲပြားစွာ ခွဲထားသည်ကို တွေ့ရှိခဲ့သည်။K700Eနှင့် SF3B1WT.ပြောင်းလဲမှု ၃၅ ခုအနက် ၃၃ ခုကို ရှည်လျားစွာဖတ်ထားသော အတွဲများဖြင့် အသစ်တွေ့ရှိခဲ့သည်။
2.Isoform-level Alternative Splicing quantification
SF3B1 တွင် Intron Retention (IR) isoforms များကို ဖော်ပြခြင်း။K700Eနှင့် SF3B1WTSF3B1 ရှိ IR isoforms များ၏ ကမ္ဘာလုံးဆိုင်ရာ စည်းမျဉ်းများကို ထုတ်ဖော်ပြသသည့် နာနိုပရီဆက်ဆက်များပေါ်တွင် အခြေခံ၍ အရေအတွက်K700E.
အကိုးအကား
Tang AD ၊ Soulette CM ၊ Baren MJV ၊ et al.နာတာရှည် lymphocytic leukemia တွင် SF3B1 ဗီဇပြောင်းလဲမှု၏ အရှည်ဆုံး စာသားမှတ်တမ်းဖော်ပြချက်သည် သိမ်းဆည်းထားသော introns[J] ၏ စည်းမျဉ်းများကို လျှော့ချပေးသည်ကို ဖော်ပြသည်။သဘာဝဆက်သွယ်ရေး။