Plante-/dyre-helgenomsekvensering
1. Tjenestefordeler
Omfattende erfaring med genomsekvensering for over 1000 arter.
Over 800 publiserte saker med en akkumulert effektfaktor på over 4000.
Omfattende bioinformatikkanalyse på variasjonskall og funksjonsanalyse.
2. Tjenestespesifikasjoner
| Plattform | Bibliotek | Anbefalt sekvensdybde | |
| Illumina | PE 150 | For SNP, InDel-anrop ≥ 10x For SV, CNY-anrop ≥ 30x
| |
| Nanopore
| 8 kb | For SV, CNY-anrop ≥ 20x | |
| Pacbio | CCS | 15 kb | For SNP, InDel, SV, CNY-anrop ≥ 10x |
3. Prøvekrav
| Plattform | Kons.(ng/μL)
| Beløp (ng)
| Renhet
| Agarose gel
| ||
| OD260/280 | OD260/230 | 1. Tydelig hovedbånd med ingen eller begrensetnedbrytning observert på gel. 2. Ingen eller begrenset RNA- eller proteinkontaminering
| ||||
| Illumina | ≥1 | ≥30
| - | - | ||
| Nanopore
| ≥30 | Avhenger av datautbytte 10 μg/celle | 1,7-2,2 | ≥1,5 | ||
| Pacbio | CCS | ≥50 | 1,7-2,2 | 1,8-2,5 | ||
4. Bioinformasjonsanalyse
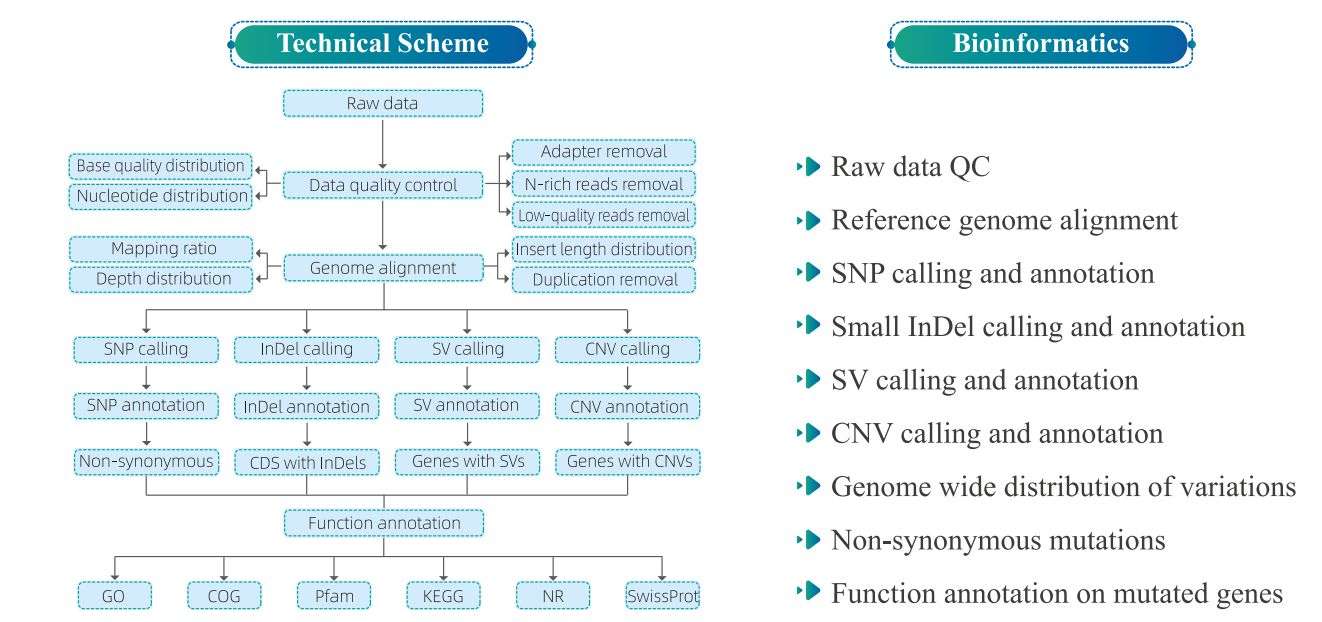
5. Servicearbeidsflyt

Prøvelevering

DNA-ekstraksjon

Bygging av bibliotek

Sekvensering

Dataanalyse

Datalevering
1) Statistikk for genomkartlegging
Tabell 1 Statistikk over kartleggingsresultat
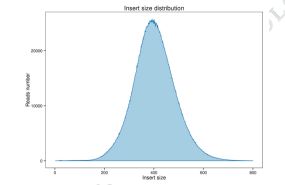
Figur 1 Fordeling av innsatsstørrelse og avlesning av dekning.
2) Variasjonsdeteksjon
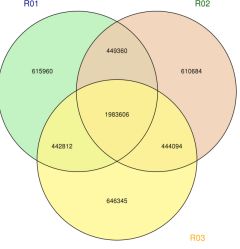
Figur 2 Statistikk og merknad av SNP/INDEL/SV blant prøver
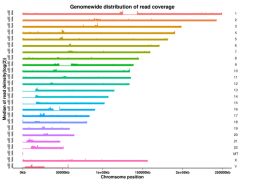
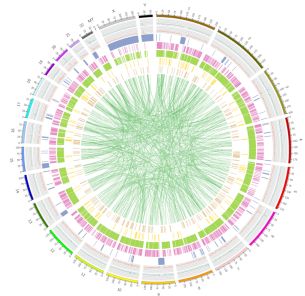
Figur 3 Genom-wide distribusjon av vatiasjoner
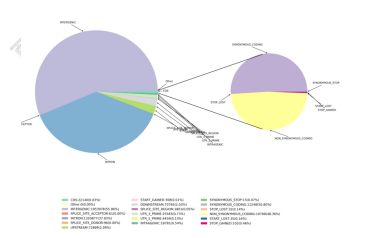
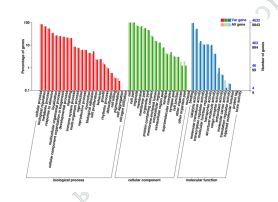
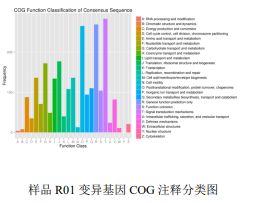
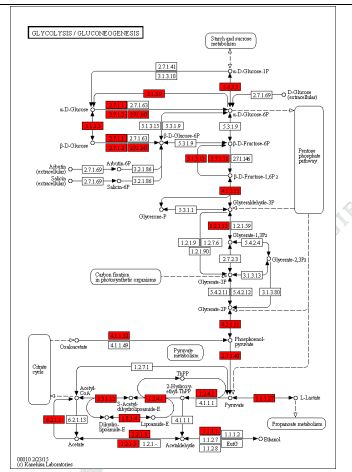
3) Funksjonell merknad av variasjoner
| 2019 | Naturkommunikasjon | Resekvensering av hele genomet avslører Brassica napus opprinnelse og genetiske loki involvert i forbedringen |
| 2020 | PNAS | Den evolusjonære opprinnelsen og domestiseringshistorien til gullfisk (Carassius auratus) |
| 2021 | Plantebioteknologisk tidsskrift | Referansegenomer til de to kultiverte juteartene |
| 2021 | Plantebioteknologisk tidsskrift | Genomiske signaturer av vegetabilsk og oljefrø allopolyploid Brassicajuncea og genetiske loki som kontrollerer akkumulering av glukosinolater |
| 2019 | Molekylær plante | Helgenom-resekvensering av en verdensomspennende samling av rapsfrø avslører genetisk grunnlag for deres økotype-divergens |
| 2022 | Hagebruksforskning | Genomomfattende assosiasjonsanalyse gir molekylær innsikt i den naturlige variasjonen av vannmelonfrøstørrelse |
| 2021 | Journal of Experimental Botany | Genomomfattende assosiasjonsstudie identifiserer varianter av GhSAD1 som gir kuldetoleranse i bomull |
| 2021 | Journal of Experimental Botany | Genomomfattende assosiasjonsstudie og transkriptomsammenligning avslører nye QTL og kandidatgener som kontrollerer kronbladstørrelsen i raps |