
Sekwencjonowanie amplikonu 16S/18S/ITS-NGS
Zalety serwisu
● Bez izolacji i szybka identyfikacja składu mikrobiologicznego w próbkach środowiskowych
● Wysoka rozdzielczość w przypadku niewielkiej ilości składników w próbkach środowiskowych
● Najnowsza analiza przepływu QIIME2 z różnorodnymi analizami pod względem bazy danych, adnotacji, OTU/ASV.
● Wysoka przepustowość, większa dokładność
● Ma zastosowanie w różnorodnych badaniach społeczności drobnoustrojów
● BMK posiada bogate doświadczenie z ponad 100 000 próbek rocznie, obejmujących glebę, wodę, gaz, osad, kał, jelita, skórę, bulion fermentacyjny, owady, rośliny itp.
● BMKCloud ułatwił interpretację danych, zawierający 45 spersonalizowanych narzędzi analitycznych
Specyfikacje usług
| SekwencjonowaniePlatforma | Biblioteka | Zalecana wydajność danych | Szacowany czas realizacji |
| Platforma Illumina NovaSeq | PE250 | Tagi 50 tys./100 tys./300 tys | 30 dni |
Analizy bioinformatyczne
● Kontrola jakości surowych danych
● Klastrowanie/odszumianie OTU (ASV)
● Adnotacja OTU
● Różnorodność alfa
● Różnorodność wersji beta
● Analiza międzygrupowa
● Analiza asocjacyjna względem czynników eksperymentalnych
● Przewidywanie genów funkcyjnych
Przykładowe wymagania i dostawa
Przykładowe wymagania:
DlaEkstrakty DNA:
| Typ próbki | Kwota | Stężenie | Czystość |
| Ekstrakty DNA | > 30 ng | > 1 ng/µl | OD260/280= 1,6-2,5 |
Dla próbek środowiskowych:
| Typ próbki | Zalecana procedura pobierania próbek |
| Gleba | Ilość próbki: ok.5 g;Pozostałą zwiędłą substancję należy usunąć z powierzchni;Zmiel duże kawałki i przepuść przez filtr 2 mm;Próbki w sterylnej probówce EP lub cyrotubie do rezerwacji. |
| Kał | Ilość próbki: ok.5 g;Zbieraj i rozdzielaj próbki do sterylnej probówki EP lub krioprobówki w celu rezerwacji. |
| Treść jelitowa | Próbki należy przetwarzać w warunkach aseptycznych.Przemyć pobraną tkankę PBS;Odwirować PBS i zebrać osad do probówek EP. |
| Osad | Ilość próbki: ok.5 g;Zbierz i podziel na porcje próbkę osadu do sterylnej probówki EP lub krioprobówki w celu rezerwacji |
| Zbiornik wodny | W przypadku próbki zawierającej ograniczoną ilość drobnoustrojów, takiej jak woda z kranu, woda ze studni itp., zebrać co najmniej 1 l wody i przepuścić przez filtr 0,22 μm, aby wzbogacić mikroorganizmy na membranie.Przechowywać membranę w sterylnej probówce. |
| Skóra | Ostrożnie zeskrob powierzchnię skóry sterylnym wacikiem lub ostrzem chirurgicznym i umieść go w sterylnej probówce. |
Zalecana dostawa próbek
Zamrozić próbki w ciekłym azocie na 3-4 godziny i przechowywać w ciekłym azocie lub w -80 stopniach z rezerwacją długoterminową.Wymagana jest wysyłka próbek z suchym lodem.
Przebieg prac serwisowych

Dostawa próbek

Budowa biblioteki

Sekwencjonowanie

Analiza danych

Usługi posprzedażowe
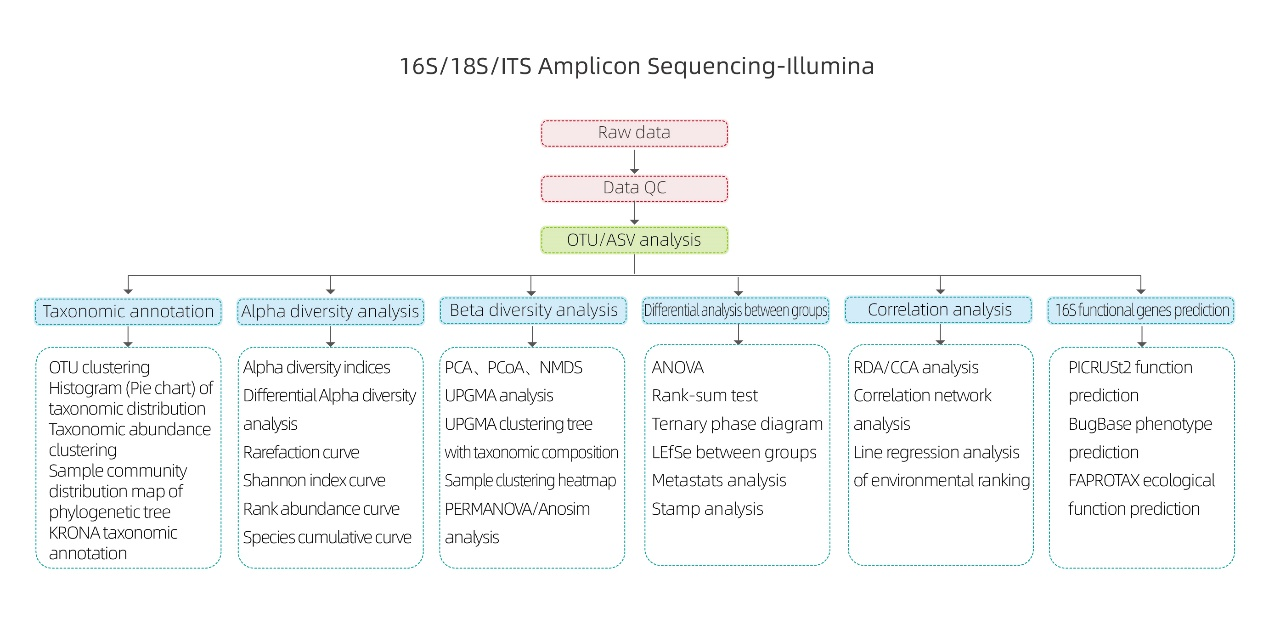
1.Rozmieszczenie gatunków
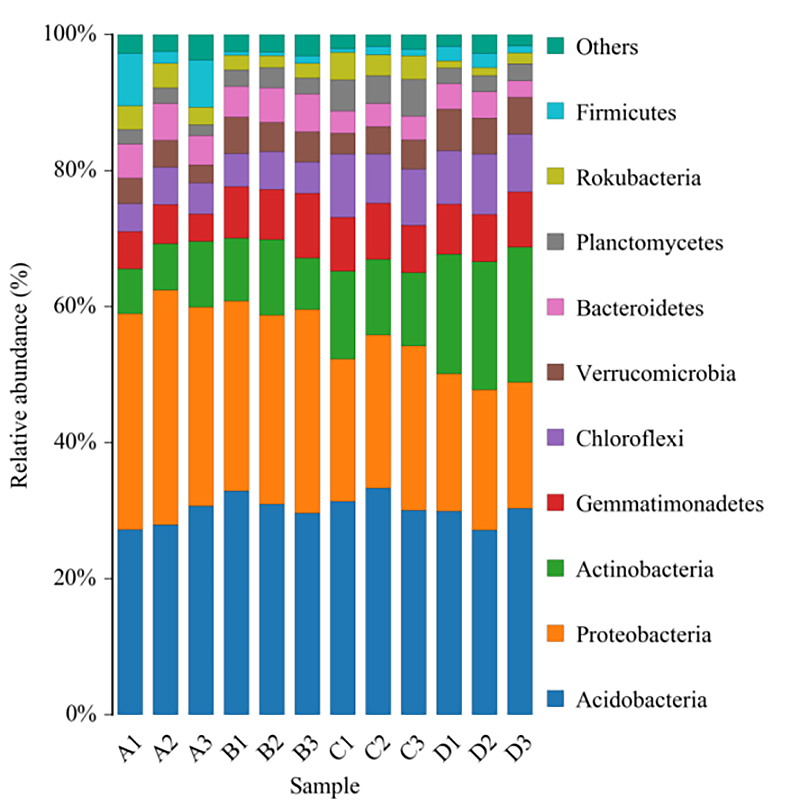
2. Mapa cieplna: grupowanie bogactwa gatunków
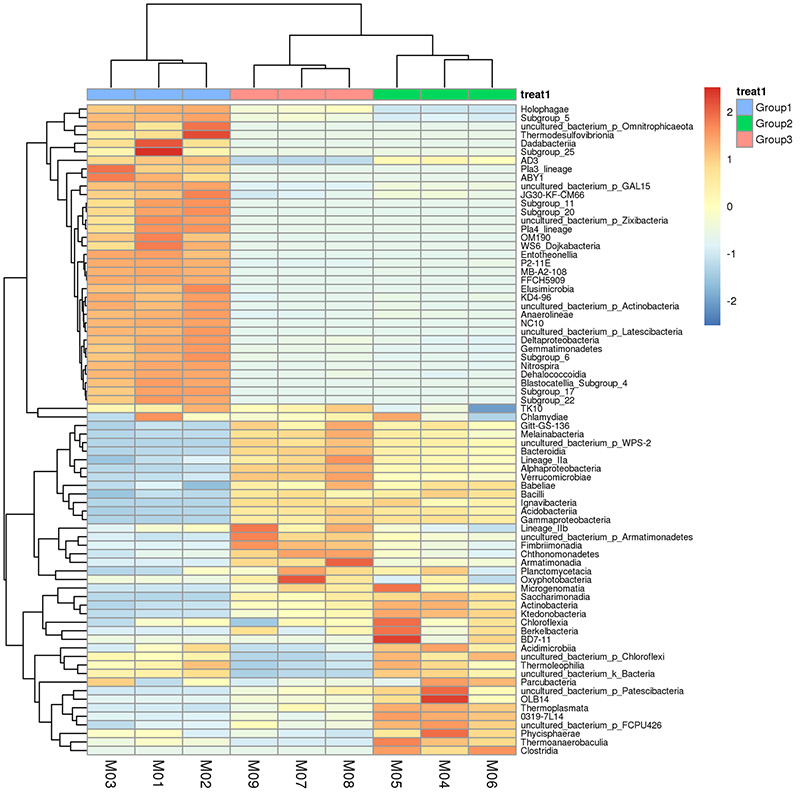
3.Rzadka krzywa frakcji
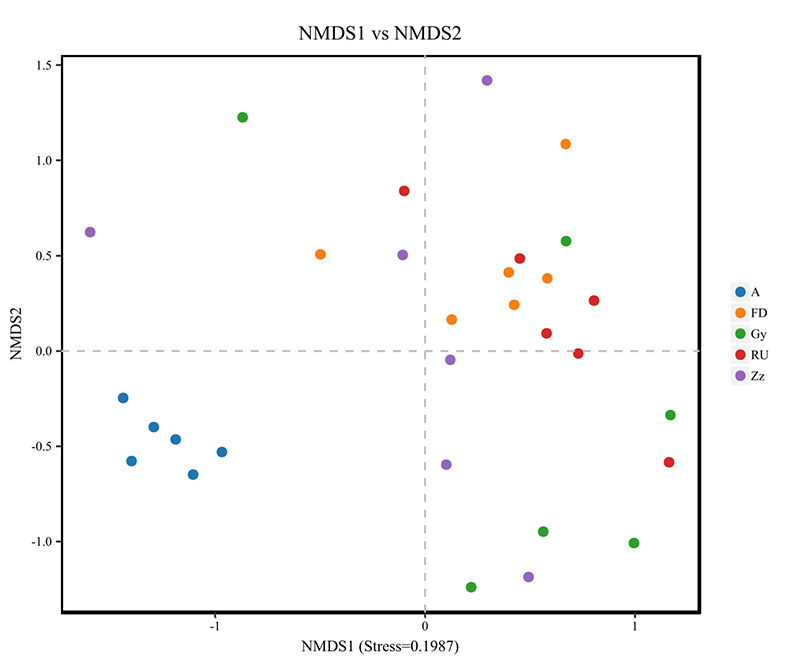
4.Analiza NMDS
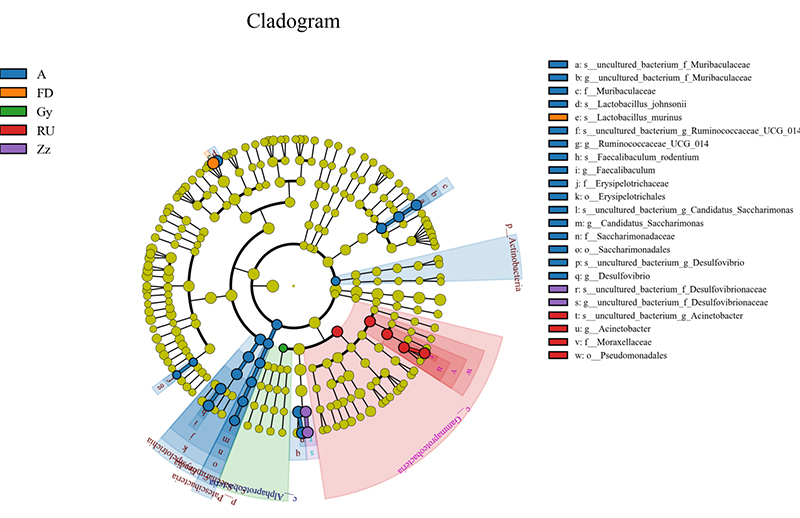
5.Analiza Lefsego
Sprawa BMK
Osoby otyłe z cukrzycą typu 2 i bez niej wykazują różną zdolność funkcjonalną i skład drobnoustrojów jelitowych
Opublikowany:Gospodarz komórkowy i mikrob, 2019
Strategia sekwencjonowania:
osoby szczupłe, niebędące cukrzykami (n=633);otyli, niebędący cukrzycą (n=494);otyłość, cukrzyca typu 2 (n=153);
Region docelowy: 16S rDNA V1-V2
Platforma: Illumina Miseq (sekwencjonowanie amplikonu w oparciu o NGS)
Podzbiór ekstraktów DNA poddano sekwencjonowaniu metagenomicznemu na Illumina Hiseq
Kluczowe wyniki
Udało się zróżnicować profile mikrobiologiczne tych chorób metabolicznych.
Porównując cechy drobnoustrojów wygenerowane przez sekwencjonowanie 16S, stwierdzono, że otyłość jest powiązana ze zmianami w składzie drobnoustrojów, cechami osobniczymi, szczególnie znacznym zmniejszeniem liczby Akkermansia, Faecalibacterium, Oscillibacter, Alistipes itp. Ponadto stwierdzono, że T2D wiąże się ze wzrostem liczby Escherichia/Shigella .
Odniesienie
Thingholm, LB i in.„Osoby otyłe z cukrzycą typu 2 i bez niej wykazują różną pojemność funkcjonalną i skład drobnoustrojów jelitowych”.Gospodarz komórkowy i mikrob26,2 (2019).











