Sekwencjonowanie DNA/RNA – Sekwenser Nanoporów
Szczegóły usługi Funkcje
| Platforma | Rozmiar biblioteki | Teoretyczna wydajność danych (na komórkę) | Dokładność pojedynczej podstawy | Aplikacje |
| Nanopor | 8 kb, 10 kb, 20 kb, ultradługie, cDNA-PCR | 70-90 Gb/komórkę | 85-92% | Wywołanie SV, De novo, Sekwencjonowanie pełnej długości, Iso-Seq, Adnotacja genu, Wykrywanie metylacji DNA |
Zalety serwisu
● Ponad 5 lat doświadczenia na platformie sekwencjonowania PacBio z tysiącami zamkniętych projektów z różnymi gatunkami.
● BMKGENE jest oficjalnym partnerem Oxford Nanopore, posiadającym podwójną platformę certyfikacji RNA/DNA.
● Istnieją główne modele sekwencerów z kompletnym wyposażeniem i wystarczającą przepustowością sekwencjonowania.
● W oparciu o platformę Nanopore opublikowano ponad 10 badań Denovo na zwierzętach i roślinach w czasopismach o międzynarodowej renomie.
Przykładowe wymagania
| Typ próbki | Kwota | Stężenie (Qubit ®) | Tom | Czystość | Inni |
| Genomowe DNA | Zależy od wymagań dotyczących danych | ≥20 ng/µl | ≥15µl | OD260/280=1,7-2,2; OD260/230≥1,5; Wyraźny pik przy 260 nm, brak zanieczyszczeń | Stężenie należy mierzyć za pomocą Qubitu i Kubitu/Nanoporu ≤ 2 |
| Całkowity RNA | ≥1,2 µg | ≥100 μg/μl | ≥15µl | OD260/280=1,7-2,5; OD260/230=0,5-2,5; brak zanieczyszczeń | Wartość RIN ≥7,5 |
Przepływ pracy w serwisie
przygotowanie próbki

Budowa biblioteki

Sekwencjonowanie

Analiza danych

Dostawa projektu
Ocena jakości danych próbki DNA
Tabela 1. Statystyki dotyczące czystych danych.
| BMKID | surowyNrSekw | surowaSumaBase | cleanSeqNum | czystaSumaBase | czysteN50Len | czysteN90Len | czystyŚredniLen | czystyMaxLen | czystaŚredniaJakość |
| DNA_BMK01 | 1 218 239 | 26.37 | 1 121 736 | 25,90 | 28014 | 15764 | 23090 | 143181 | 9 |
Ocena jakości danych próbki RNA
Tabela 1. Statystyki dotyczące czystych danych.
| Nazwa pliku | Identyfikator klienta | PrzeczytajNum | BaseNum | N50 | Średnia długość | Maksymalna długość | Średni wynik Q |
| RNA_BMK001 | C2 | 8947708 | 4 047 230 083 | 398 | 452 | 129227 | Pytanie 12 |
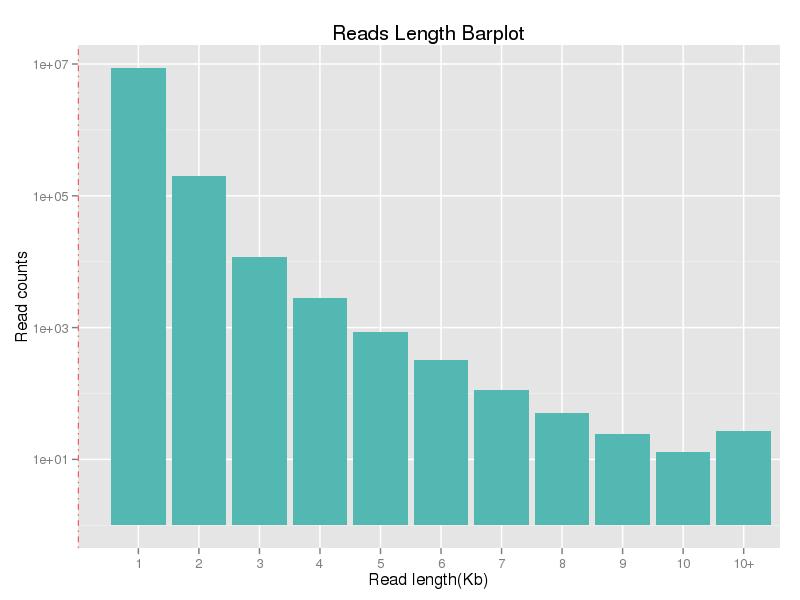
Rysunek 1. Rozkład długości odczytu
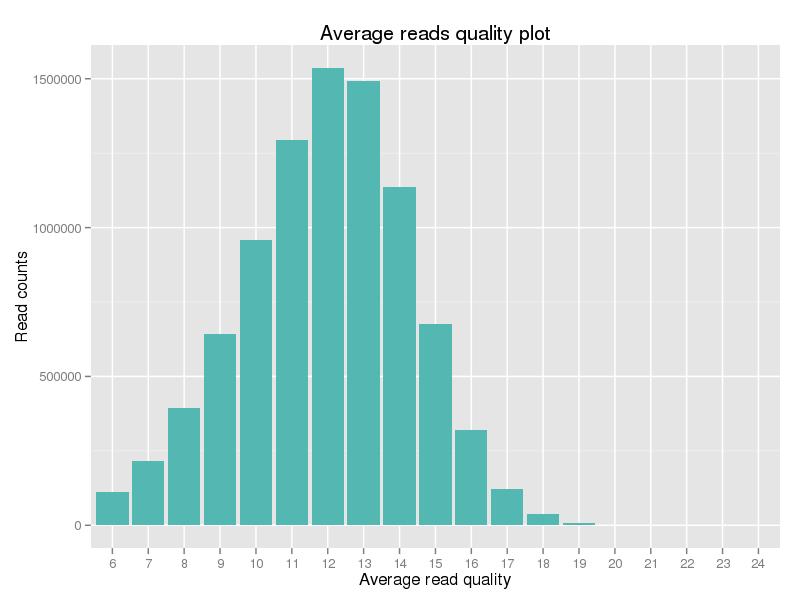
Rysunek 2. Rozkład Wyniku Jakości czystych danych
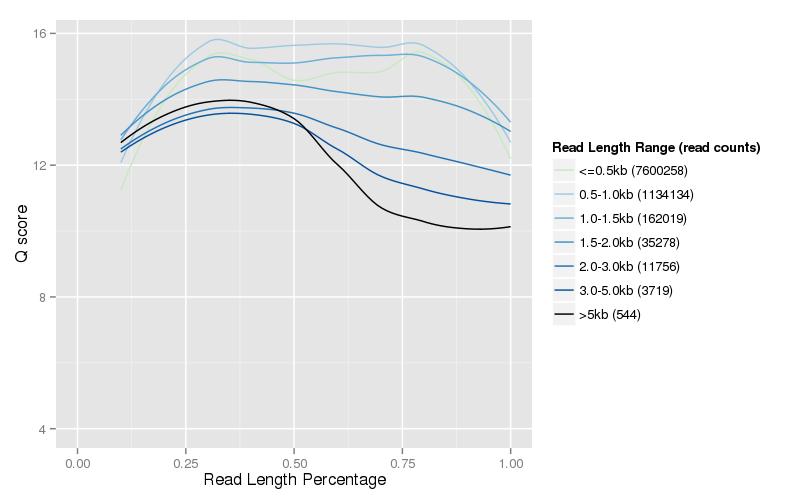
Rysunek 3. Rozkład długości i wyniku jakości czystych danych