Sekwencjonowanie całego genomu roślin/zwierząt
1. Zalety usług
Bogate doświadczenie w sekwencjonowaniu genomów ponad 1000 gatunków.
Ponad 800 opublikowanych przypadków o skumulowanym współczynniku wpływu ponad 4000.
Kompleksowa analiza bioinformatyczna dotycząca wywoływania wariacji i analizy funkcji.
2. Specyfikacje Usług
| Platforma | Biblioteka | Zalecana głębokość sekw | |
| Ilumina | PE 150 | Dla SNP, InDel dzwoni ≥ 10x W przypadku SV, CNY dzwonią ≥ 30x
| |
| Nanopor
| 8 kilobajtów | W przypadku SV, CNY dzwonienie ≥ 20x | |
| Pacbio | CCS | 15 kb | Dla SNP, InDel, SV, CNY dzwonienie ≥ 10x |
3. Przykładowe wymagania
| Platforma | Stężenie (ng/μL)
| Kwota (ng)
| Czystość
| Żel agarozowy
| ||
| OD260/280 | OD260/230 | 1. Wyczyść pasmo główne bez lub z ograniczeniamidegradacja obserwowana na żelu. 2. Brak lub ograniczone zanieczyszczenie RNA lub białkiem
| ||||
| Ilumina | ≥1 | ≥30
| - | - | ||
| Nanopor
| ≥30 | Zależy od wydajności danych 10 µg/komórkę | 1,7-2,2 | ≥1,5 | ||
| Pacbio | CCS | ≥50 | 1,7-2,2 | 1,8-2,5 | ||
4. Analiza bioinformacyjna
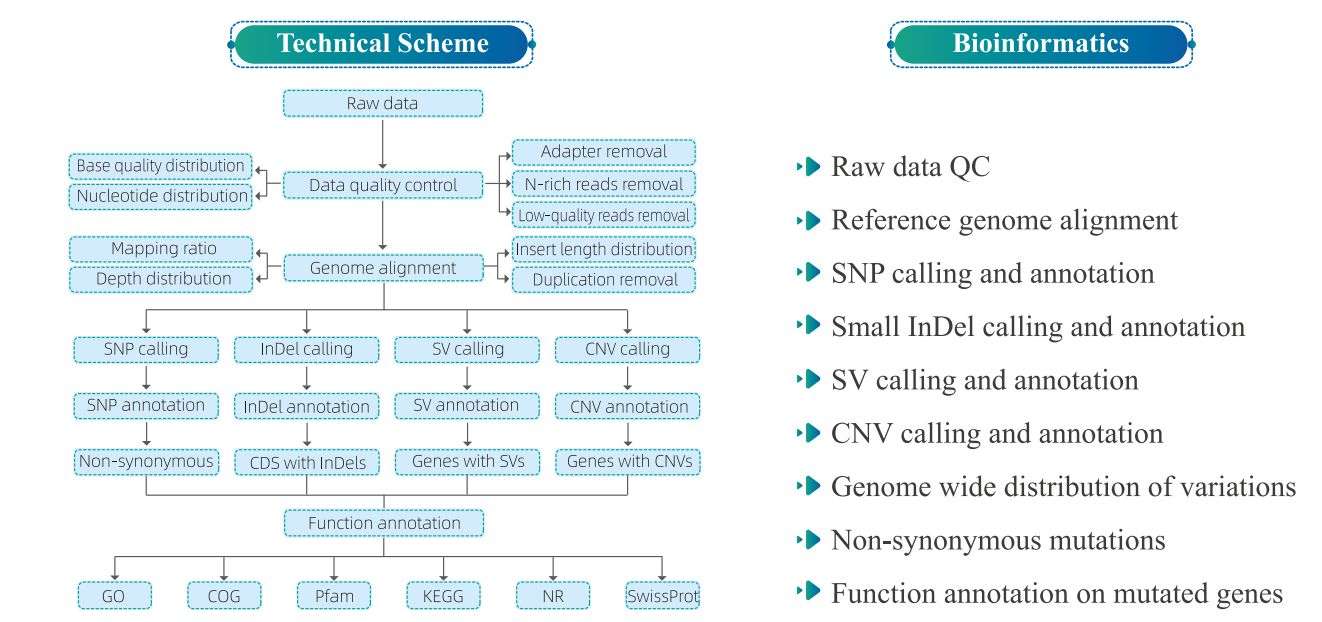
5. Przebieg prac serwisowych

Dostawa próbek

Ekstrakcja DNA

Budowa biblioteki

Sekwencjonowanie

Analiza danych

Dostarczanie danych
1) Statystyka mapowania genomu
Tabela 1 Statystyka wyniku mapowania
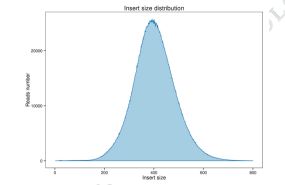
Rysunek 1 Rozkład wielkości wkładek i pokrycia odczytów.
2) Zmienne wykrywanie
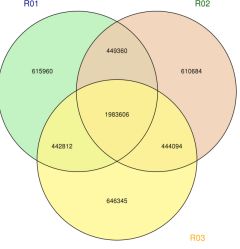
Rysunek 2 Statystyka i adnotacja SNP/INDEL/SV wśród próbek
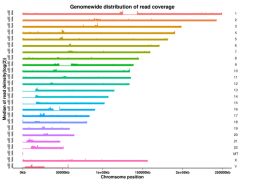
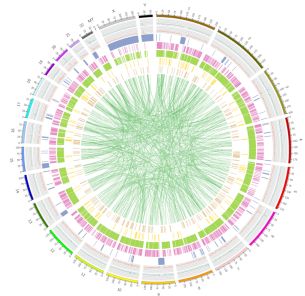
Rycina 3. Rozkład watów w całym genomie
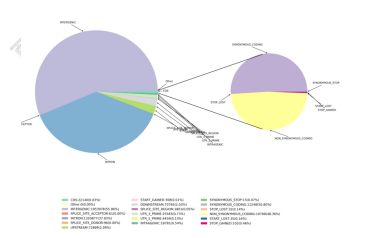
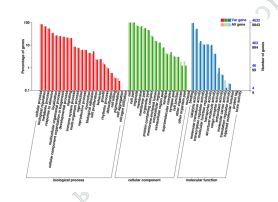
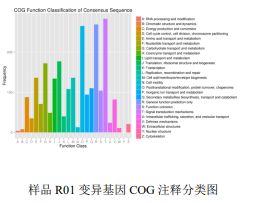
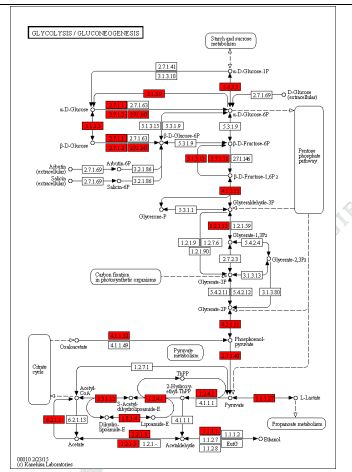
3) Adnotacja funkcjonalna wariantów
| 2019 | Komunikacja przyrodnicza | Ponowne sekwencjonowanie całego genomu ujawnia pochodzenie Brassica napus i loci genetyczne zaangażowane w jego poprawę |
| 2020 | PNAS | Ewolucyjne pochodzenie i historia udomowienia złotej rybki (Carassius auratus) |
| 2021 | Dziennik biotechnologii roślin | Genomy referencyjne dwóch uprawianych gatunków juty |
| 2021 | Dziennik biotechnologii roślin | Sygnatury genomowe allopoliploidów warzyw i nasion oleistych Brassicajuncea oraz loci genetyczne kontrolujące akumulację glukozynolanów |
| 2019 | Roślina molekularna | Ponowne sekwencjonowanie całego genomu ogólnoświatowej kolekcji nasion rzepaku ujawnia genetyczne podstawy rozbieżności ich ekotypów |
| 2022 | Badania ogrodnicze | Analiza asocjacji obejmująca cały genom dostarcza molekularnego wglądu w naturalną zmienność wielkości nasion arbuza |
| 2021 | Journal of Experimental Botaniki | Badanie asocjacyjne obejmujące cały genom identyfikuje warianty GhSAD1 nadające bawełnie tolerancję na zimno |
| 2021 | Journal of Experimental Botaniki | Badanie asocjacji całego genomu i porównanie transkryptomu ujawniają nowe geny QTL i geny kandydujące, które kontrolują wielkość płatków rzepaku |