Sekwencjonowanie małego RNA – Illumina
Cechy
● Wybór wielkości RNA przed przygotowaniem biblioteki
● Analiza bioinformatyczna skupiona na przewidywaniu miRNA i ich celach
Zalety serwisu
●Kompleksowa analiza bioinformatyczna:umożliwienie identyfikacji zarówno znanych, jak i nowych miRNA, identyfikacji celów miRNA i odpowiedniej adnotacji funkcjonalnej oraz wzbogacenia o wiele baz danych (KEGG, GO)
●Rygorystyczna kontrola jakości: wdrażamy podstawowe punkty kontroli na wszystkich etapach, od przygotowania próbek i bibliotek po sekwencjonowanie i bioinformatykę.To skrupulatne monitorowanie zapewnia niezmiennie wysoką jakość wyników.
●Wsparcie posprzedażowe: Nasze zaangażowanie wykracza poza ukończenie projektu i obejmuje 3-miesięczny okres obsługi posprzedażnej.W tym czasie oferujemy monitorowanie projektu, pomoc w rozwiązywaniu problemów oraz sesje pytań i odpowiedzi, aby odpowiedzieć na wszelkie pytania związane z wynikami.
●Rozległa wiedza specjalistyczna: mając na swoim koncie pomyślne zakończenie wielu projektów sRNA obejmujących ponad 100 gatunków w różnych dziedzinach badań, nasz zespół wnosi do każdego projektu bogate doświadczenie.
Przykładowe wymagania i dostawa
| Biblioteka | Platforma | Zalecane dane | Kontrola danych |
| Wybrany rozmiar | Ilumina SE50 | 10–20 mln odczytów | Q30≥85% |
Przykładowe wymagania:
Nukleotydy:
| Stężenie (ng/μl) | Ilość (µg) | Czystość | Uczciwość |
| ≥ 80 | ≥ 0,5 | OD260/280=1,7-2,5 OD260/230=0,5-2,5 Na żelu widoczne jest ograniczone lub żadne zanieczyszczenie białkiem lub DNA. | RIN≥6,5; 5,0 ≥ 28 S/18 S ≥ 1,0; ograniczone lub żadne wzniesienie linii bazowej |
● Rośliny:
Korzeń, łodyga lub płatek: 450 mg
Liść lub nasiona: 300 mg
Owoce: 1,2 g
● Zwierzę:
Serce lub jelita: 450 mg
Wnętrzności lub mózg: 240 mg
Mięśnie: 600 mg
Kości, włosy lub skóra: 1,5 g
● Stawonogi:
Owady: 9g
Skorupiaki: 450 mg
● Krew pełna: 2 rurki
● Komórki: 106 komórki
● Surowica i osocze:6 ml
Zalecana dostawa próbek
Pojemnik:
Probówka wirówkowa o pojemności 2 ml (nie zaleca się stosowania folii aluminiowej)
Przykładowe oznakowanie: Grupa+replika, np. A1, A2, A3;B1, B2, B3... ...
Wysyłka:
1.Suchy lód: Próbki należy zapakować do worków i zakopać w suchym lodzie.
2. Probówki RNAstable: Próbki RNA można suszyć w probówkach do stabilizacji RNA (np. RNAstable®) i przesyłać w temperaturze pokojowej.
Przebieg prac serwisowych

Projekt eksperymentu

Dostawa próbek

Ekstrakcja RNA

Budowa biblioteki

Sekwencjonowanie

Analiza danych

Usługi posprzedażowe
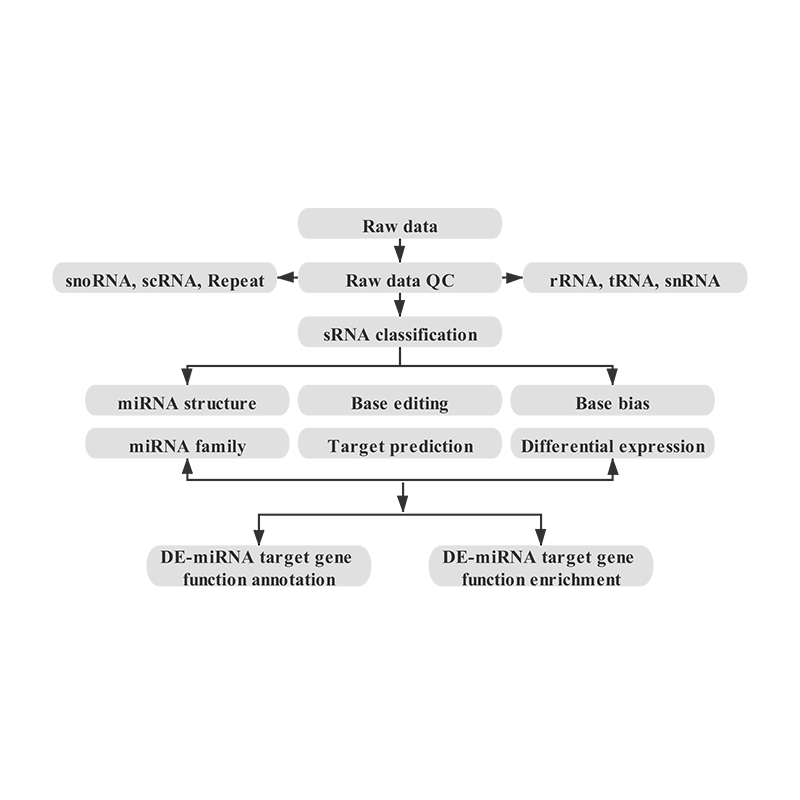
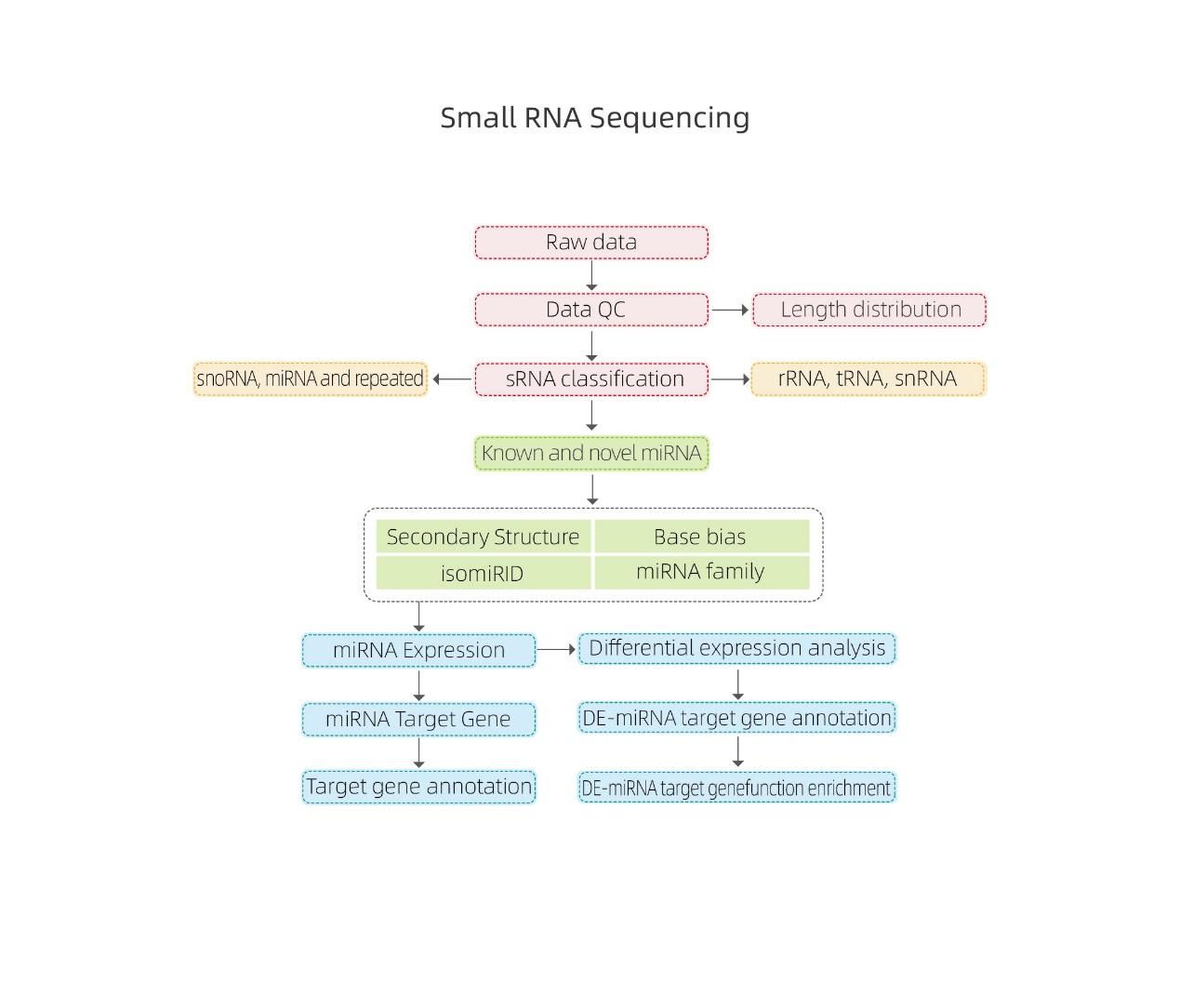
Bioinformatyka
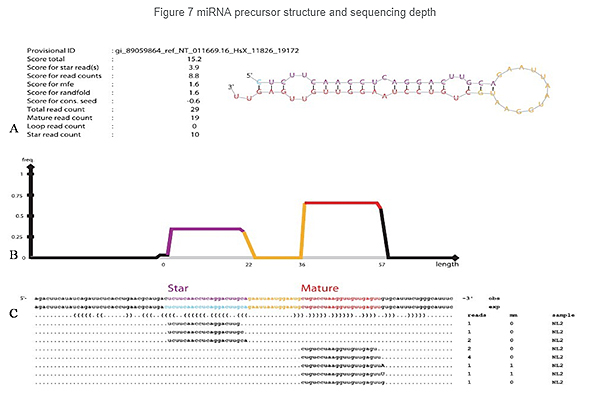
Identyfikacja miRNA: struktura i głębokość
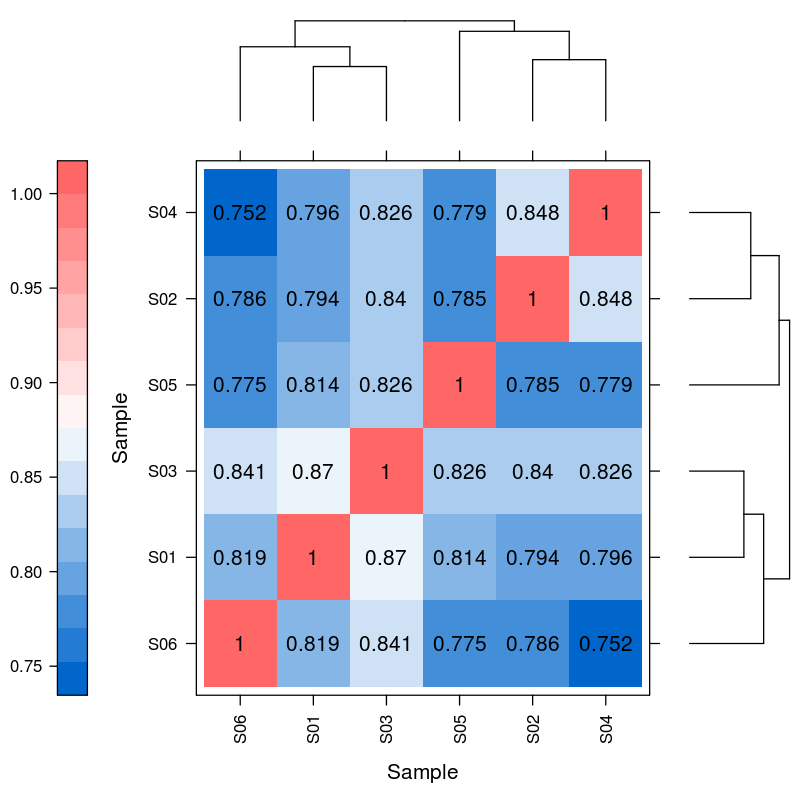
Różnicowa ekspresja miRNA – grupowanie hierarchiczne
Adnotacja funkcjonalna celu miRNA o różnej ekspresji
Zapoznaj się z postępami badawczymi, jakie umożliwiły usługi sekwencjonowania sRNA firmy BMKGene, poprzez wyselekcjonowany zbiór publikacji.
Chen, H. i in.(2023) „Infekcje wirusowe hamują biosyntezę i fotosyntezę saponin u Panax notoginseng”, Plant Physiology and Biochemistry, 203, s. 1.108038. doi: 10.1016/J.PLAPHY.2023.108038.
Li, H. i in.(2023) „Roślinne białko FREE1 zawierające domenę FYVE wiąże się ze składnikami mikroprocesora w celu tłumienia biogenezy miRNA”, donosi EMBO, 24(1).doi: 10.15252/EMBR.202255037/SUPPL_FILE/EMBR202255037-SUP-0004-SDATAFIG4.TIF.
Yu, J. i in.(2023) „MikroRNA Ame-Bantam-3p kontroluje rozwój larw poczwarek poprzez celowanie w gen 8 domen podobnych do wielu naskórkowych czynników wzrostu (megf8) u pszczół miodnych, Apis mellifera”, International Journal of Molecular Sciences, 24(6), s. .5726. doi: 10.3390/IJMS24065726/S1.
Zhang, M. i in.(2018) „Zintegrowana analiza miRNA i genów związanych z jakością mięsa ujawnia, że Gga-MiR-140-5p wpływa na śródmięśniowe odkładanie tłuszczu u kurczaków”, Cellular Physiology and Biochemistry, 46(6), s. 2421–2433.doi: 10.1159/000489649.