
Sequenciamento de Amplicon 16S/18S/ITS-NGS
Vantagens do serviço
● Identificação rápida e sem isolamento da composição microbiana em amostras ambientais
● Alta resolução em componentes pouco abundantes em amostras ambientais
● O mais recente fluxo de análise QIIME2 com diversas análises em termos de banco de dados, anotação, OTU/ASV.
● Alto rendimento e maior precisão
● Aplicável a diversos estudos de comunidades microbianas
● A BMK possui ampla experiência com mais de 100.000 amostras/ano, abrangendo solo, água, gás, lodo, fezes, intestinos, pele, caldo de fermentação, insetos, plantas, etc.
● BMKCloud facilitou a interpretação de dados contendo 45 ferramentas de análise personalizadas
Especificações de serviço
| SequenciamentoPlataforma | Biblioteca | Rendimento de dados recomendado | Tempo estimado de resposta |
| Plataforma Illumina NovaSeq | PE250 | Etiquetas 50K/100K/300K | 30 dias |
Análises de bioinformática
● Controle de qualidade de dados brutos
● Clusterização/eliminação de ruído de OTU (ASV)
● Anotação OTU
● Diversidade alfa
● Diversidade beta
● Análise intergrupo
● Análise de associação contra fatores experimentais
● Predição genética de função
Requisitos de amostra e entrega
Requisitos de amostra:
ParaExtratos de DNA:
| Tipo de amostra | Quantia | Concentração | Pureza |
| Extratos de DNA | > 30 ng | > 1 ng/μl | DO260/280 = 1,6-2,5 |
Para amostras ambientais:
| Tipo de amostra | Procedimento de amostragem recomendado |
| Solo | Quantidade de amostragem: aprox.5g;A substância murcha restante precisa ser removida da superfície;Triture pedaços grandes e passe por filtro de 2 mm;Alíquotas de amostras em tubo EP estéril ou tubo ciro para reserva. |
| Fezes | Quantidade de amostragem: aprox.5g;Coletar e alíquotar amostras em tubo EP estéril ou criotubo para reserva. |
| Conteúdo intestinal | As amostras precisam ser processadas sob condições assépticas.Lave o tecido coletado com PBS;Centrifugue o PBS e colete o precipitante em tubos EP. |
| Lodo | Quantidade de amostragem: aprox.5g;Coletar e alíquotar amostra de lodo em tubo EP estéril ou criotubo para reserva |
| Corpo d'água | Para amostras com quantidade limitada de micróbios, como água de torneira, água de poço, etc., colete pelo menos 1 L de água e passe por um filtro de 0,22 μm para enriquecer micróbios na membrana.Armazene a membrana em tubo estéril. |
| Pele | Raspe cuidadosamente a superfície da pele com um cotonete estéril ou lâmina cirúrgica e coloque-o em um tubo estéril. |
Entrega de amostra recomendada
Congele as amostras em nitrogênio líquido por 3-4 horas e armazene em nitrogênio líquido ou -80 graus para reserva de longo prazo.É necessário enviar amostras com gelo seco.
Fluxo de trabalho de serviço

Entrega de amostra

Construção de biblioteca

Sequenciamento

Análise de dados

Serviços pós-venda
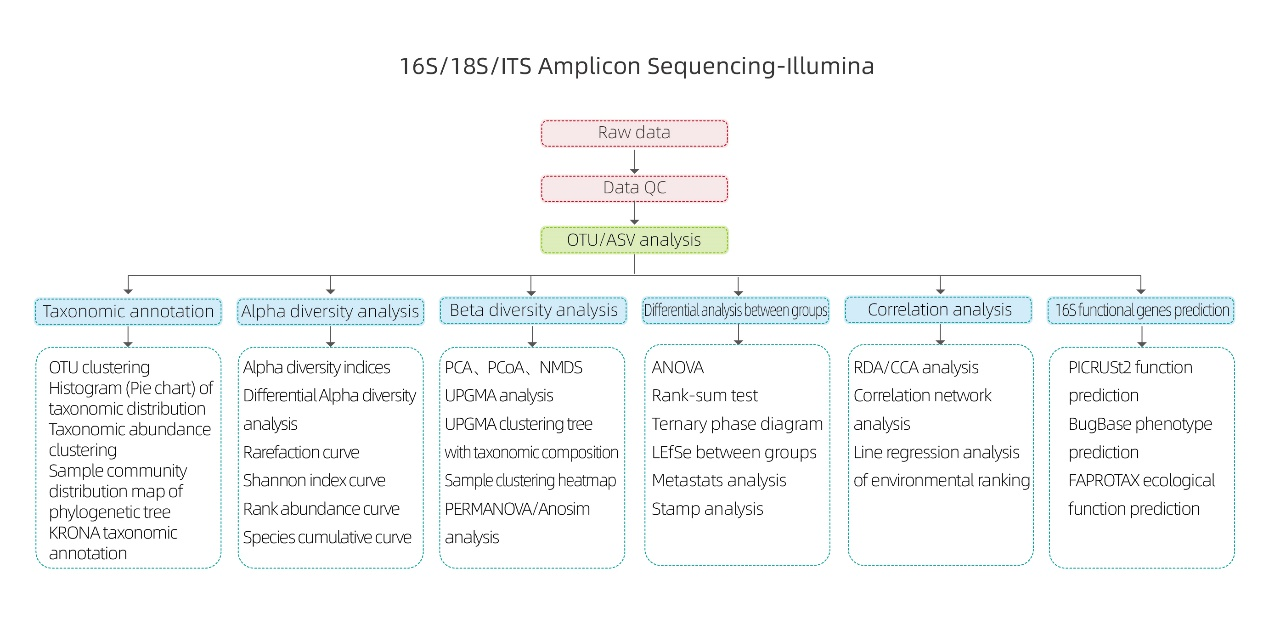
1.Distribuição de espécies
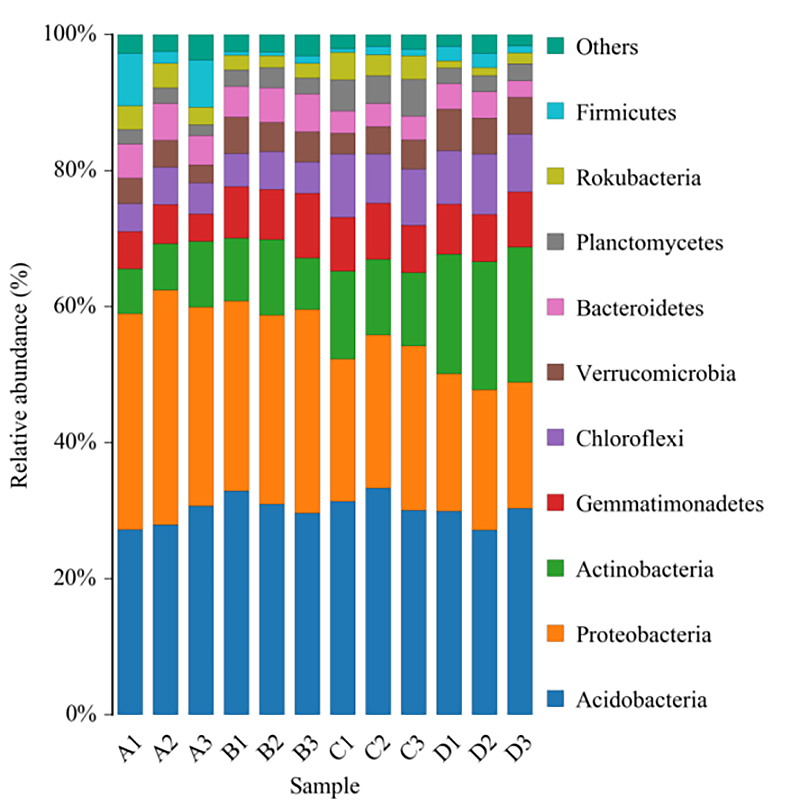
2.Mapa de calor: agrupamento de riqueza de espécies
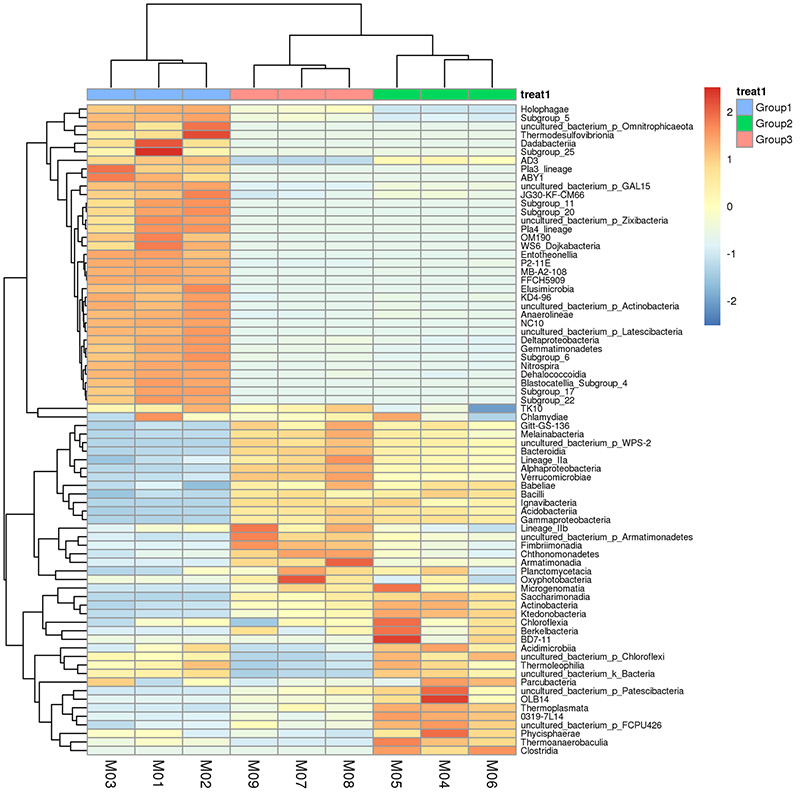
3. Curva de facção rara
4.Análise NMDS
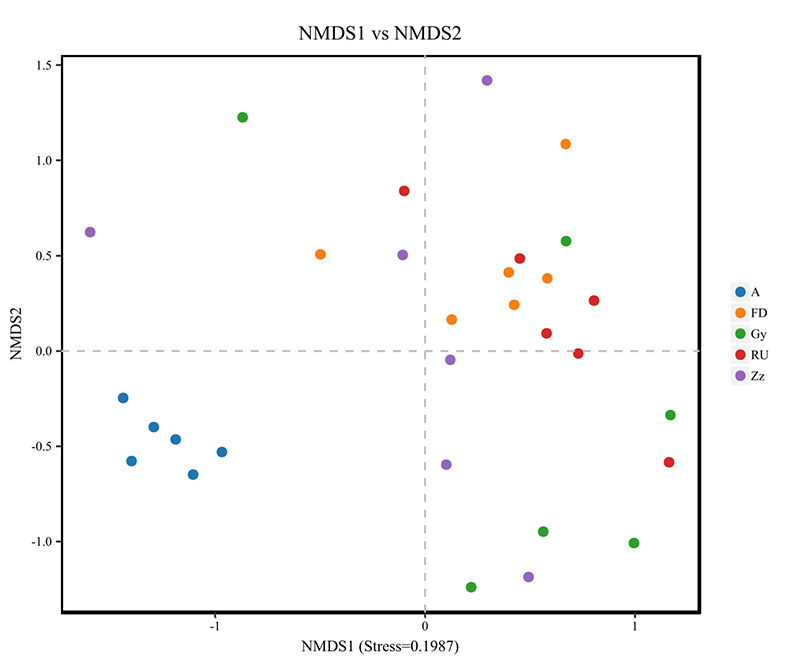
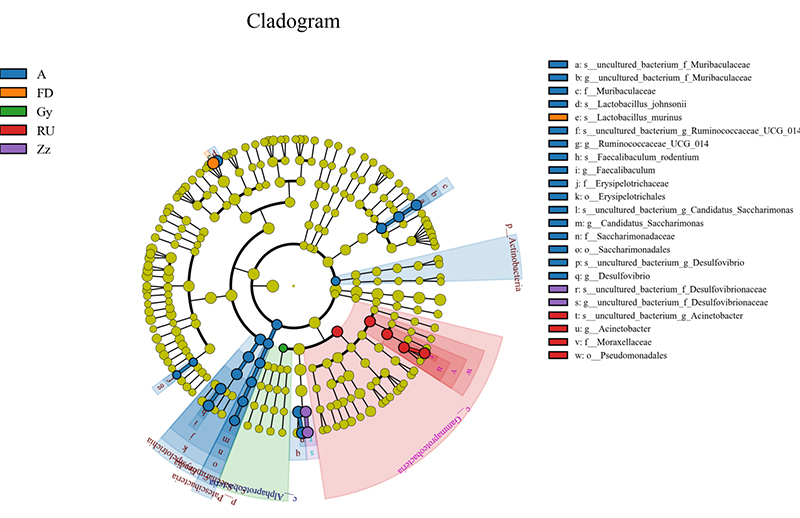
5. Análise Lefse
Caso BMK
Indivíduos obesos com e sem diabetes tipo 2 apresentam diferentes capacidades funcionais e composições microbianas intestinais
Publicados:Hospedeiro celular e micróbio, 2019
Estratégia de sequenciamento:
Magro não diabético (n=633);Obesos não diabéticos (n=494);Diabetes tipo 2 obeso (n=153);
Região alvo: 16S rDNA V1-V2
Plataforma: Illumina Miseq (sequenciamento de amplicon baseado em NGS)
Subconjunto de extratos de DNA foram submetidos ao sequenciamento metagenômico no Illumina Hiseq
Principais resultados
Os perfis microbianos destas doenças metabólicas foram diferenciados com sucesso.
Ao comparar as características microbianas geradas pelo sequenciamento 16S, descobriu-se que a obesidade estava associada a mudanças na composição microbiana, características individuais, especialmente diminuição significativa em Akkermansia, Faecalibacterium, Oscillibacter, Alistipes, etc. .
Referência
Thingholm, LB, et al.“Indivíduos obesos com e sem diabetes tipo 2 apresentam diferentes capacidades funcionais e composições microbianas intestinais.”Hospedeiro celular e micróbio26.2(2019).











