TRANSCRIPTÔMICA
natureza
COMUNICAÇÕES
A caracterização completa do transcrito da mutação SF3B1 na leucemia linfocítica crônica revela regulação negativa de íntrons retidos
Transcrições completas |Sequenciamento de nanoporos |Análise alternativa de isoformas
Fundo
SFoi amplamente relatado que mutações omáticas no fator de splicing SF3B1 estão associadas a vários tipos de câncer, incluindo leucemia linfocítica crônica (LLC), melanoma uveal, câncer de mama, etc. Além disso, estudos transcriptômicos de leitura curta revelaram padrões de splicing aberrantes induzidos por mutações no SF3B1.No entanto, os estudos sobre esses padrões de splicing alternativos têm sido limitados há muito tempo ao nível do evento e à falta de conhecimento no nível da isoforma devido à limitação de transcrições montadas de leitura curta.Aqui, a plataforma de sequenciamento de nanoporos foi introduzida para gerar transcrições completas, o que possibilitou a investigação das isoformas AS.
Design experimental
Experimentos
Agrupamento:1. CLL-SF3B1(WT) 2. CLL-SF3B1(mutação K700E);3. Células B normais
Estratégia de sequenciamento:Sequenciamento da biblioteca MinION 2D, sequenciamento da biblioteca PromethION 1D;dados de leitura curta das mesmas amostras
Plataforma de sequenciamento:ONT MinION;ONT Promethion;
Análise Bioinformática
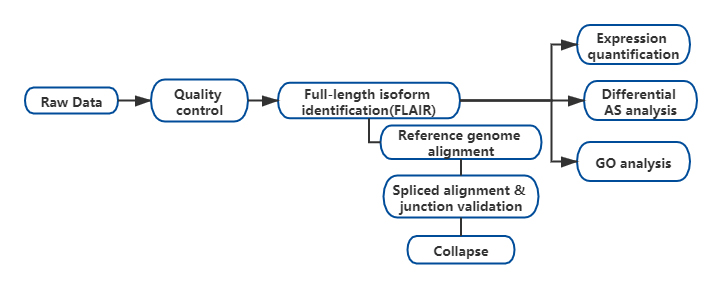
Resultados
Aum total de 257 milhões de leituras foram geradas a partir de 6 amostras de LLC e 3 células B.Em média, 30,5% dessas leituras foram identificadas como transcrições completas.
FA análise de isoformas alternativas de RNA de comprimento total (FLAIR) foi desenvolvida para gerar um conjunto de isoformas de alta confiança.FLAIR pode ser resumido como:
Nalinhamento de leituras de anoporo: identificar a estrutura geral do transcrito com base no genoma de referência;
Scorreção de junção de emenda: erros de sequência corretos (vermelho) com local de emenda de íntrons anotados, íntrons de dados de leitura curta ou ambos;
Colapso: resumir isoformas representativas com base em cadeias de junção de emenda (conjunto de primeira passagem).Selecione isofrom de alta confiança com base no número de leituras de suporte (Limite: 3).
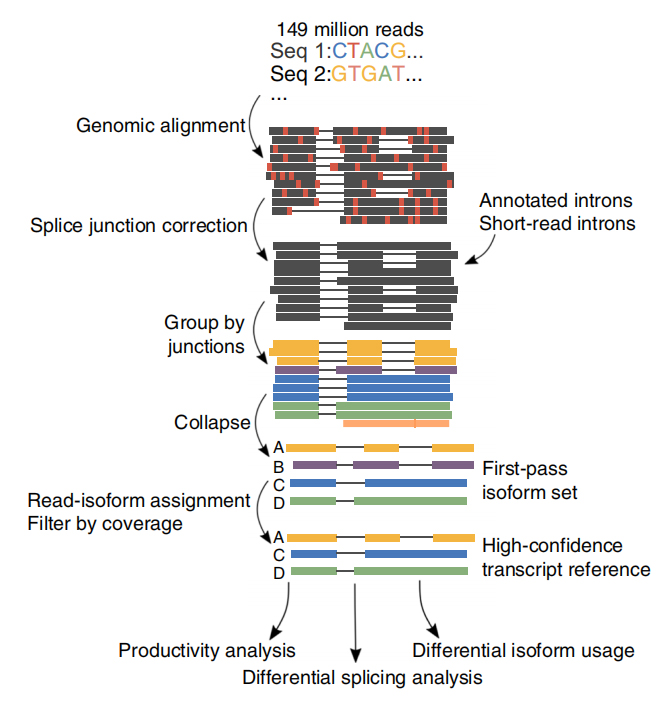
Figura 1. Análise FLAIR para identificar isoformas completas associadas à mutação SF3B1 na LLC
FLAIR identificou 326.699 isoformas emendadas de alta confiança, 90% das quais são isoformas novas.Descobriu-se que a maioria dessas isoformas não anotadas eram novas combinações de junções de splice conhecidas (142.971), enquanto as demais novas isoformas continham íntron retido (21.700) ou novo éxon (3594).
Lsequências de leitura ong permitem a identificação de locais de splice alterados por SF3B1-K700E mutantes no nível da isoforma.Descobriu-se que 35 3'SSs alternativos e 10 5'SSs alternativos apresentavam splicing significativamente diferencial entre SF3B1-K700E e SF3B1-WT.33 das 35 alterações foram descobertas recentemente por sequências de leitura longa.Nos dados do Nanopore, a distribuição da distância entre 3'SSs alterados por SF3B1-K700E e picos de locais canônicos é de cerca de -20 pb, o que difere significativamente de uma distribuição de controle, semelhante ao que foi relatado em sequências de leitura curta de CLL.Foram analisadas isoformas do gene ERGIC3, onde uma nova isoforma contendo o sítio de splice proximal foi encontrada mais abundante em SF3B1-K700E.Tanto o 3'SS proximal quanto o distal foram associados a padrões distintos de AS, gerando múltiplas isoformas.
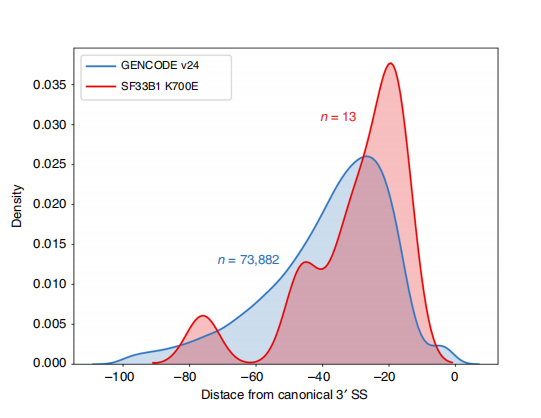
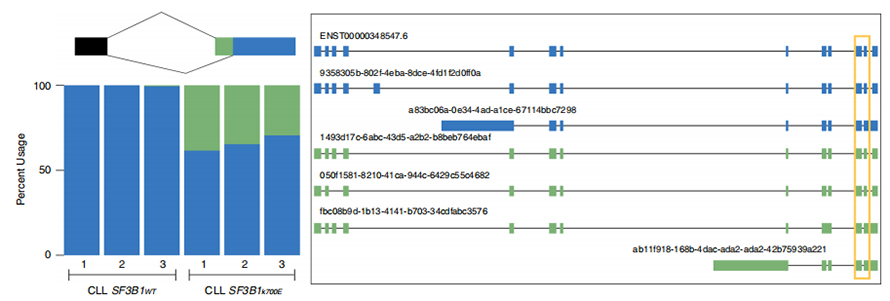
Figura 2. Padrões alternativos de splicing 3′ identificados com dados de sequenciamento de nanoporos
A análise de uso de eventos de IR tem sido limitada há muito tempo em análises baseadas em leituras curtas devido à confiança na identificação e quantificação de IR.A expressão das isoformas de IR em SF3B1-K700E e SF3B1-WT foi quantificada com base em sequências de nanoporos, revelando uma regulação negativa global das isoformas de IR em SF3B1-K700E .
Figura 4. Intensidade agrícola e conectividade de rede em três sistemas agrícolas (A e B);Análise florestal aleatória (C) e relação entre intensidade agrícola e colonização de FMAs (D)
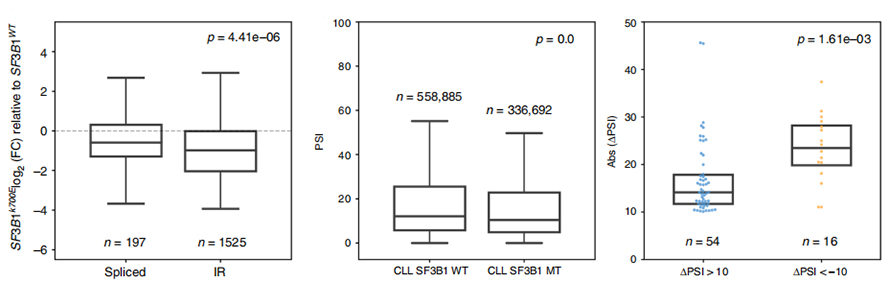
Figura 3. Os eventos de retenção de íntrons são mais fortemente regulados negativamente na LLC SF3B1-K700E
Tecnologia
Sequenciamento de leitura longa Nanopore
NO sequenciamento de anopore é uma tecnologia de sequenciamento de sinal elétrico em tempo real de molécula única.
DDNA ou RNA de fita dupla se ligará à proteína nanoporosa incorporada no biofilme e se desenrolará sob a liderança da proteína motora.
DAs cadeias de NA/RNA passam através da proteína do canal nanoporo a uma certa taxa sob a ação da diferença de voltagem.
Mas oléculas geram diferentes sinais elétricos de acordo com a estrutura química.
Ra detecção de sequências em tempo real é obtida por chamada de base.
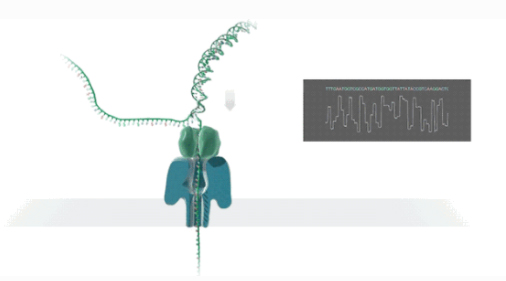
Desempenho do sequenciamento completo do transcriptoma
√ Saturação de dados
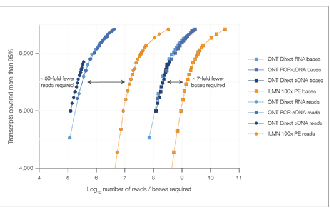
São necessárias 7 vezes menos leituras para atingir uma saturação de dados comparável.
√ Identificação da estrutura da transcrição
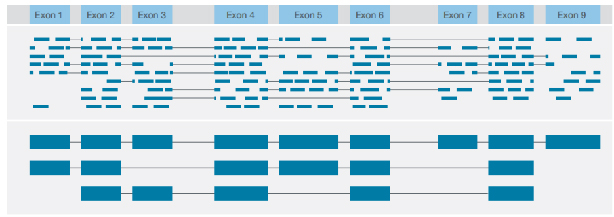
Identificação de diversas variantes estruturais com leitura completa e consensual de cada transcrição
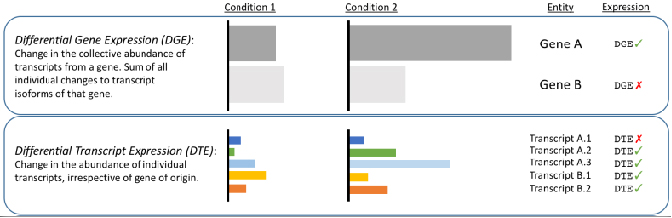
√ Análise diferencial em nível de transcrição - Revele alterações ocultas por leituras curtas
Referência
Tang AD, Soulette CM, Baren MJV, et al.A caracterização completa do transcrito da mutação SF3B1 na leucemia linfocítica crônica revela regulação negativa de íntrons retidos [J].Comunicações da Natureza.
Tecnologia e destaques visa compartilhar as mais recentes aplicações bem-sucedidas de diferentes tecnologias de sequenciamento de alto rendimento em diversas áreas de pesquisa, bem como ideias brilhantes em design experimental e mineração de dados.
Horário da postagem: 08 de janeiro de 2022

