ТРАНСКРИПТОМІКА
природи
ЗВ'ЯЗОК
Характеристика повнорозмірного транскрипту мутації SF3B1 при хронічному лімфолейкозі виявляє зниження регуляції утриманих інтронів
Повноформатні стенограми|Секвенування нанопор|Аналіз альтернативних ізоформ
Фон
SШироко повідомлялося, що оматичні мутації у факторі сплайсингу SF3B1 асоціюються з різними видами раку, включаючи хронічний лімфолейкоз (ХЛЛ), увеальну меланому, рак молочної залози тощо. Крім того, дослідження транскриптомії короткого читання виявили аномальні моделі сплайсингу, індуковані мутаціями SF3B1.Однак дослідження цих альтернативних моделей сплайсингу довгий час обмежувалися рівнем подій і відсутністю знань на рівні ізоформ через обмеження зібраних стенограм короткого читання.Тут була представлена платформа секвенування нанопор для генерації повнорозмірних транскриптів, що дозволило досліджувати ізоформи AS.
Експериментальний дизайн
Експерименти
Групування:1. CLL-SF3B1(WT) 2. CLL-SF3B1(мутація K700E);3. Нормальні В-клітини
Стратегія послідовності:Секвенування бібліотеки MinION 2D, секвенування бібліотеки PromethION 1D;дані короткого зчитування з тих самих зразків
Платформа секвенування:ОНТ Мініон;ОНТ ПрометІОН;
Біоінформаційний аналіз
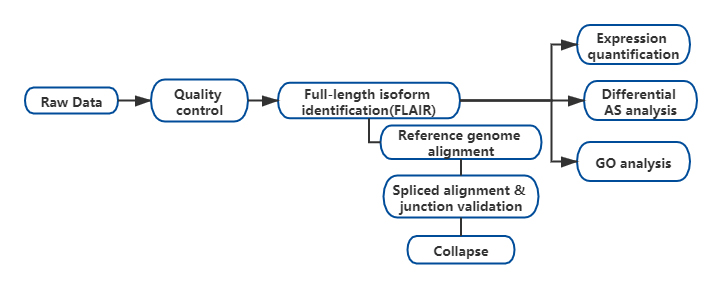
Результати
Азагалом було згенеровано 257 мільйонів зчитувань із 6 зразків CLL та 3 B-клітин.У середньому 30,5% цих читань були ідентифіковані як повноформатні стенограми.
FАльтернативний аналіз ізоформ РНК у довжині (FLAIR) був розроблений для створення набору високонадійних ізоформ.FLAIR можна коротко описати так:
Nanopore read alignment: визначити загальну структуру транскрипту на основі еталонного геному;
Sкорекція сплайсингу: виправте помилки послідовності (червоний) із сайтом сплайсингу з анотованих інтронів, інтронів з короткозчитаних даних або обох;
Collapse: узагальнення репрезентативних ізоформ на основі ланцюгів сплайсингу (набір першого проходу).Виберіть ізофром із високою достовірністю на основі кількості підтримуваних читань (поріг: 3).
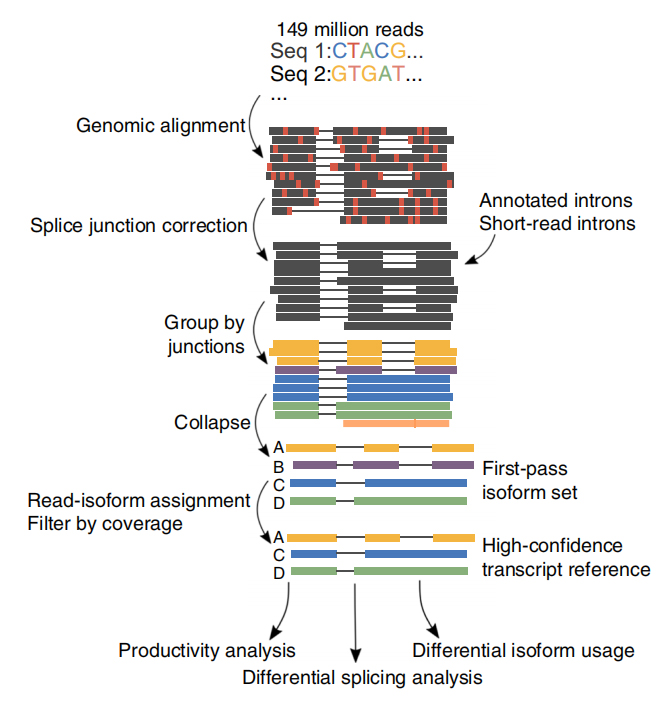
Рисунок 1. Аналіз FLAIR для ідентифікації ізоформ повної довжини, пов’язаних із мутацією SF3B1 у ХЛЛ
FLAIR Ідентифікував 326 699 високонадійних сплайсованих ізоформ, 90% з яких є новими ізоформами.Було виявлено, що більшість із цих неанотованих ізоформ є новими комбінаціями відомих з’єднань сплайсингу (142 971), тоді як решта нових ізоформ містили або збережений інтрон (21 700), або новий екзон (3594).
LПослідовності, які постійно читаються, дозволяють ідентифікувати мутантні SF3B1-K700E -змінені сайти сплайсингу на рівні ізоформи.Було виявлено, що 35 альтернативних 3'SS і 10 альтернативних 5'SS мають значну різницю між SF3B1-K700E і SF3B1-WT.33 з 35 змін були нещодавно виявлені послідовностями довгострокового зчитування.У даних Nanopore розподіл відстані між SF3B1-K700E-зміненими 3'SSs до піків канонічних сайтів становить близько -20 bp, що суттєво відрізняється від контрольного розподілу, подібно до того, про що повідомлялося в послідовностях короткого зчитування CLL.Було проаналізовано ізоформи гена ERGIC3, де виявлено, що нова ізоформа, що містить проксимальний сайт сплайсингу, більш поширена в SF3B1-K700E.Як проксимальний, так і дистальний 3'SS були пов'язані з чіткими моделями AS, що генерують кілька ізоформ.
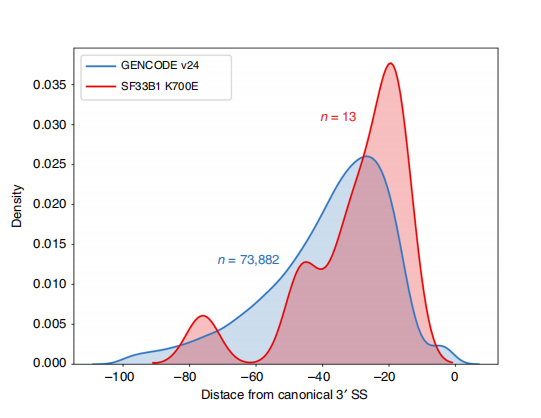
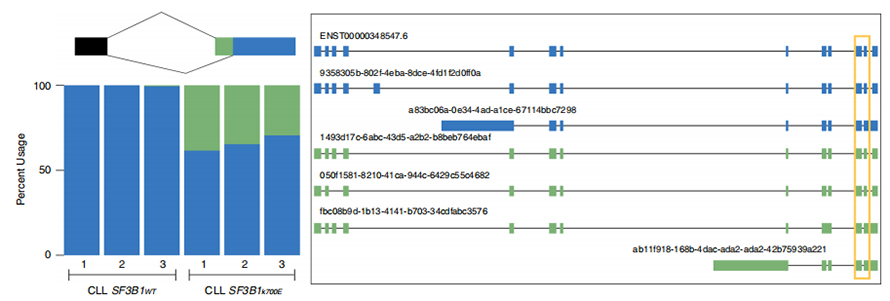
Малюнок 2. Альтернативні моделі сплайсингу 3′, визначені за допомогою даних секвенування нанопор
Аналіз використання подій ІЧ довгий час обмежувався аналізом на основі короткого читання через впевненість у ідентифікації та кількісному визначенні ІЧ.Експресію IR-ізоформ у SF3B1-K700E та SF3B1-WT було визначено кількісно на основі послідовностей нанопор, що виявило глобальне зниження рівня IR-ізоформ у SF3B1-K700E.
Рисунок 4. Інтенсивність сільського господарства та підключення до мережі в трьох системах ведення сільського господарства (A і B);Випадковий аналіз лісу (C) і зв’язок між інтенсивністю сільського господарства та колонізацією AMF (D)
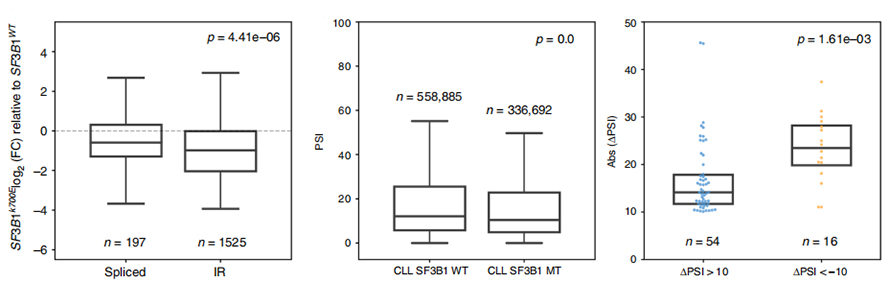
Малюнок 3. Події утримування Intron сильніше знижуються в CLL SF3B1-K700E
технології
Нанопорове довгозчитуване секвенування
Nанопорне секвенування — це технологія секвенування електричних сигналів однієї молекули в реальному часі.
Dдволанцюгова ДНК або РНК зв’яжеться з нанопористим білком, вбудованим у біоплівку, і розмотується під проводом моторного білка.
DНитки НА/РНК проходять через білок нанопорового каналу з певною швидкістю під дією різниці напруг.
Mмолекули генерують різні електричні сигнали відповідно до хімічної структури.
RПостійне виявлення послідовностей досягається викликом бази.
Виконання повнорозмірного секвенування транскриптомів
√ Насиченість даними
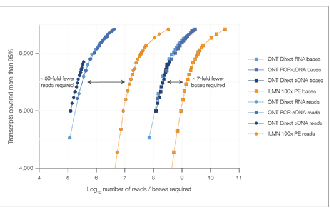
Для досягнення порівнянної насиченості даних потрібно в 7 разів менше читань.
√ Ідентифікація структури транскрипту
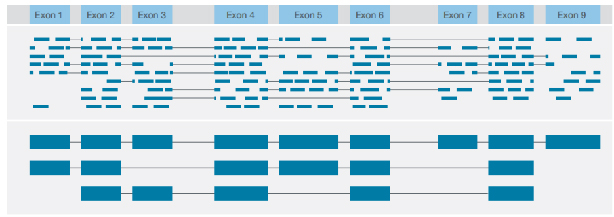
Ідентифікація різноманітних структурних варіантів із консенсусним повним зчитуванням кожного стенограми
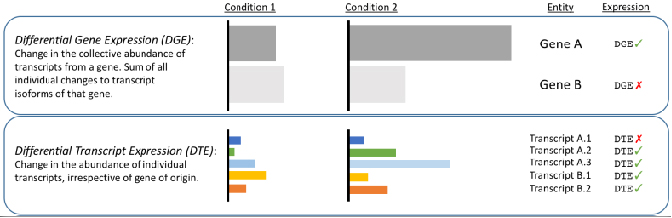
√ Диференціальний аналіз на рівні транскрипту - виявлення змін, прихованих короткими читаннями
довідка
Tang AD, Soulette CM, Baren MJV та ін.Характеристика повнорозмірного транскрипту мутації SF3B1 при хронічному лімфоцитарному лейкозі виявила зниження регуляції збережених інтронів [J].Комунікації природи.
Техніка та основні моменти має на меті поділитися найновішим успішним застосуванням різних високопродуктивних технологій секвенування в різних дослідницьких сферах, а також блискучими ідеями в експериментальному дизайні та аналізі даних.
Час публікації: 08 січня 2022 р

