GENOME EVOLUTION
kinaiyahan
KOMUNIKASYON
Ang mga han-ay sa genome nagpadayag sa mga ruta sa pagkatibulaag sa kalibutan ug nagsugyot sa mga panagsama nga pagpahiangay sa genetic sa ebolusyon sa seahorse
PacBio |Illumina |Hi-C |WGS |Genetic Diversity |Kasaysayan sa Demograpiko |Pag-agos sa Gene
Ang Pacbio sequencing, genome de novo assembly ug annotation services gihatag sa Biomarker Technologies.
Mga highlight
1. Usa ka taas nga kalidad nga chromosome-level seahorse (Hippocampus erectus) genome nga adunay contig N50 nga 15.5 Mb nakuha.
2. Kinatibuk-ang 358 ka genome gikan sa tibuok kalibutan nga 21 ka espisye sa seahorse ang gisunod.
3. Ang mga seahorse nga milambo sa ulahing bahin sa Oligocene ug ang misunod nga circum-global nga kolonisasyon nga mga ruta giila ug nalambigit sa pagbag-o sa dynamics sa mga sulog sa dagat ug paleotemporal nga pagbukas sa dagat
4. Ang genetic nga basehan sa nagbalikbalik nga "bony spines" adaptive phenotype nalambigit sa independente nga mga pagpuli sa usa ka yawe nga gene sa pag-uswag.
5. Ang pag-rafting pinaagi sa mga sulog sa dagat nagbayad sa dili maayo nga pagkatibulaag ug ang paspas nga pagpahiangay nagpadali sa pagkolonya sa bag-ong mga puy-anan
Mga nahimo
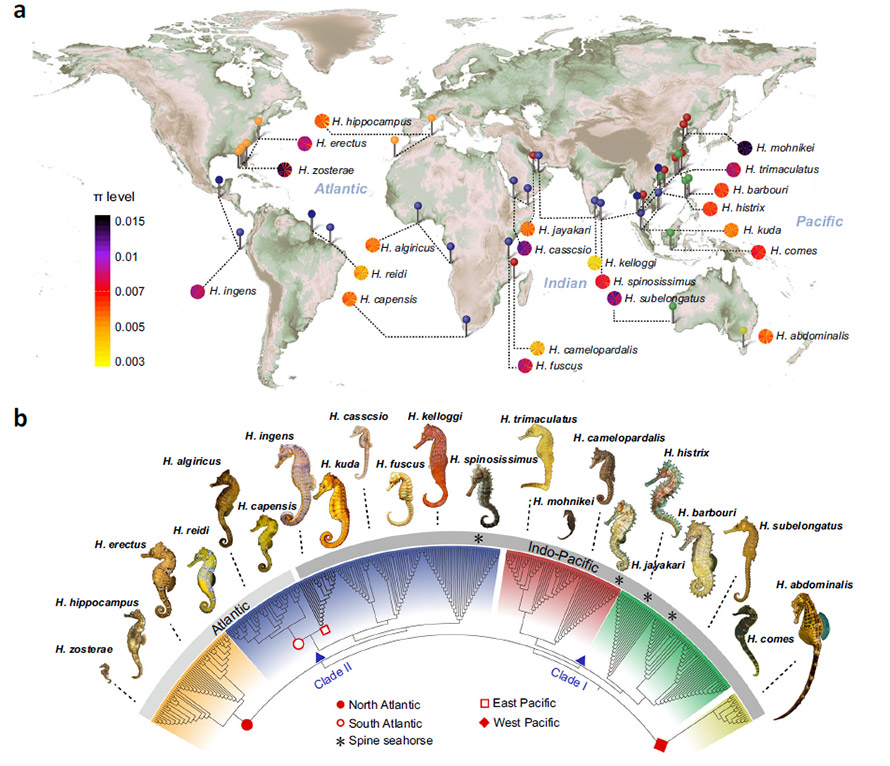
Fig. 1 Genetic diversity ug phylogenetic nga relasyon sa 358 ka seahorse specimens
aGeographic nga sampling nga mga lokasyon alang sa mga sampol nga seahorse nga adunay mga pattern sa nucleotide diversity (π) sa 21 ka seahorse species sa 22 ka chromosome.b Ang silingang kahoy nga gitukod gamit ang genome-wide SNP nga 358 ka mga seahorse.Ang mga simbolo sa pin sa lokasyon sa (a) ug background sa sanga sa (b) katumbas sa usag usa.
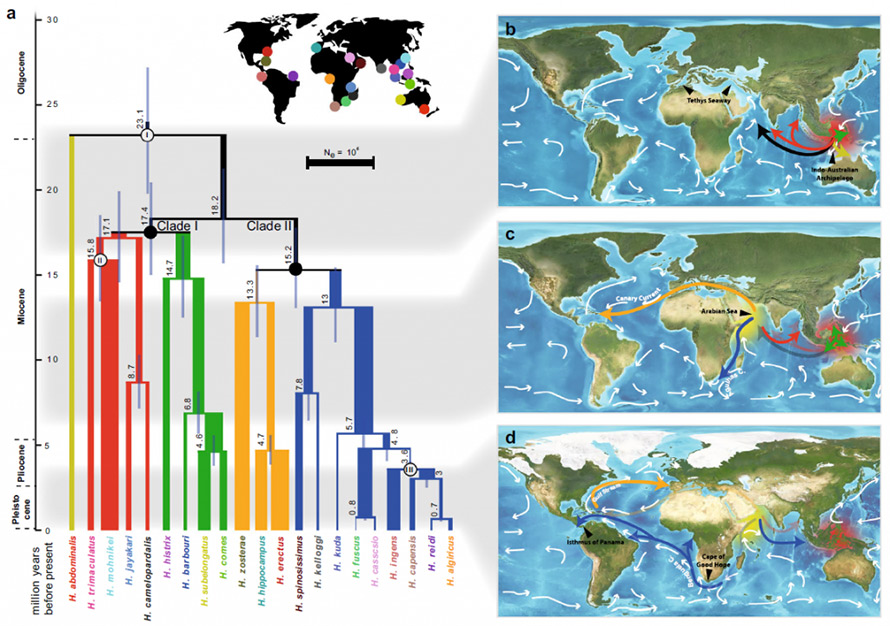
Fig. 2 Kolonisasyon ug kasaysayan sa demograpiko sa mga seahorse
aAng phylogenetic tree ug divergence time gibanabana alang sa 21 ka espisye sa seahorse.Ang gibag-on sa linya sa sanga katumbas sa gibanabana nga gidak-on sa populasyon (Ne) ug ang mga kolor nagpakita sa lainlaing mga linya.Ang mga simbolo I–III nagpakita sa mga punto sa pagkakalibrate.b–d Gitagna nga mga ruta sa kolonisasyon (kolor nga mga arrow) sa mga seahorse base sa divergence time, distribution, vicariance events, ug mga sulog sa dagat (white arrow).b Ang Indo-Australian Archipelago mao ang sentro sa gigikanan (pula nga marka) sa genus Hippocampus sa wala pa ang mga seahorse nagkadaiya ug nagkatag sa tibuok kalibutan 18–23 Ma.c Ang mga seahorse sa sinugdan mikolonya sa Dagat Atlantiko pinaagi sa pagbukas sa dagat sa Tethyan, nga, human sa pagsira niini (Terminal nga Hitabo sa panahon sa 7–13 Ma), nagbulag niining kaliwat nga Tethyan gikan sa kaliwat niini nga igsoon sa Indian Ocean.Ang naulahi, sa ulahi paspas nga nagkadaiya (yellow nga marka) sa Dagat sa Arabia, nga nagtukod usa ka ikaduha nga sentro sa paglainlain sa seahorse.d Ang ikaduhang seahorse colonization event sa Atlantic Ocean nahitabo gikan sa Indian Ocean mga 5Ma pinaagi sa pag-agi sa South Africa nga tumoy, ug sa katapusan miabot sa East Pacific Ocean pinaagi sa bukas pa nga Panama seaway nga gibana-bana nga 3.6 Ma.
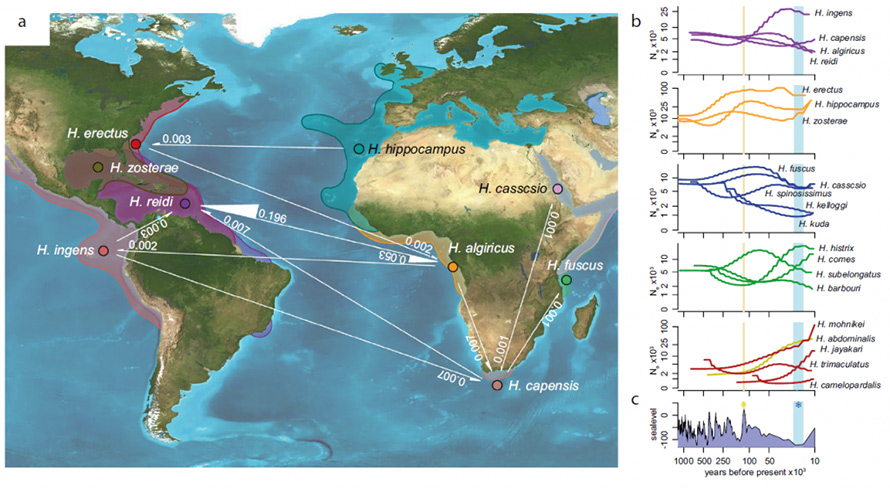
Fig. 3 Ang dagan sa gene ug pag-usab-usab sa epektibong gidak-on sa populasyon
aAng dagan sa gene nakit-an tali sa mga espisye nga nagpuyo sa South Atlantic Ocean.Gipakita ang dagan sa gene duol sa puti nga mga linya ingon nga ang rate sa paglalin nga nakuha sa G-PhoCS.Ang gibag-on ug direksyon sa mga udyong katumbas sa mga rate ug direksyon sa dagan sa gene, matag usa.b Pag-usab-usab sa epektibong gidak-on sa populasyon sa PSMC.Ang x axis nagrepresentar sa oras sa mga tuig sa wala pa ang karon samtang ang y axis nagrepresentar sa epektibo nga gidak-on sa populasyon.Ang mga tsart giorganisar sa panguna sumala sa geograpikanhong pag-apod-apod alang sa matag usa sa mga espisye nga adunay lainlaing mga lugar sa pag-apod-apod.c Pagbag-o sa lebel sa dagat sa miaging 1 ka milyon ka tuig sa metro33.Ang yellow nga linya nagpaila sa katapusang global interglacial peak samtang ang cyan shade nagpakita sa katapusang glacial maximum period.
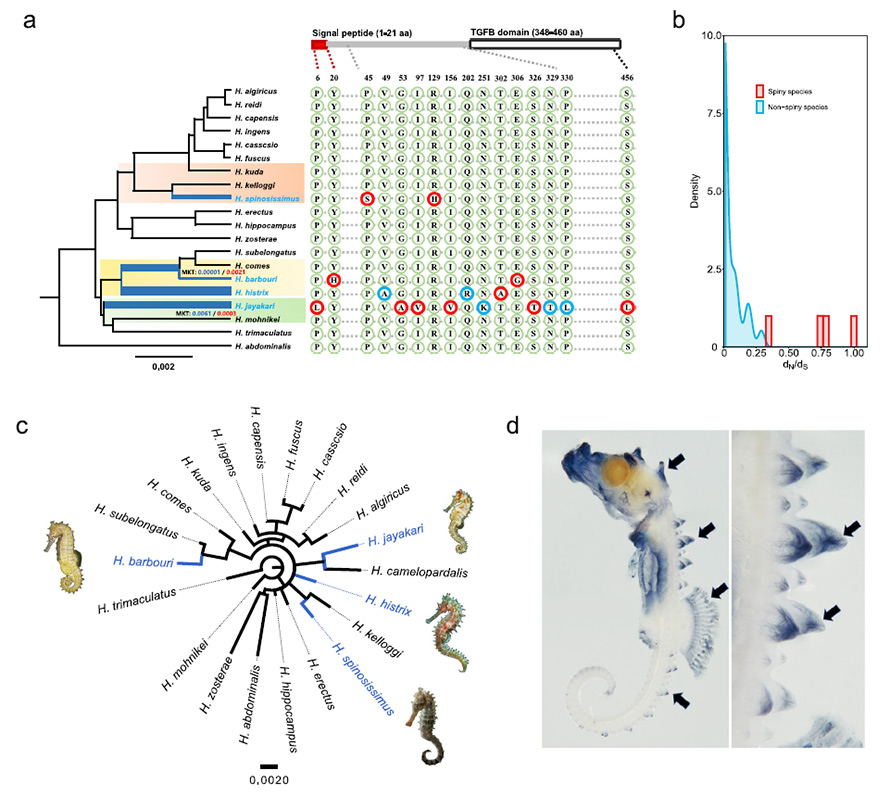
Fig. 4 Ang ebolusyon sa mga tunok.
aSa wala, Species tree nga nagpakita sa independente nga ebolusyon sa mga spine sa mga seahorse.Ang gitas-on sa sanga nagpakita sa gidaghanon sa mga substitusyon matag site.Upat ka spiny seahorse species ang gipasiugda sa asul.Ang mas baga nga mga sanga katumbas sa mas taas nga rate sa nonsynonymous-to-synonymous substitutions (dN/dS) para sa bmp3 gene.Ang Canonical ug generalized McDonald ug Kreitman test (MKT) alang sa bmp3 gene gihimo alang sa tulo ka pairwise sister species nga adunay divergent spiny ug non-spiny features nga gipasiugda sa mga kolor sa background, kansang lebel sa kahulogan gipakita sa p value nga adunay blue ug red font, matag usa.Sa tuo, ang pagtandi sa amino acid substitutions sa bmp3 protein, polymorphic ug fixed substitutions sa spiny seahorse gipakita sa pula ug asul nga mga lingin, matag usa.b Distribution sa dN/dS values sa bmp3 sa spiny seahorse kumpara sa non-spiny species.c Independent nga ebolusyon sa phylogenetic tree nga gitukod pag-usab alang sa protina nga gi-encode sa bmp3.d Whole-mount in situ hybridization sa bmp3 sa Hippocampus erectus.
Reperensya
Li C ug uban pa.Ang mga han-ay sa genome nagpadayag sa mga ruta sa pagkatibulaag sa kalibutan ug nagsugyot sa mga panagsama nga pagpahiangay sa genetic sa ebolusyon sa seahorse.Nat Commun.2021 Peb 17;12(1):1094.
Mga Balita ug Highlight nagtumong sa pagpaambit sa pinakabag-o nga malampuson nga mga kaso sa Biomarker Technologies, pagkuha sa nobela nga mga kalampusan sa siyensya ingon man sa mga prominenteng teknik nga gigamit sa panahon sa pagtuon.
Oras sa pag-post: Ene-06-2022

