EVOLUZIONE DEL GENOMA
natura
COMUNICAZIONI
Le sequenze del genoma rivelano percorsi di dispersione globale e suggeriscono adattamenti genetici convergenti nell'evoluzione dei cavallucci marini
PacBio |Illumina |Ciao-C |WGS |Diversità genetica |Storia demografica |Flusso genico
I servizi di sequenziamento Pacbio, assemblaggio del genoma de novo e annotazione sono stati forniti da Biomarker Technologies.
Punti salienti
1. È stato ottenuto un genoma di cavalluccio marino (Hippocampus erectus) a livello cromosomico di alta qualità con contig N50 di 15,5 Mb.
2. Sono stati risequenziati un totale di 358 genomi di 21 specie di cavallucci marini di tutto il mondo.
3. I cavallucci marini evoluti nel tardo Oligocene e le successive rotte di colonizzazione circum-globale vengono identificati e collegati alle mutevoli dinamiche delle correnti oceaniche e delle aperture paleotemporali delle vie marittime
4. La base genetica del fenotipo adattivo ricorrente delle “spine ossee” è collegata a sostituzioni indipendenti in un gene chiave dello sviluppo.
5.Il rafting attraverso le correnti oceaniche compensa la scarsa dispersione e il rapido adattamento facilita la colonizzazione di nuovi habitat
Risultati
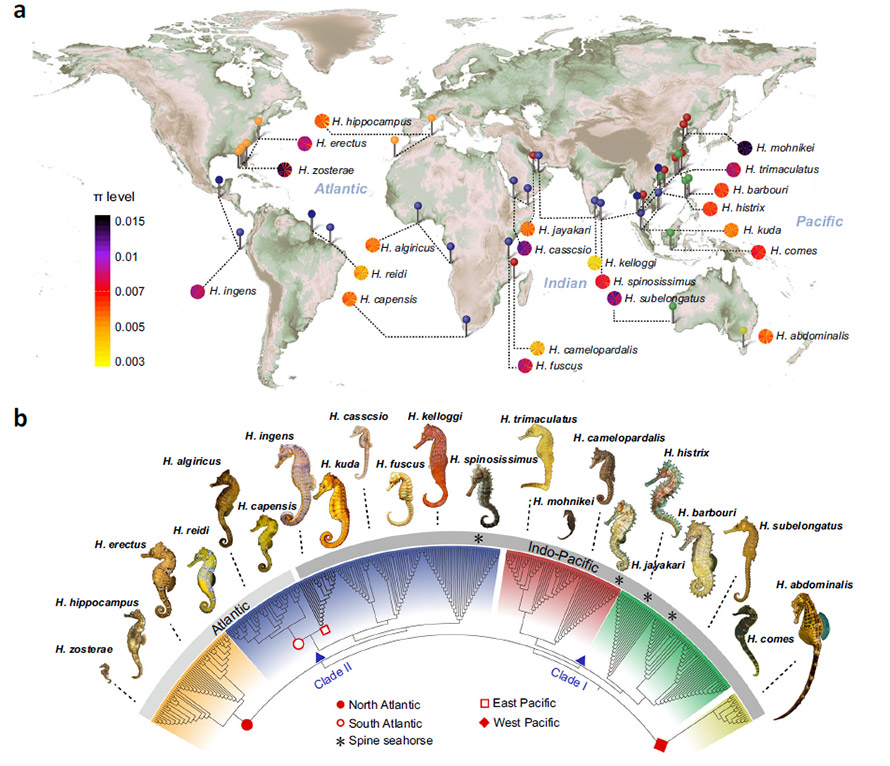
Fig. 1 Diversità genetica e relazioni filogenetiche di 358 esemplari di cavalluccio marino
UNPosizioni geografiche di campionamento per i cavallucci marini campionati con modelli di diversità nucleotidica (π) delle 21 specie di cavallucci marini su 22 cromosomi.b Albero adiacente costruito con SNP dell'intero genoma di 358 cavallucci marini.I simboli dei perni di posizione in (a) e lo sfondo del ramo in (b) corrispondono tra loro.
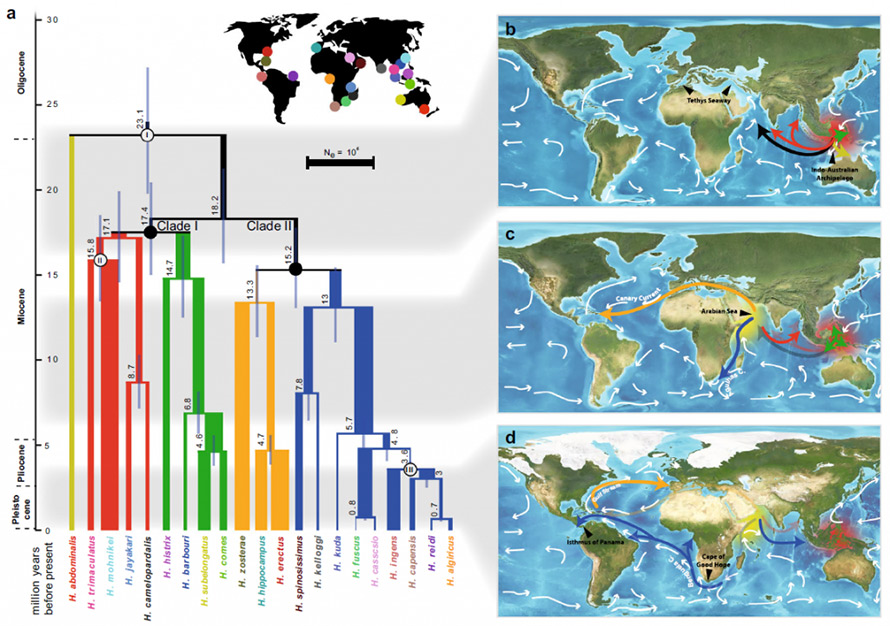
Fig. 2 Colonizzazione e storia demografica dei cavallucci marini
UNAlbero filogenetico e stime del tempo di divergenza per 21 specie di cavallucci marini.Lo spessore della linea di diramazione corrisponde alle stime della dimensione della popolazione (Ne) e i colori indicano lignaggi diversi.I simboli I–III indicano i punti di calibrazione.b – d Rotte di colonizzazione previste (frecce colorate) dei cavallucci marini in base al tempo di divergenza, alla distribuzione, agli eventi di vicarianza e alle correnti oceaniche (frecce bianche).b L'arcipelago indo-australiano era il centro di origine (segno rosso) del genere Hippocampus prima che i cavallucci marini si diversificassero e si disperdessero a livello globale 18-23 milioni di anni fa.c I cavallucci marini colonizzarono inizialmente l'Oceano Atlantico attraverso l'apertura della via marittima di Tetide, che, dopo la sua chiusura (evento terminale durante 7-13 milioni di anni fa), separò questo lignaggio di Tetide dal suo lignaggio gemello dell'Oceano Indiano.Quest'ultimo, successivamente, si è rapidamente diversificato (marcatura gialla) nel Mar Arabico, costituendo un secondo centro di diversificazione dei cavallucci marini.d Un secondo evento di colonizzazione dei cavallucci marini dell'Oceano Atlantico si è verificato dall'Oceano Indiano a circa 5 Ma, passando per la punta sudafricana e infine arrivando nell'Oceano Pacifico orientale attraverso la via marittima di Panama, ancora aperta, a circa 3,6 Ma.
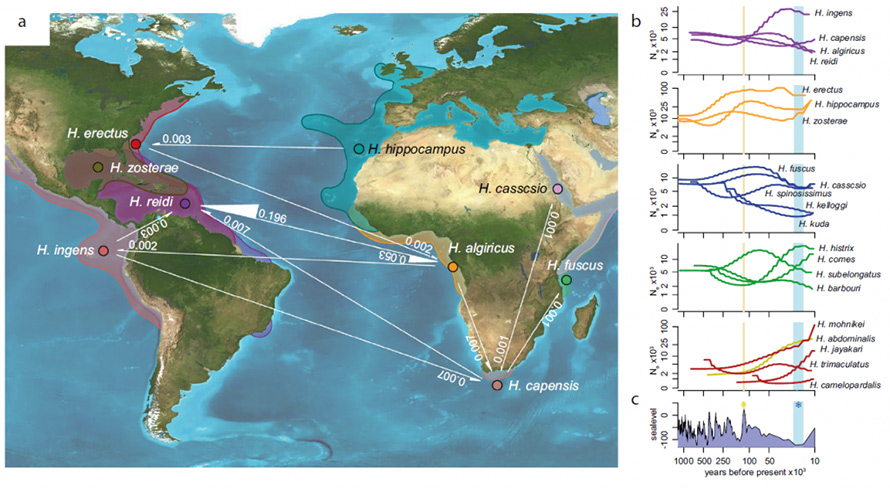
Fig. 3 Flusso genico e fluttuazioni nella dimensione effettiva della popolazione
UNFlusso genetico rilevato tra specie che abitano l'Oceano Atlantico meridionale.Il flusso genico è mostrato vicino alle linee bianche come velocità di migrazione dedotta da G-PhoCS.Lo spessore e la direzione delle frecce corrispondono rispettivamente alla velocità e alla direzione del flusso genico.b Fluttuazioni nella dimensione effettiva della popolazione per PSMC.L'asse x rappresenta il tempo in anni prima del presente mentre l'asse y rappresenta la dimensione effettiva della popolazione.Le carte sono organizzate principalmente in base alla distribuzione geografica per ciascuna delle specie con aree di distribuzione diverse.c Variazione del livello del mare durante l'ultimo milione di anni in metri33.La linea gialla indica l'ultimo picco interglaciale globale mentre la tonalità ciano indica l'ultimo periodo massimo glaciale.
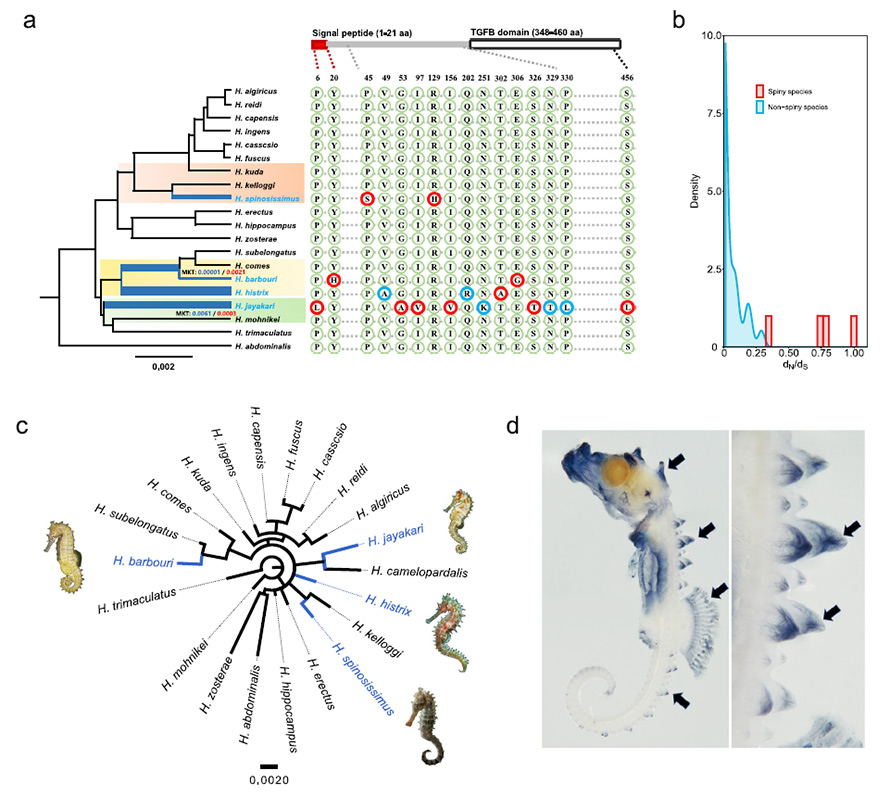
Fig. 4 L'evoluzione delle spine.
UNA sinistra, albero delle specie che mostra l'evoluzione indipendente delle spine nei cavallucci marini.La lunghezza del ramo indica il numero di sostituzioni per sito.Quattro specie di cavallucci marini spinosi sono evidenziate in blu.I rami più spessi corrispondono a tassi più elevati di sostituzioni da non sinonimi a sinonimi (dN/dS) per il gene bmp3.Il test canonico e generalizzato di McDonald e Kreitman (MKT) per il gene bmp3 è stato eseguito per tre specie sorelle a coppie con caratteristiche spinose e non spinose divergenti evidenziate dai colori di sfondo, i cui livelli di significatività erano indicati rispettivamente dal valore p con carattere blu e rosso.A destra, il confronto delle sostituzioni di amminoacidi nella proteina bmp3, le sostituzioni polimorfiche e fisse nei cavallucci marini spinosi sono indicate rispettivamente con cerchi rossi e blu.b Distribuzione dei valori dN/dS in bmp3 nei cavallucci marini spinosi rispetto alle specie non spinose.c Evoluzione indipendente nell'albero filogenetico ricostruito per la proteina codificata da bmp3.d Ibridazione in situ a montaggio intero di bmp3 nell'ippocampo erectus.
Riferimento
Li C et al.Le sequenze del genoma rivelano percorsi di dispersione globale e suggeriscono adattamenti genetici convergenti nell'evoluzione dei cavallucci marini.Nat Comune.2021 17 febbraio;12(1):1094.
Novità e punti salienti mira a condividere gli ultimi casi di successo con Biomarker Technologies, catturando nuovi risultati scientifici e tecniche importanti applicate durante lo studio.
Orario di pubblicazione: 06-gennaio-2022

