
Sequenziamento metagenomico -NGS
Vantaggi del servizio
● Esente da isolamento e coltivazione per la profilazione della comunità microbica
● Alta risoluzione nel rilevamento di specie a bassa abbondanza nei campioni ambientali
● L'idea di “meta-” integra tutte le caratteristiche biologiche a livello funzionale, a livello di specie e a livello genetico, il che riflette una visione dinamica più vicina alla realtà.
● BMK accumula una vasta esperienza in diversi tipi di campioni con oltre 10.000 campioni elaborati.
Specifiche del servizio
| piattaforma | Sequenziamento | Dati consigliati | Tempo di consegna |
| Piattaforma Illumina NovaSeq | PE150 | 6G/10G/20G | 45 giorni lavorativi |
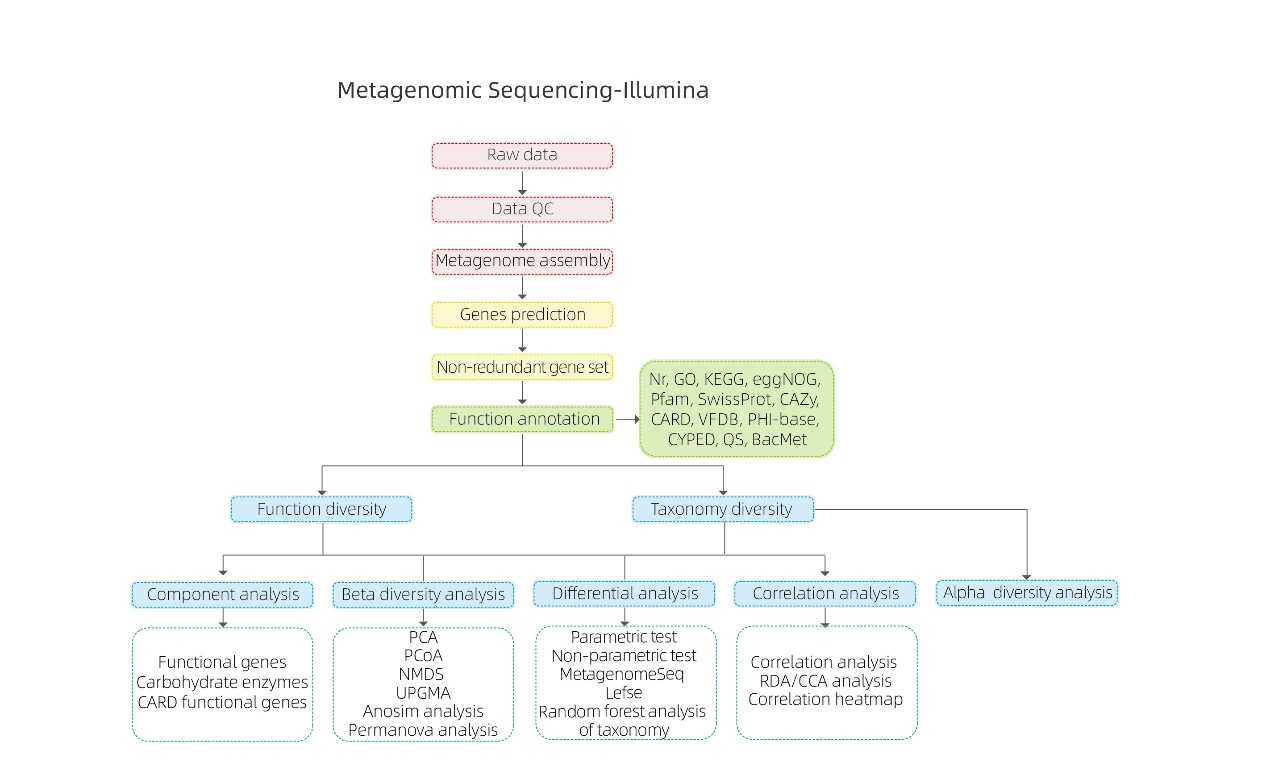
Analisi bioinformatiche
● Controllo della qualità dei dati grezzi
● Assemblaggio del metagenoma
● Set di geni non ridondanti e annotazione
● Analisi della diversità delle specie
● Analisi della diversità delle funzioni genetiche
● Analisi intergruppo
● Analisi di associazione con fattori sperimentali
Requisiti e consegna del campione
Requisiti del campione:
PerEstratti di DNA:
| Tipo di campione | Quantità | Concentrazione | Purezza |
| Estratti di DNA | > 30 ng | > 1 ng/μl | DE260/280= 1,6-2,5 |
Per i campioni ambientali:
| Tipo di campione | Procedura di campionamento consigliata |
| Suolo | Importo campione: ca.5 g;La sostanza appassita rimanente deve essere rimossa dalla superficie;Macinare pezzi grandi e passare attraverso un filtro da 2 mm;Campioni aliquoti in provette EP sterili o cirotubi per la prenotazione. |
| Feci | Importo campione: ca.5 g;Raccogliere e aliquotare i campioni in provette EP sterili o criotubi per la prenotazione. |
| Contenuto intestinale | I campioni devono essere trattati in condizioni asettiche.Lavare il tessuto raccolto con PBS;Centrifugare il PBS e raccogliere il precipitante in provette EP. |
| Fango | Importo campione: ca.5 g;Raccogliere e aliquotare il campione di fango in una provetta EP sterile o in una crioprovetta per la prenotazione |
| Corpo d'acqua | Per campioni con una quantità limitata di microbi, come acqua di rubinetto, acqua di pozzo, ecc., raccogliere almeno 1 litro di acqua e passare attraverso un filtro da 0,22 μm per arricchire la flora microbica sulla membrana.Conservare la membrana in un tubo sterile. |
| Pelle | Raschiare accuratamente la superficie della pelle con un batuffolo di cotone sterile o una lama chirurgica e inserirla in una provetta sterile. |
Consegna del campione consigliata
Congelare i campioni in azoto liquido per 3-4 ore e conservarli in azoto liquido o a -80 gradi per una prenotazione a lungo termine.È richiesta la spedizione del campione con ghiaccio secco.
Flusso di lavoro del servizio

Consegna del campione

Costruzione della biblioteca

Sequenziamento

Analisi dei dati

Servizi post-vendita
1.Istogramma: distribuzione delle specie
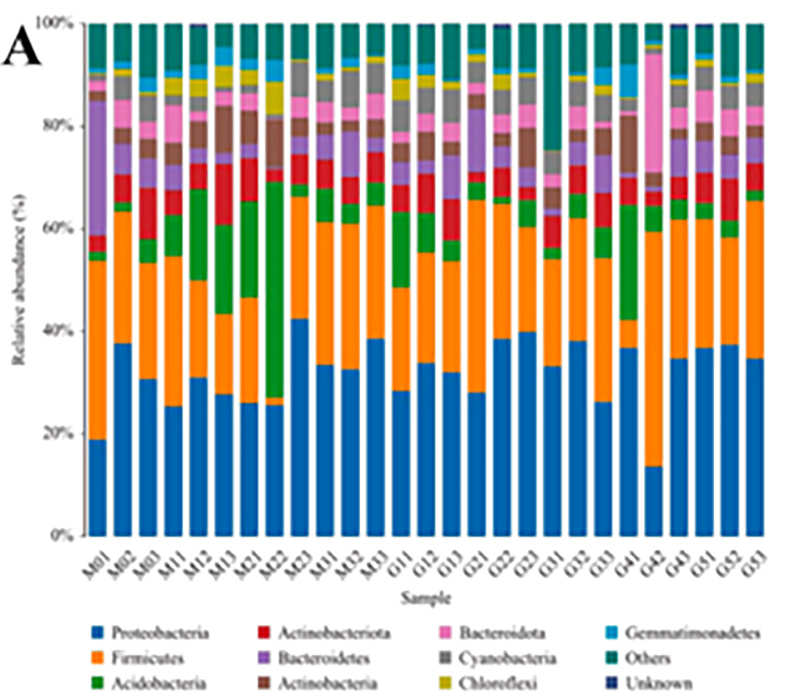
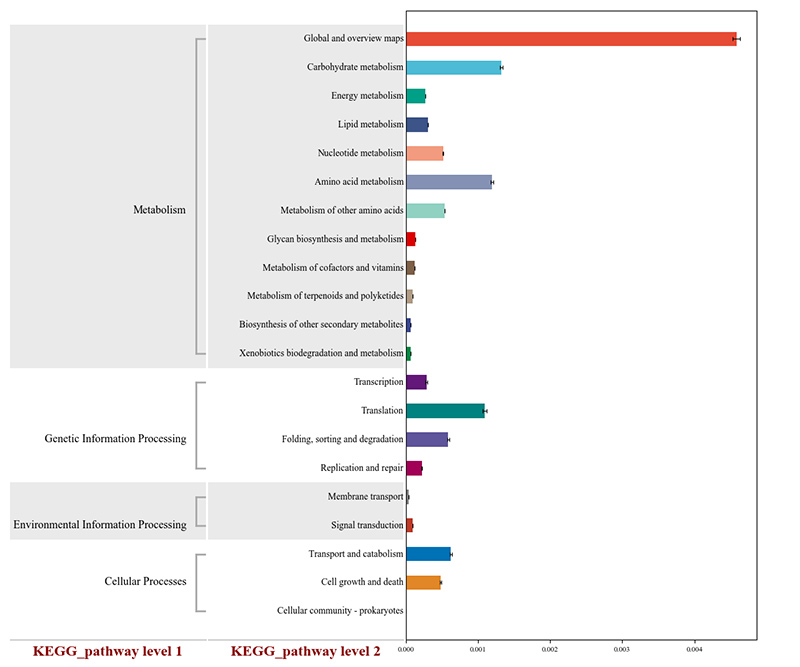
2. Geni funzionali annotati nelle vie metaboliche KEGG
3.Mappa termica: funzioni differenziali basate sull'abbondanza relativa dei geni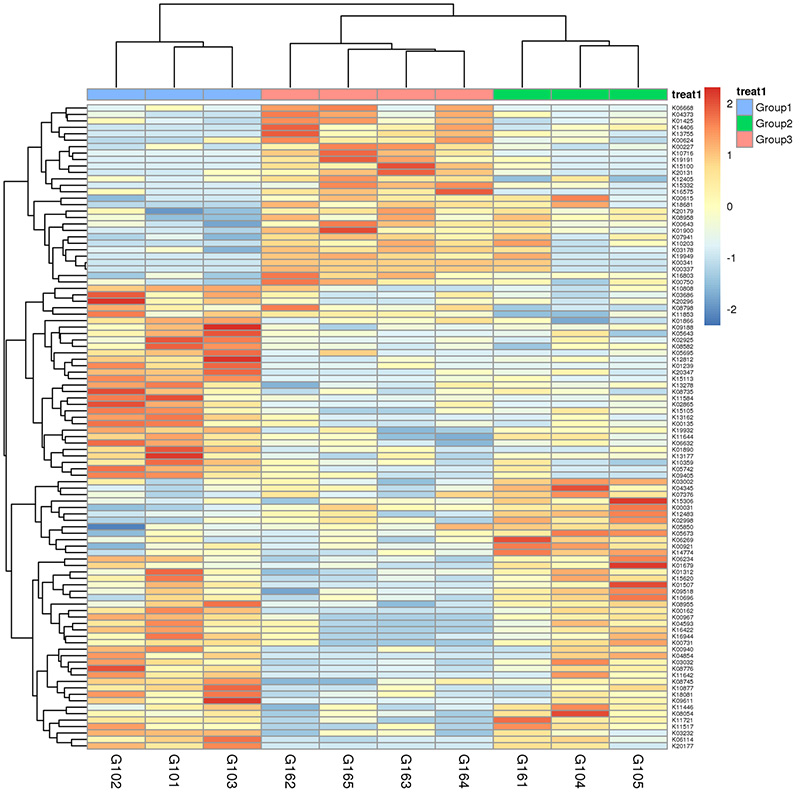 4.Circo di geni di resistenza agli antibiotici CARD
4.Circo di geni di resistenza agli antibiotici CARD
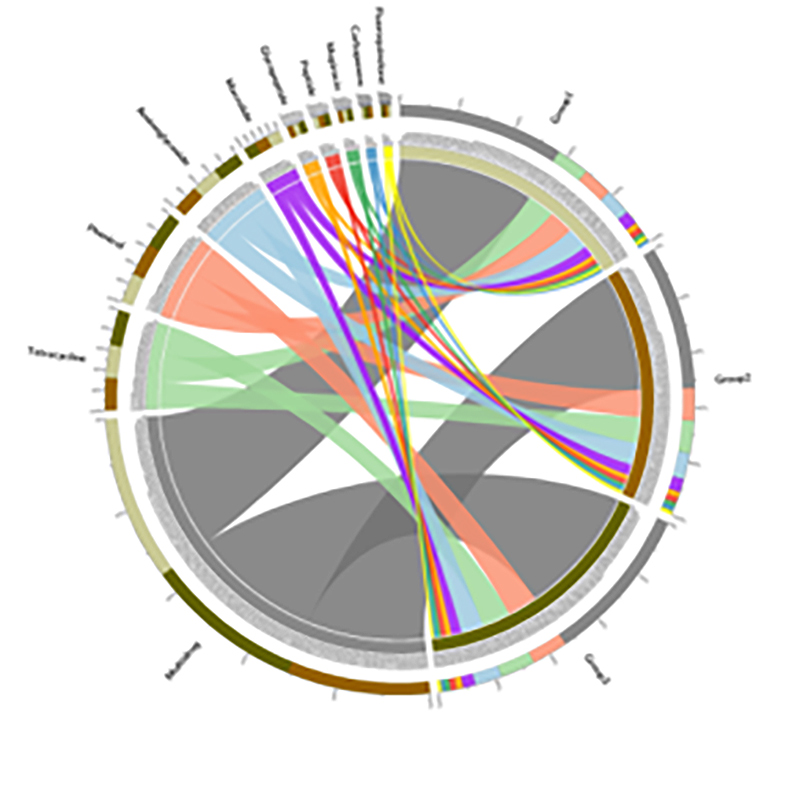
Caso BMK
Prevalenza di geni di resistenza agli antibiotici e di agenti patogeni batterici lungo il continuum delle radici del suolo-mangrovie
Pubblicato:Giornale dei materiali pericolosi, 2021
Strategia di sequenziamento:
Materiali: estratti di DNA di quattro frammenti di campioni associati a radici di mangrovie: compartimenti di suolo non piantato, rizosfera, episfera ed endosfera
Piattaforma: Illumina HiSeq 2500
Obiettivi: metagenoma
Regione V3-V4 del gene rRNA 16S
Risultati chiave
Sono stati elaborati il sequenziamento metagenomico e il profilo metabarcoding sul continuum suolo-radice degli alberelli di mangrovie per studiare la diffusione dei geni di resistenza agli antibiotici (ARG) dal suolo alle piante.I dati metagenomici hanno rivelato che il 91,4% dei geni di resistenza agli antibiotici erano comunemente identificati in tutti e quattro i compartimenti del suolo sopra menzionati, il che mostrava un andamento continuo.Il sequenziamento dell'amplicone dell'rRNA 16S ha generato 29.285 sequenze, che rappresentano 346 specie.Combinando la profilazione delle specie mediante sequenziamento degli ampliconi, si è scoperto che questa diffusione è indipendente dal microbiota associato alle radici, tuttavia potrebbe essere facilitata dalla mobilità degli elementi genetici.Questo studio ha identificato il flusso di ARG e agenti patogeni dal suolo alle piante attraverso il continuum suolo-radice interconnesso.
Riferimento
Wang, C., Hu, R., Strong, PJ, Zhuang, W., & Shu, L..(2020).Prevalenza di geni di resistenza agli antibiotici e di agenti patogeni batterici lungo il continuum delle radici suolo-mangrovie.Giornale dei materiali pericolosi, 408, 124985.











