Metagenomica (NGS)
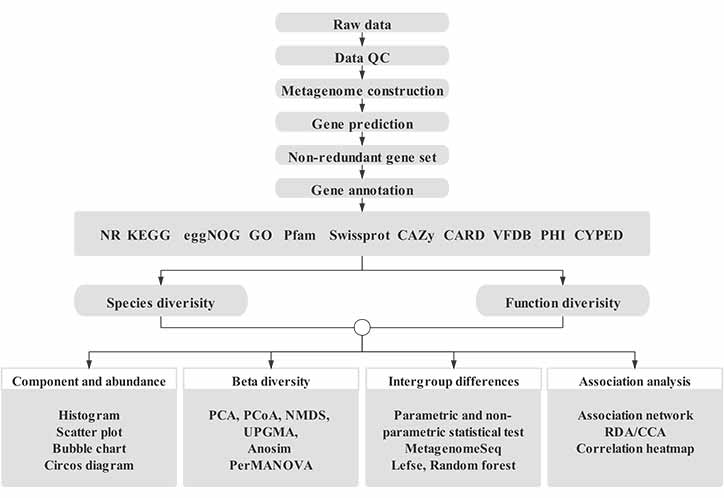
Questa piattaforma di analisi è progettata per l'analisi dei dati metagenomici shotgun sulla base di anni di esperienza.Consiste in un flusso di lavoro integrato contenente varie analisi metagenomiche comunemente necessarie, tra cui elaborazione dei dati, studi a livello di specie, studi a livello di funzione genetica, binning del metagenoma, ecc. Inoltre, strumenti di data mining personalizzati sono disponibili nel flusso di lavoro di analisi standard, inclusa query su geni e specie , impostazione dei parametri, generazione di figure personalizzate, ecc.
Identificazione della trascrizione
Flusso di lavoro della bioinformatica