GENOOMI EVOLUTSIOON
loodusgeneetika
Kvaliteetne genoomikomplekt tõstab esile rukki genoomilised omadused ja agronoomiliselt olulised geenid
PacBio |Illumina |Bionano optiline kaart |Hi-C genoomikomplekt |Geneetiline kaart |Valikuline pühkimine |RNA-Seq |ISO-seq |SLAF-seq
Biomarker Technologies pakkus selles uuringus tehnilist tuge Pacbio sekveneerimise, Hi-C järjestuse ja andmete analüüsi kohta.
Esiletõstmised
1.Saadi esimene kromosoomitasemel kvaliteetne rukki genoom, mille ühe kromosoomi suurus on suurem kui 1 Gb.
2. Võrreldes Tu, Aet ja Hv genoomiga, täheldati rukki genoomis ainulaadset hiljutist LTR-RT sündmust, mis oli vastutav rukki genoomi suuruse laienemise eest.
3. Erinevus rukki ja diploidse nisu vahel toimus pärast odra eraldamist nisust, kusjuures kahe sündmuse lahknemisajad olid ligikaudu 9,6 ja 15 MYA.
FT geenide fosforüülimine võib kontrollida rukki varajast suundumust.
4. Selektiivne pühkimisanalüüs näitab ScID1 võimalikku osalust rubriigi kuupäeva reguleerimises ja selle tõenäolist valikut rukkis kodustamise teel
Taust
Taust
Rukis on väärtuslik toidu- ja söödakultuur, oluline geneetiline ressurss nisu ja tritikale parandamiseks ning asendamatu materjal kõrreliste tõhusates genoomikauuringutes.Hiinas kasvatatav varajase õitsemise sort Weining rukis on silmapaistev oma laia toimespektriga vastupidavuse poolest nii jahukaste kui ka triiprooste vastu.Rukki eliidi tunnuste geneetilise ja molekulaarse aluse mõistmiseks ning rukki ja sellega seotud põllukultuuride genoomi- ja aretusuuringute edendamiseks sekveneerisime ja analüüsisime siin Weiningi rukki genoomi.
Saavutused
Rukki genoom
Rukki genoom konstrueeriti PacBio SMRT lugemiste, lühiloetava Illumina sekveneerimise, samuti kromatiini konformatsiooni püüdmise (Hi-C), geneetilise kaardistamise ja BioNano analüüsi põhjal.Kokkupandud kontiigid (7,74 Gb) moodustasid 98,47% hinnangulisest genoomi suurusest (7,86 Gb), kusjuures 93,67% kontiigidest (7,25 Gb) oli määratud seitsmele kromosoomile.Korduvad elemendid moodustasid 90, 31% kokkupandud genoomist.
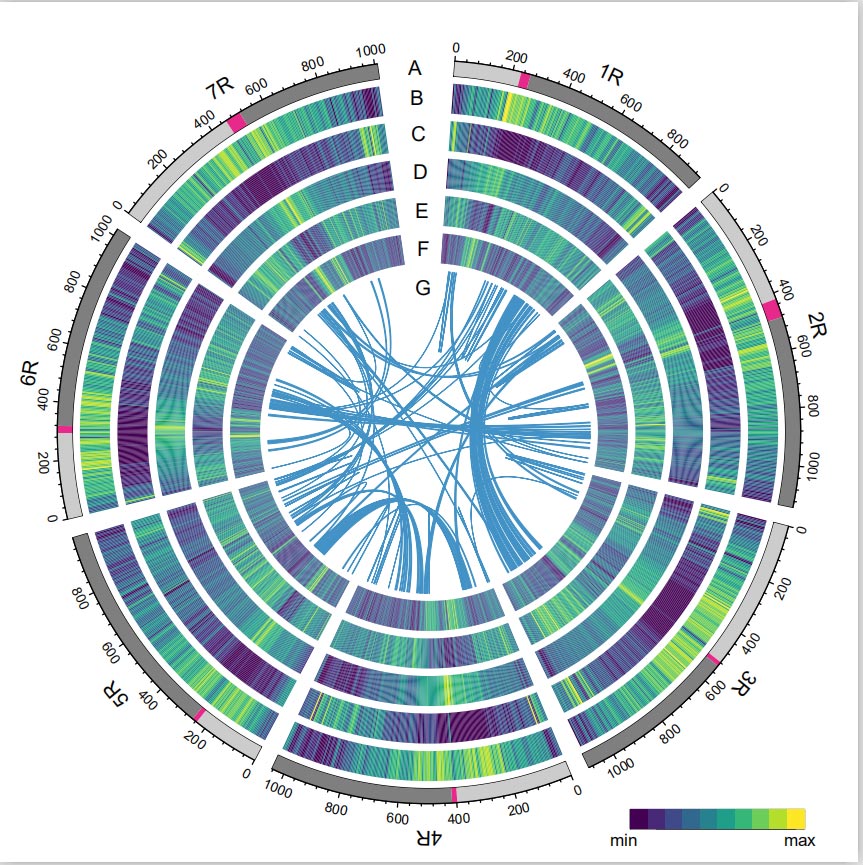
Rukki genoom
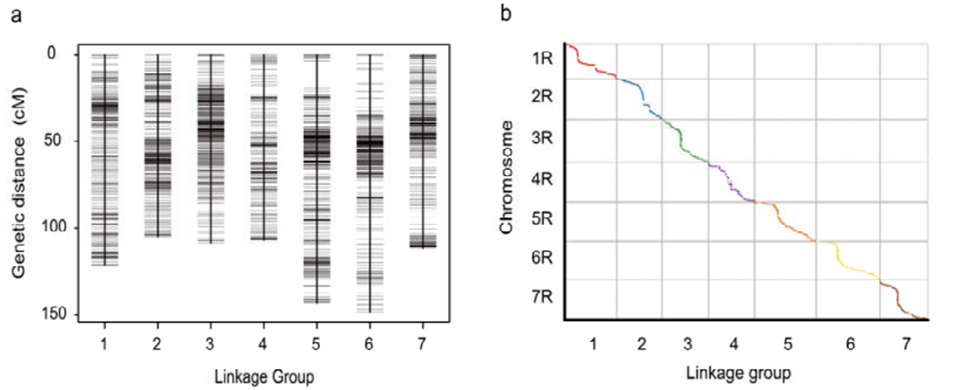
Geneetilise seose kaart (WJ), mis on välja töötatud 295 F2 taime abil, mis on saadud kahe rukki maatõu (Weining × Jingzhou) ristumisest.
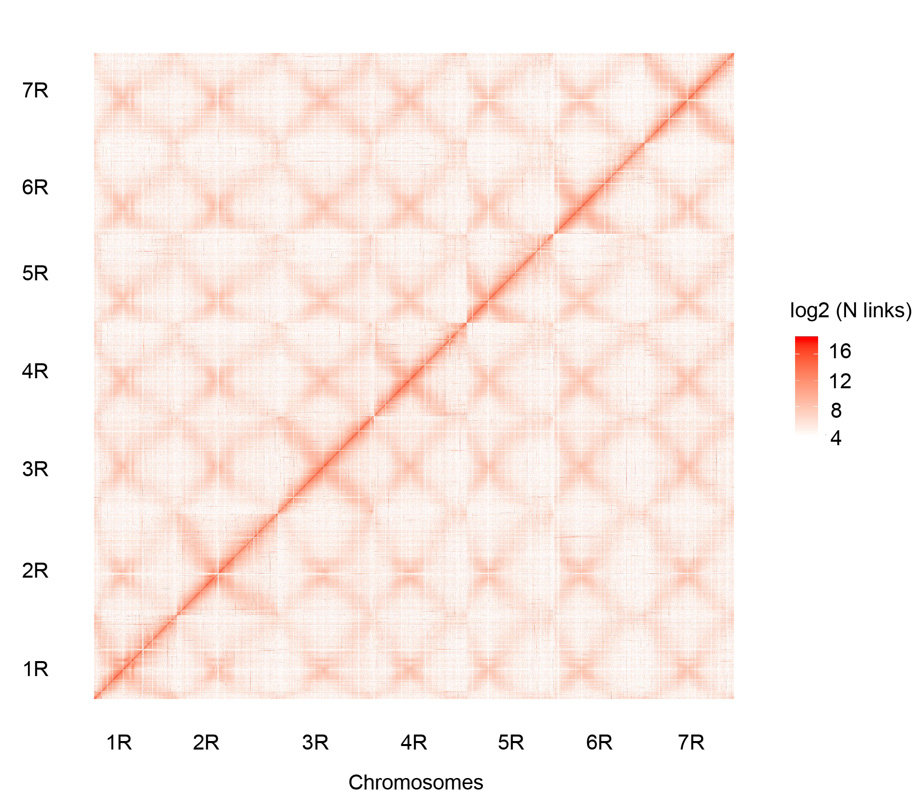
Seitsme kokkupandud Weiningi rukkikromosoomi (1R – 7R) Hi-C kontaktikaart
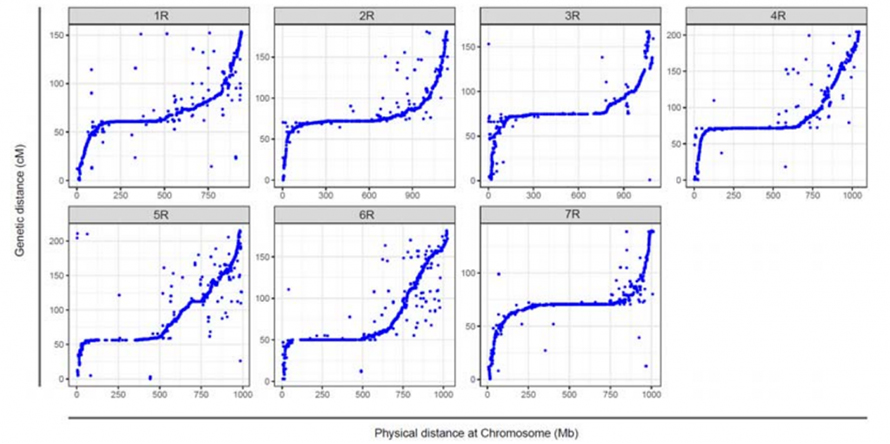
Joondamine Weiningi rukki seitsme kokkupandud kromosoomi ja seitsme rukki sidestusrühma vahel, mis töötati välja Lo7 x Lo255 RIL populatsiooni abil
Rukki genoomi LTR Assembly Index (LAI) väärtus leiti olevat 18,42 ja 1393 (96,74%) 1440 kõrgelt konserveerunud BUSCO geenist tuvastati. Need tulemused viitavad sellele, et Weiningi rukki genoomi järjestus on mõlemas intergeenses geenis kõrge kvaliteediga. ja geneetilised piirkonnad.Kokku ennustati 86 991 valku kodeerivat geeni, sealhulgas 45 596 kõrge usaldusväärsusega (HC) geeni ja 41 395 madala usaldusväärsusega geeni (LC) geeni.
2. TE-de analüüs
TE-de analüüs.TE-dena märgiti kokku 6, 99 Gb, mis moodustab 90, 31% Weiningi komplektist, mis sisaldas 2 671 941 elementi, mis kuulusid 537 perekonda.See TE sisaldus oli selgelt kõrgem kui varem teatatud Ta (84, 70%), Tu (81, 42%), Aet (84, 40%), WEW (82, 20%) või Hv (80, 80%) puhul.Pikad terminaalsed korduvad retrotransposoonid (LTR-RT), sealhulgas Gypsy, Copia ja klassifitseerimata RT elemendid, olid domineerivad TE-d ning 1 hõivasid 84,49% annoteeritud TE sisaldusest ja 76,29% kokkupandud Weiningi genoomist;CACTA DNA transposoonid olid suuruselt teisel kohal, moodustades 11, 68% annoteeritud TE sisaldusest ja 10, 55% kokkupandud Weiningi genoomist.
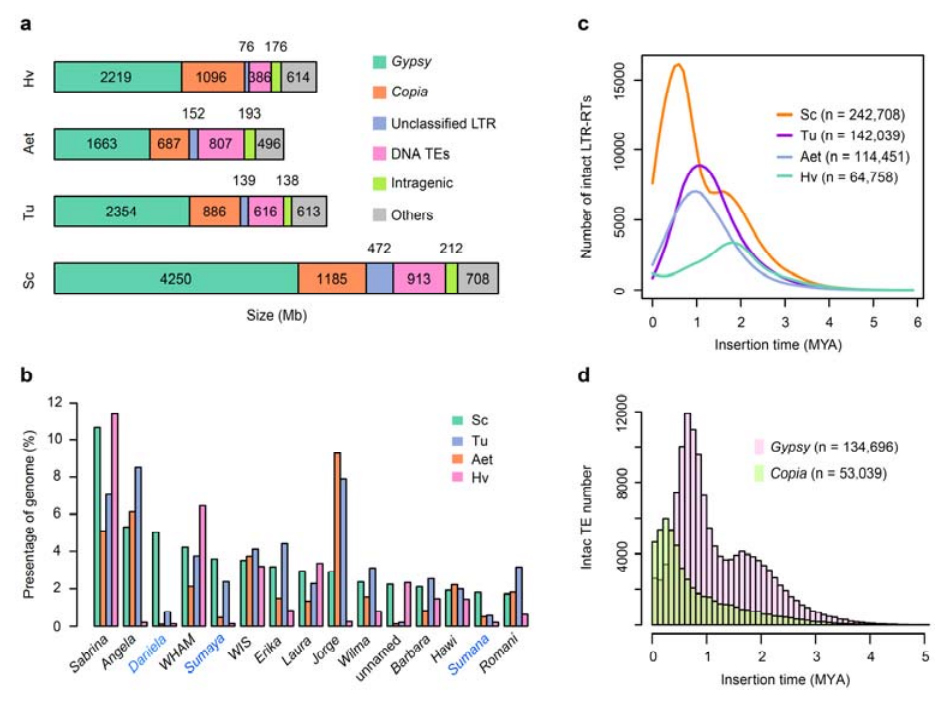
Rukki transposoonelementide analüüs
Weiningi rukkil oli suhteliselt suur osa hiljutistest LTR-RT-dest, mille amplifikatsiooni tipp ilmnes umbes 0,5 miljonit aastat tagasi (MYA), mis oli nelja liigi seas kõige värskem;teine tipp, mis esines ligikaudu 1,7 MYA, oli vanem ja seda täheldati ka odral.Superperekonna tasandil leiti Weiningi rukkis väga hiljutised Copia elementide pursked 0, 3 MYA juures, samas kui mustlaste RT-de võimendused kujundasid domineerivalt LTR-RT purske dünaamika bimodaalset jaotusmustrit.
3. Rukki genoomi evolutsiooni ja kromosoomide sünteeside uurimine
Erinevus rukki ja diploidse nisu vahel toimus pärast odra eraldamist nisust, kusjuures kahe sündmuse lahknemisajad olid vastavalt ligikaudu 9,6 ja 15 MYA.1R, 2R, 3R olid täiesti kollineaarsed vastavalt nisu 1., 2. ja 3. rühma kromosoomidega.4R, 5R, 6R, 7R leiti, et eksisteerivad suuremahulised fusioonid ja segmendid.
4. Geenide dubleerimise ja nende mõju tärklise biosünteesi geenidele analüüs
Nimelt oli Weining rukki tandemlikult dubleeritud geenide (TDG) ja proksimaalselt dubleeritud geenide (PDG) arv suurem kui Tu, Aet, Hv, Bd ja Os puhul.Transponeeritud dubleeritud geene (TrDG-d) oli samuti rohkem kui neid, mis leiti spetsiaalselt Tu ja Aet jaoks.Rukki genoomi laienemisega kaasneb suurem arv geenide dubleerimist.Suurenenud TE-pursked rukkis võisid põhjustada TrDG-de arvu suurenemist.
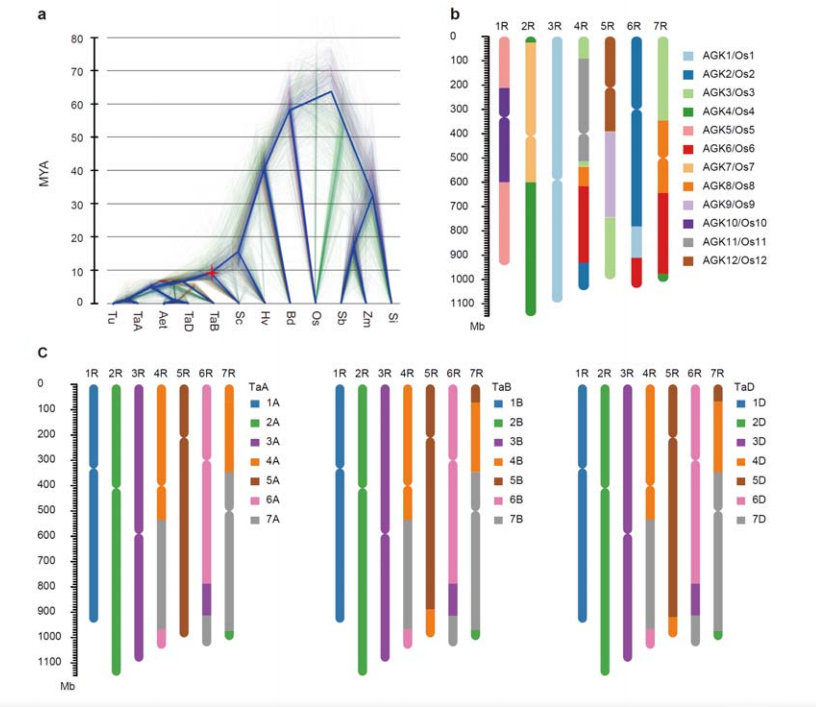
Rukki genoomi evolutsioonilised ja kromosoomi sünteesi analüüsid
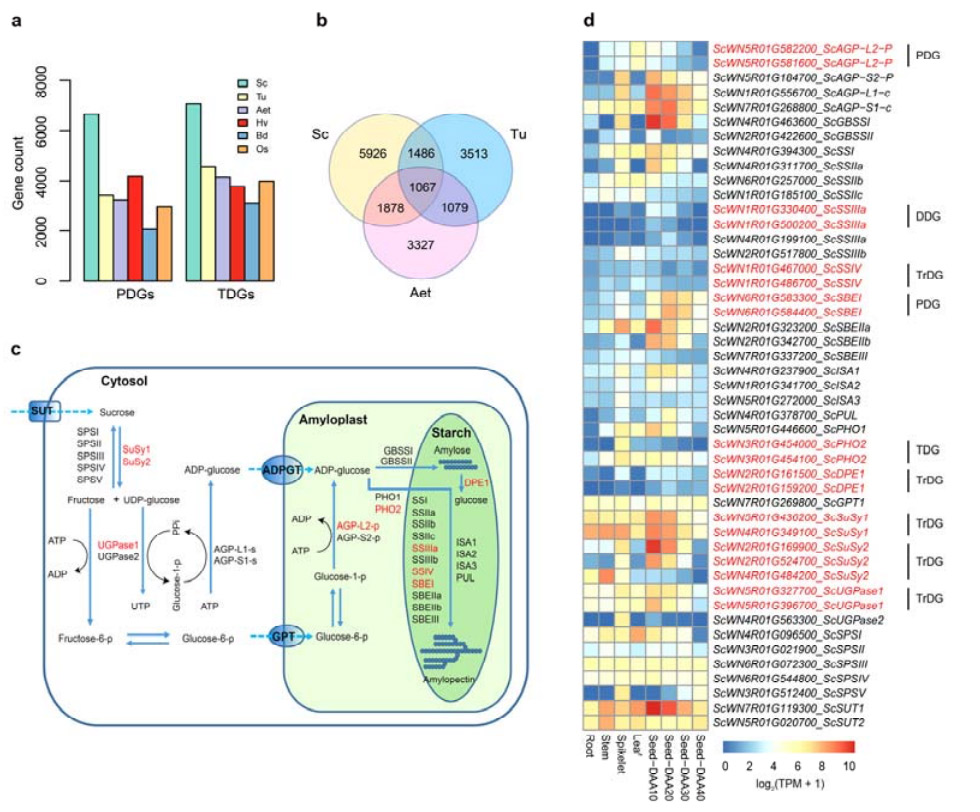
Rukki geenide dubleerimise ja nende mõju tärklise biosünteesiga seotud geenide (SBRG) mitmekesisusele analüüs
5. Rukkiseemne säilitamise valgu (SSP) geeni lookuste dissektsioon
1R või 2R puhul on tuvastatud neli kromosomaalset lookust (Sec-1 kuni Sec-4), mis määravad rukki SSP-d.α-gliadiini geenid arenesid alles hiljuti nisus ja lähedastes liikides pärast nisu ja rukki lahknemist.
6. Transkriptsioonifaktori (TF) ja haigusresistentsuse geenide uurimine
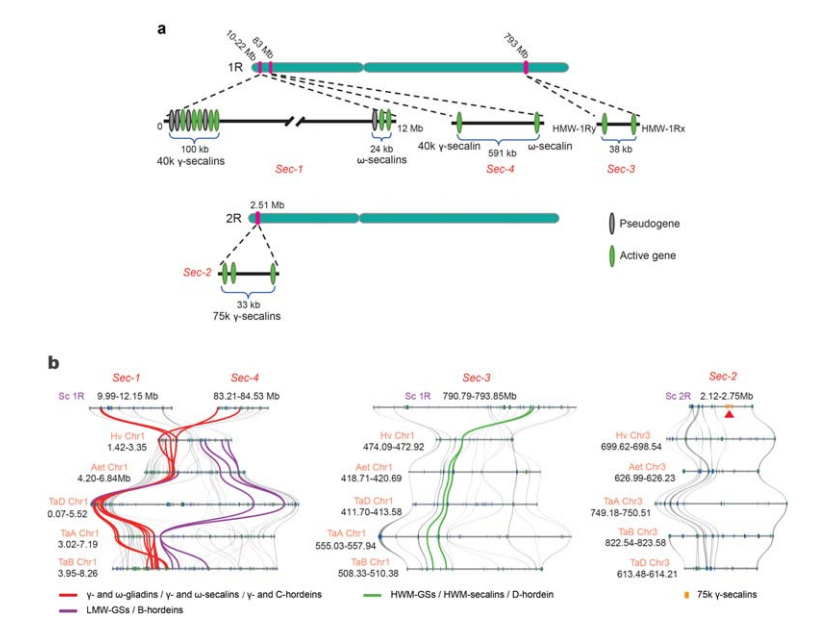
Rukki sekaliini lookuste analüüs
Weiningi rukkil oli rohkem haigusresistentsusega seotud (DRA) geene (1989, lisaandmed 3) kui Tu (1621), Aet (1758), Hv (1508), Bd (1178), Os (1575) ja A (1836) geene. ), pehme nisu B (1728) ja D (1888) subgenoomid.
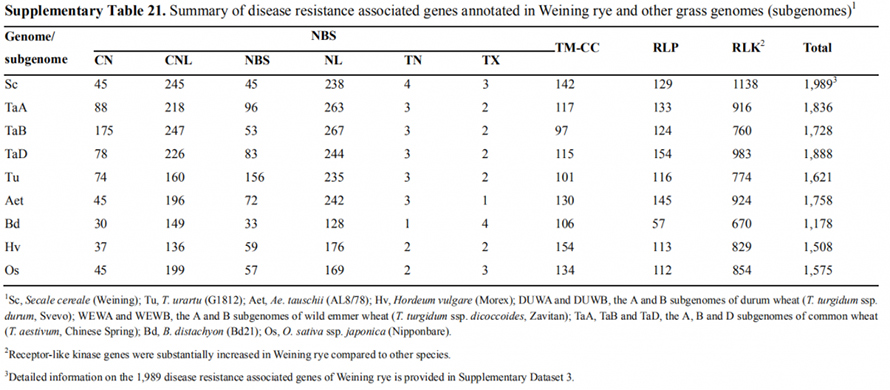
7. Varajase suunatunnusega seotud geeniekspressiooni tunnuste uurimine
Weiningi genoomi koostamisel märgiti kaks FT geeni, millel oli pika päeva tingimustes suhteliselt kõrge ekspressioon, ScFT1 ja ScFT2.ScFT2 (S76 ja T132) fosforüülimise kahel aminohappejäägil leiti seos aeglase kontrolliga
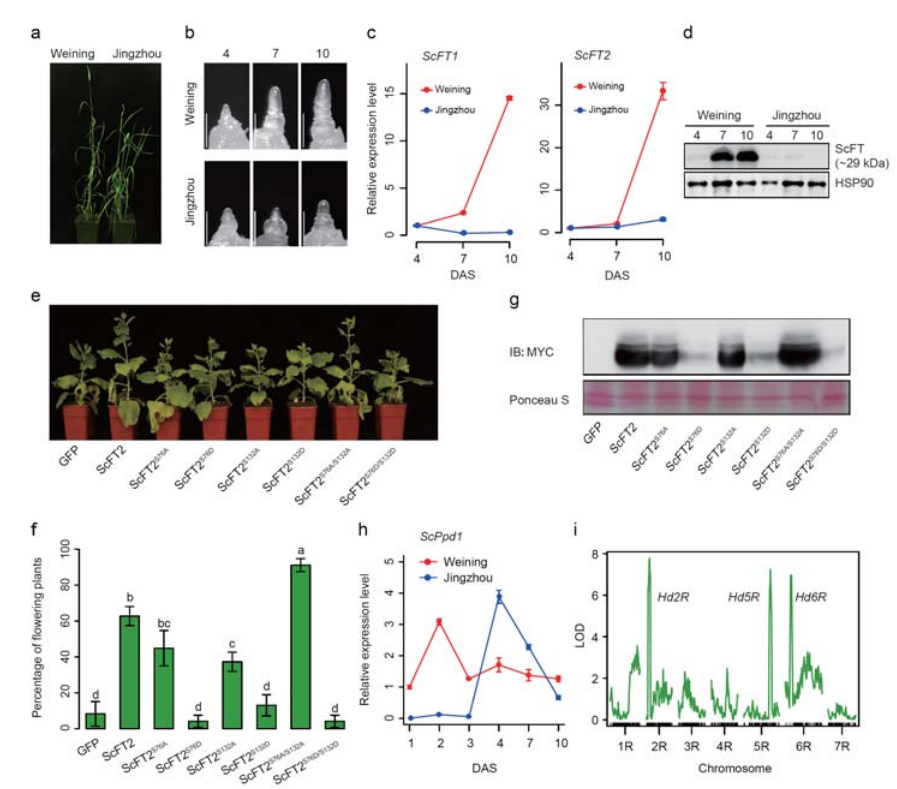
Arengu- ja geeniekspressiooni tunnused, mis on seotud Weiningi rukki varajase peamise tunnusega
8. Rukki kodustamises potentsiaalselt seotud kromosoomipiirkondade ja lookuste kaevandamine
Kultiveeritud rukki ja S. vavilovii vahelise selektiivse analüüsi läbiviimiseks kasutati kokku 123 647 SNP-d.11 selektiivset pühkimissignaali, mis on tuvastatud reduktsiooniindeksi (DRI), fikseerimisindeksi (FST) ja XP-CLR meetodiga.Leiti, et ScID1 võib olla seotud rubriigi kuupäeva reguleerimisega.
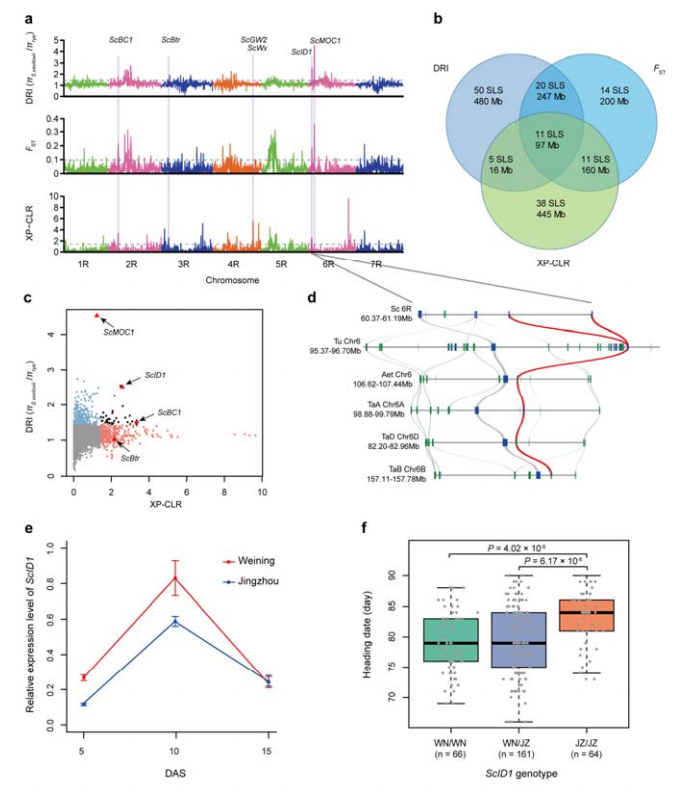
Rukki kodustamisega potentsiaalselt seotud kromosomaalsete piirkondade ja lookuste tuvastamine ja analüüs
Viide
Li GW et al.Kvaliteetne genoomikomplekt tõstab esile rukki genoomilised omadused ja agronoomiliselt olulised geenid.Loodusgeneetika (2021)
Uudised ja tipphetked eesmärk on jagada uusimaid edukaid juhtumeid ettevõttega Biomarker Technologies, jäädvustada nii uudseid teadussaavutusi kui ka uuringu käigus kasutatud silmapaistvaid tehnikaid.
Postitusaeg: jaanuar 05-2022

