GENOMA EVOLŪCIJA
dabas ģenētika
Augstas kvalitātes genoma komplekts izceļ rudzu genoma īpašības un agronomiski svarīgus gēnus
PacBio |Illumina |Bionano optiskā karte |Hi-C genoma montāža |Ģenētiskā karte |Selektīvās slaucīšanas |RNA-Seq |ISO-seq |SLAF-seq
Biomarker Technologies sniedza tehnisko atbalstu Pacbio sekvencēšanai, Hi-C sekvencēšanai un datu analīzi šajā pētījumā.
Izceļ
1.Iegūts pirmais hromosomu līmeņa augstas kvalitātes Rudzu genoms, kura vienas hromosomas izmērs ir lielāks par 1 Gb.
2. Salīdzinot ar Tu, Aet un Hv genomu, rudzu genomā tika novēroti unikāli neseni LTR-RT notikumi, kas bija atbildīgi par rudzu genoma lieluma paplašināšanos.
3. Atšķirība starp rudziem un diploīdiem kviešiem notika pēc miežu atdalīšanas no kviešiem, un divu notikumu atšķirības laiks bija aptuveni 9,6 un 15 MYA.
FT gēnu fosforilācija var kontrolēt rudzu agrīno virziena iezīmi.
4. Selektīvā slaucīšanas analīze norāda uz iespējamu ScID1 iesaistīšanos pozīcijas datuma regulēšanā un tā iespējamo atlasi, pieradinot rudzos
Fons
Fons
Rudzi ir vērtīga pārtikas un lopbarības kultūra, nozīmīgs ģenētiskais resurss kviešu un tritikāles uzlabošanai un neaizstājams materiāls efektīviem salīdzinošajiem genomikas pētījumiem stiebrzālēm.Weining rudzi, agri ziedoša Ķīnā audzēta šķirne, ir izcila, pateicoties tā plaša spektra izturībai gan pret miltrasu, gan svītru rūsu.Lai izprastu rudzu elites īpašību ģenētisko un molekulāro bāzi un veicinātu genoma un selekcijas pētījumus rudziem un radniecīgām kultūrām, mēs šeit sekvencējām un analizējām Weining rudzu genomu.
Sasniegumi
Rudzu genoms
Rudzu genoms tika izveidots, ķemmējot PacBio SMRT nolasījumus, īsi nolasāmo Illumina sekvencēšanu, kā arī tos, kas iegūti no hromatīna konformācijas uztveršanas (Hi-C), ģenētiskās kartēšanas un BioNano analīzes.Samontētie kontigi (7, 74 Gb) veidoja 98, 47% no aplēstā genoma lieluma (7, 86 Gb), un 93, 67% kontigu (7, 25 Gb) tika piešķirti septiņām hromosomām.Atkārtoti elementi veidoja 90, 31% no samontētā genoma.
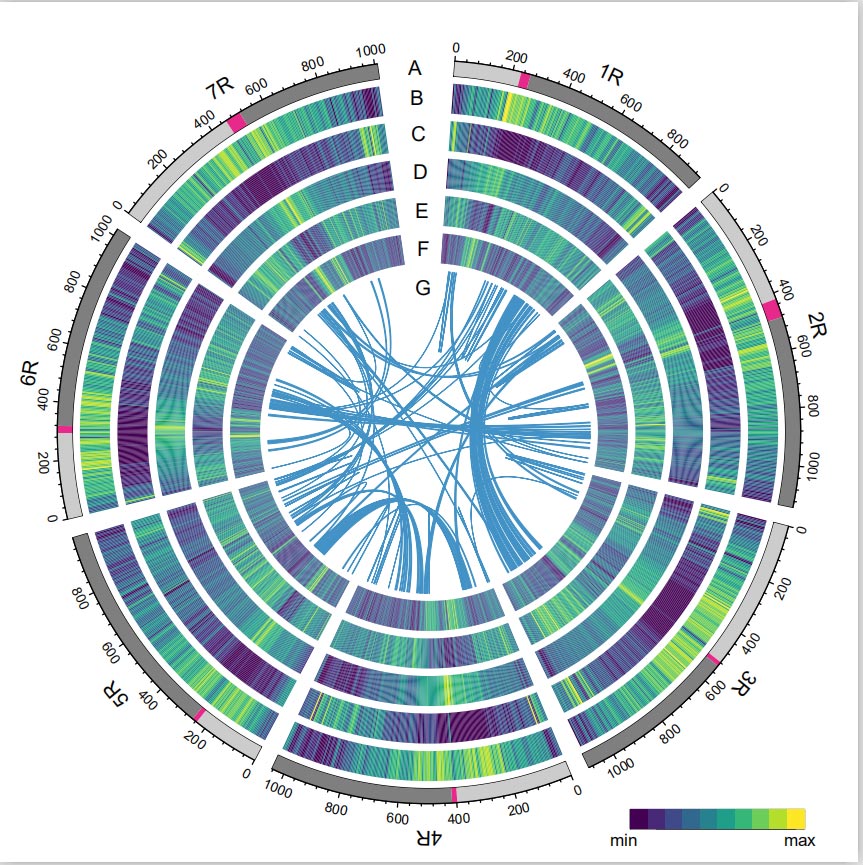
Rudzu genoms
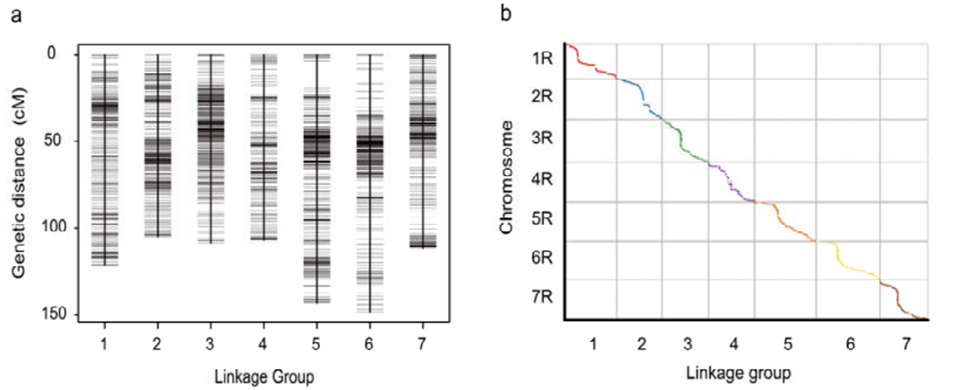
Ģenētiskās saiknes karte (WJ), kas izstrādāta, izmantojot 295 F2 augus, kas iegūti, krustojot divas rudzu zemes rases (Weining × Jingzhou)
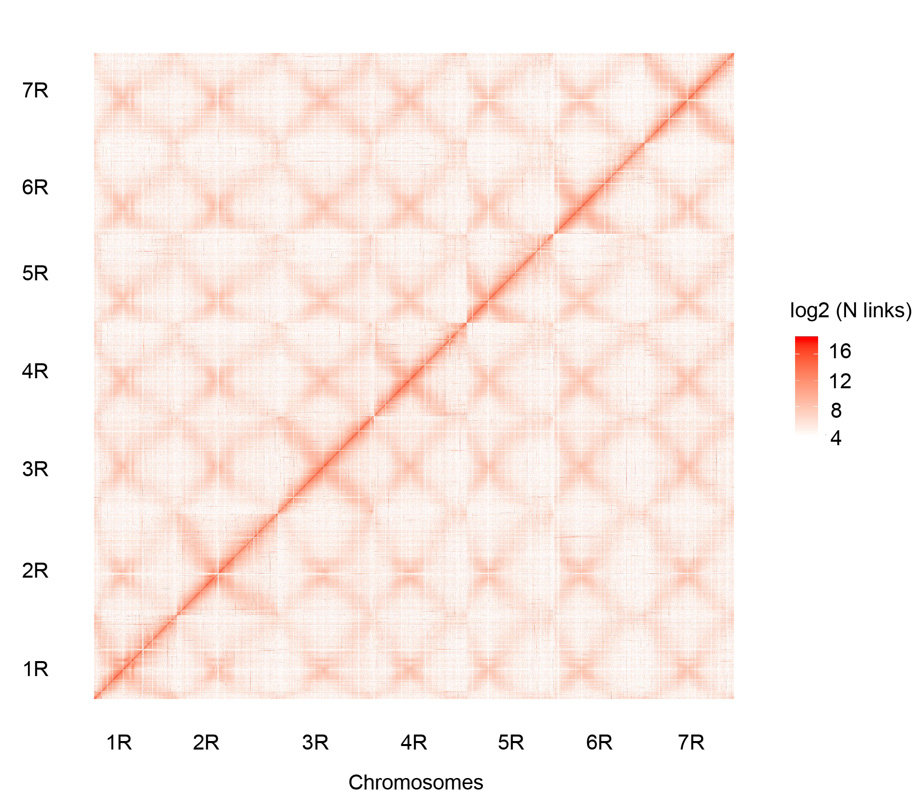
Hi-C kontaktu karte septiņām samontētām Vainingas rudzu hromosomām (1R–7R)
Izlīdzināšana starp septiņām saliktajām Weining rudzu hromosomām un septiņām rudzu saišu grupām, kas izstrādātas, izmantojot Lo7 x Lo255 RIL populāciju
Tika konstatēts, ka rudzu genoma LTR Assembly Index (LAI) vērtība ir 18,42 un tika identificēti 1393 (96,74%) no 1440 ļoti konservētiem BUSCO gēniem. Šie rezultāti liecina, ka Vainingas rudzu genoma secība ir kvalitatīva abos starpgēnos gēnos. un ģenētiskie reģioni.Kopumā tika prognozēts 86 991 proteīnu kodējošs gēns, tostarp 45 596 augstas ticamības (HC) gēni un 41 395 zemas ticamības gēnu (LC) gēni.
2. TE analīze
TE analīze.Kopā 6, 99 Gb, kas veido 90, 31% Weining komplekta, tika anotēti kā TE, kas ietvēra 2 671 941 elementu, kas pieder 537 ģimenēm.Šis TE saturs bija nepārprotami augstāks nekā iepriekš ziņots par Ta (84,70%), Tu (81,42%), Aet (84,40%), WEW (82,20%) vai Hv (80,80%).Garie termināli atkārtojošie retrotranspozoni (LTR-RT), ieskaitot čigānu, kopiju un neklasificētos RT elementus, bija dominējošie TE, un tie aizņēma 84,49% no anotētā TE satura un 76,29% no samontētā Vaininga genoma;CACTA DNS transpozoni bija otrie visizplatītākie TE, kas veidoja 11, 68% no anotētā TE satura un 10, 55% no samontētā Weining genoma.
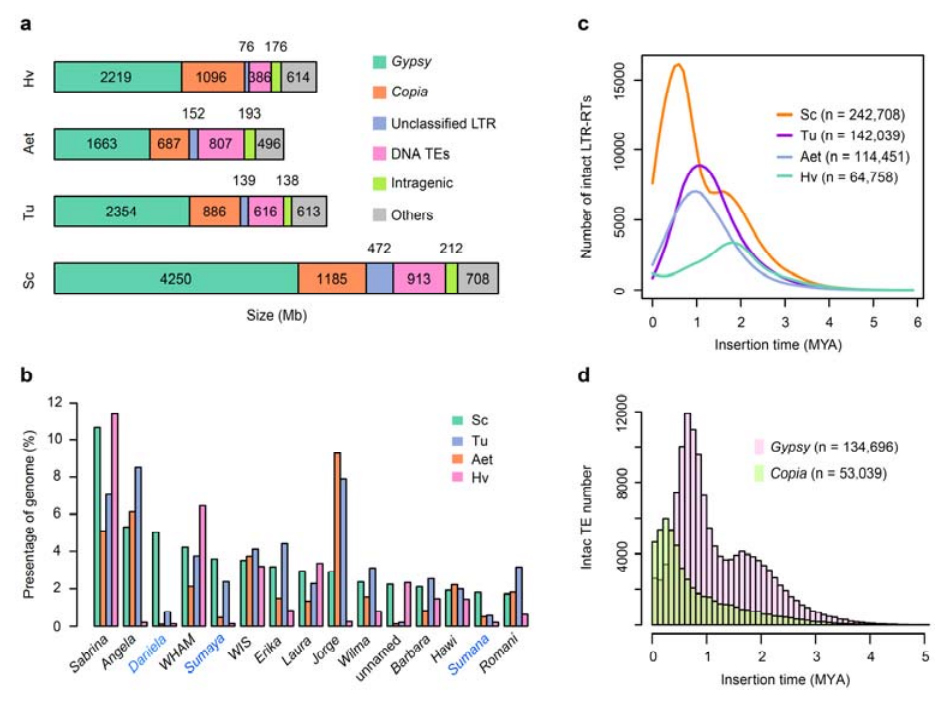
Rudzu transposona elementu analīze
Weining rudziem bija salīdzinoši liels neseno LTR-RT ievietojumu īpatsvars ar pastiprinājuma maksimumu pirms aptuveni 0,5 miljoniem gadu (MYA), kas bija jaunākais no četrām sugām;otrs maksimums, kas bija aptuveni 1,7 MYA, bija vecāks un tika novērots arī miežiem.Virsģimenes līmenī tika konstatēti pavisam nesen Copia elementu uzliesmojumi Vainingas rudzos ar 0, 3 MYA, savukārt čigānu RT pastiprinājumi dominēja LTR-RT pārrāvuma dinamikas bimodālā sadalījuma modeli.
3. Rudzu genoma evolūcijas un hromosomu sintēnu izpēte
Atšķirība starp rudziem un diploīdiem kviešiem notika pēc miežu atdalīšanas no kviešiem, un abu notikumu atšķirības laiks bija attiecīgi aptuveni 9, 6 un 15 MYA.1R, 2R, 3R bija pilnībā kolineāri ar kviešu 1., 2. un 3. grupas hromosomām.4R, 5R, 6R, 7R tika konstatēti liela mēroga saplūšanas un segmenti.
4. Gēnu dublēšanās un to ietekmes uz cietes biosintēzes gēniem analīze
Jāatzīmē, ka Weining rudzu tandēmā dublēto gēnu (TDG) un proksimāli dublēto gēnu (PDG) skaits bija lielāks nekā Tu, Aet, Hv, Bd un Os.Transponēto dublēto gēnu (TrDG) bija arī vairāk nekā to, kas īpaši atrasti Tu un Aet.Rudzu genoma paplašināšanos pavada lielāks gēnu dublēšanās skaits.Palielinātie TE uzliesmojumi rudzos varēja izraisīt paaugstinātu TrDG skaitu.
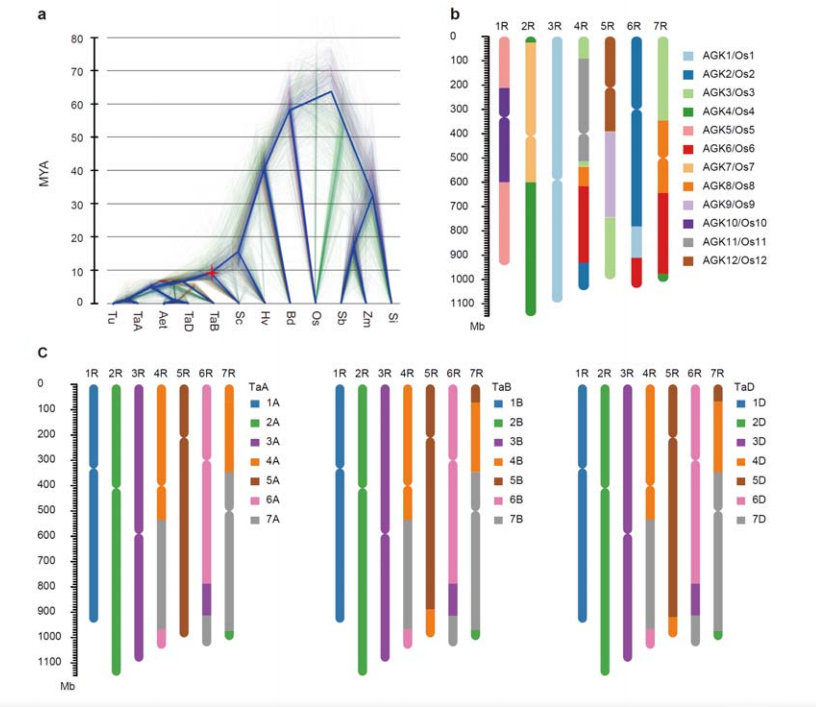
Rudzu genoma evolūcijas un hromosomu sintēzes analīzes
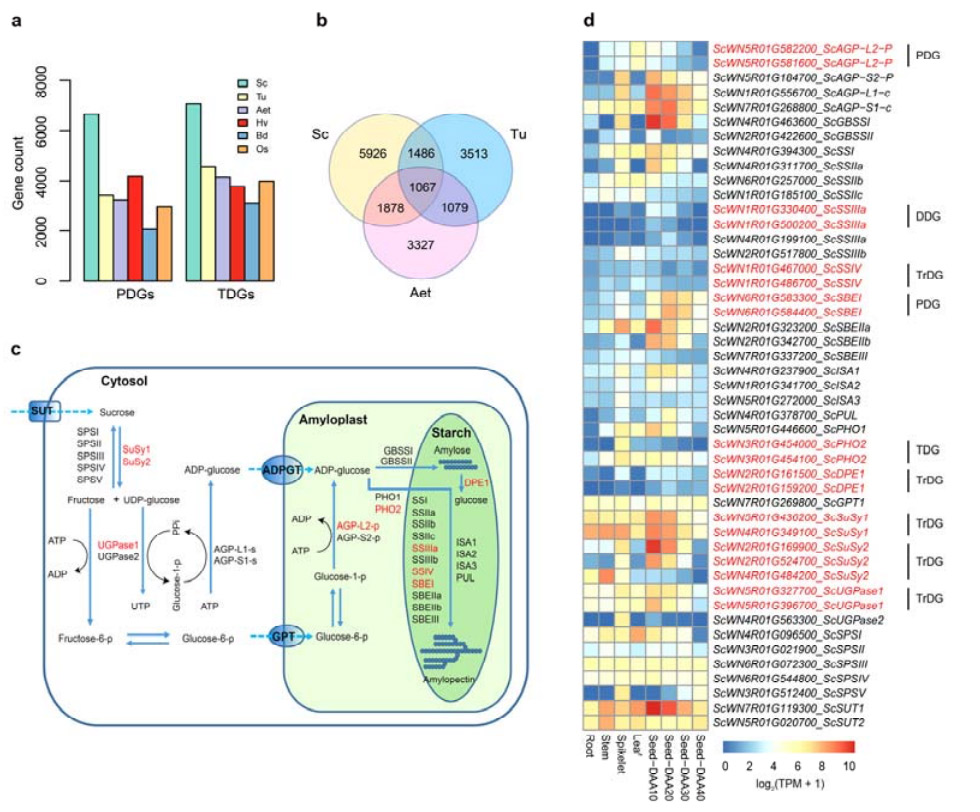
Rudzu gēnu dublēšanās analīze un to ietekme uz cietes biosintēzes gēnu (SBRG) daudzveidību
5. Rudzu sēklu uzglabāšanas proteīna (SSP) gēnu lokusu sadalīšana
Uz 1R vai 2R ir identificēti četri hromosomu loki (Sec-1 līdz Sec-4), kas norāda rudzu SSP.α-gliadīna gēni kviešos un cieši radniecīgās sugās attīstījās tikai nesen pēc kviešu atšķiršanās no rudziem.
6. Transkripcijas faktora (TF) un slimību rezistences gēnu izpēte
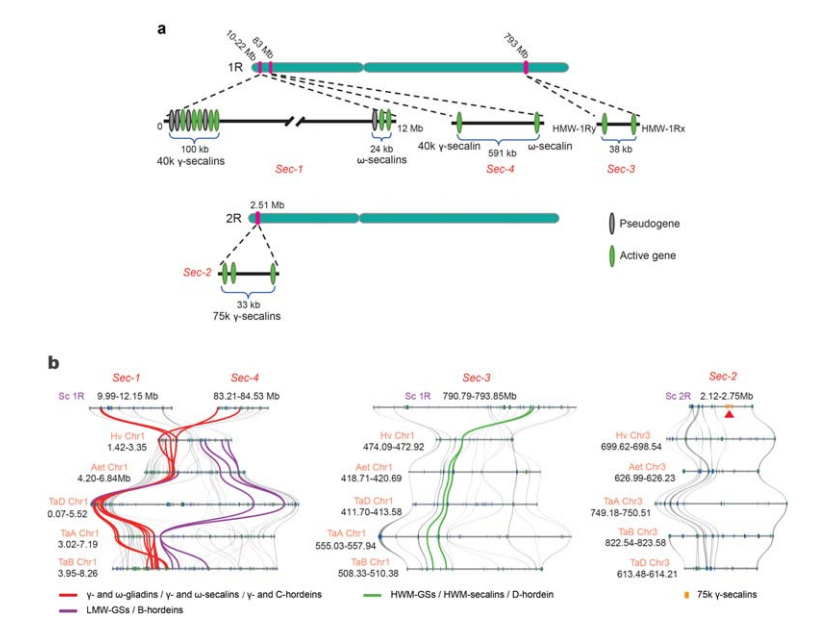
Rudzu sekalīna lokusu analīze
Weining rudziem bija vairāk ar slimību rezistenci saistītu (DRA) gēnu (1989, 3. papildu dati) nekā Tu (1621), Aet (1758), Hv (1508), Bd (1178), Os (1575) un A (1836). ), B (1 728) un D (1 888) mīksto kviešu subgenomi.
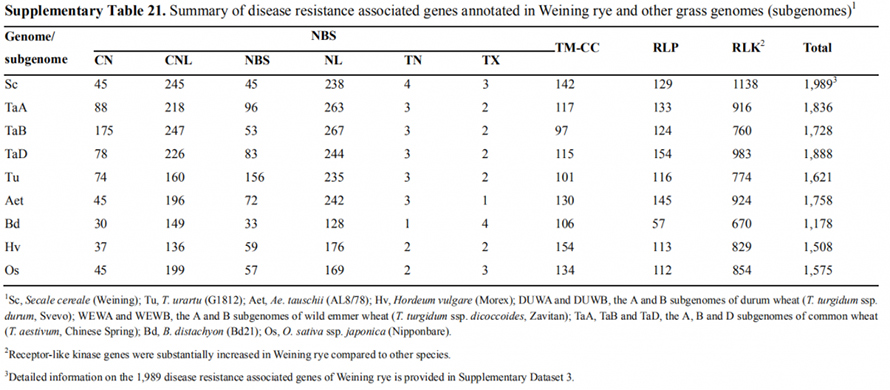
7. Gēnu ekspresijas pazīmju izpēte, kas saistītas ar agrīnu virziena pazīmi
Vaininga genoma montāžā tika anotēti divi FT gēni ar salīdzinoši augstu ekspresiju garas dienas apstākļos, ScFT1 un ScFT2.Tika konstatēta divu ScFT2 (S76 un T132) fosforilācijas aminoskābju atlikumu saistība ar laika kontroles samazināšanos
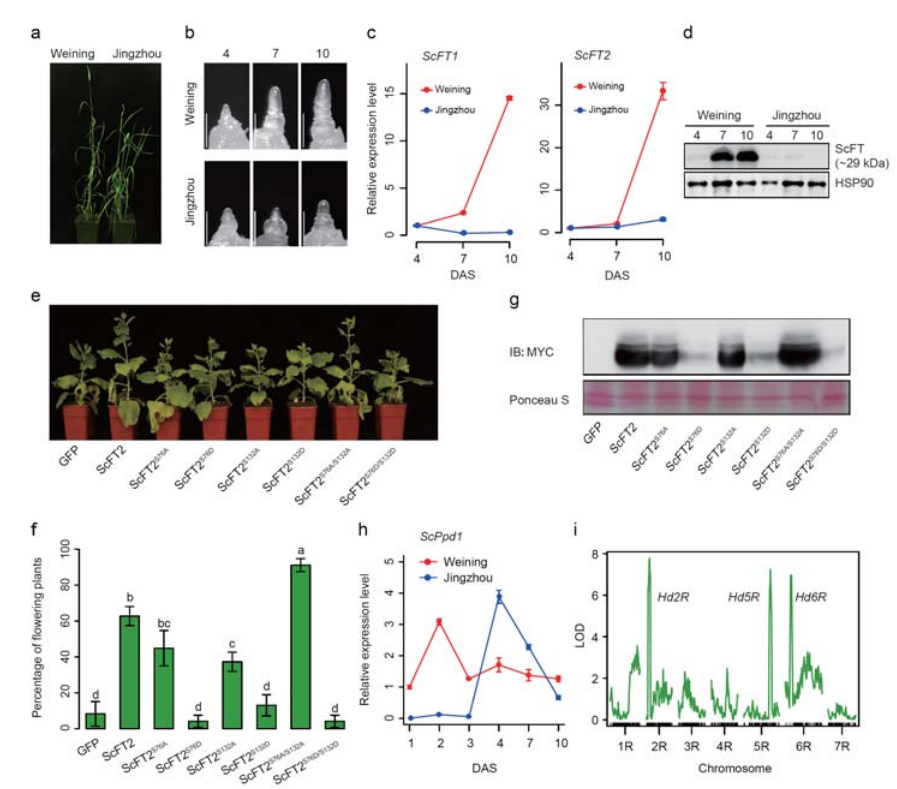
Attīstības un gēnu ekspresijas iezīmes, kas saistītas ar Weining rudzu agrīno virziena iezīmi
8. Rudzu pieradināšanā potenciāli iesaistīto hromosomu reģionu un loku ieguve
Kopā tika izmantoti 123 647 SNP, lai veiktu selektīvu slaucīšanas analīzi starp kultivētajiem rudziem un S. vavilovii.11 selektīvie slaucīšanas signāli, kas identificēti ar samazināšanas indeksu (DRI), fiksācijas indeksu (FST) un XP-CLR metodi.Tika konstatēts, ka ScID1 varētu būt saistīts ar pozīcijas datuma regulēšanu.
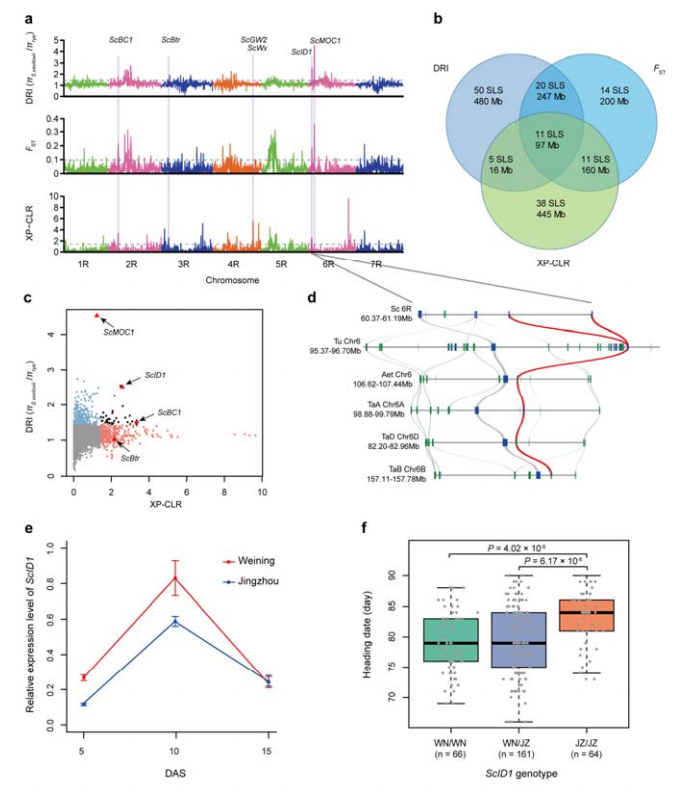
Hromosomu reģionu un loku identificēšana un analīze, kas potenciāli saistīti ar rudzu pieradināšanu
Atsauce
Li GW et al.Augstas kvalitātes genoma komplekts izceļ rudzu genoma īpašības un agronomiski svarīgus gēnus.Dabas ģenētika (2021)
Jaunumi un svarīgākie notikumi mērķis ir dalīties ar jaunākajiem veiksmīgajiem gadījumiem ar Biomarker Technologies, tverot jaunus zinātnes sasniegumus, kā arī ievērojamas metodes, kas izmantotas pētījuma laikā.
Izlikšanas laiks: Jan-05-2022

