EVOLUZIONE DEL GENOMA
genetica della natura
Un assemblaggio del genoma di alta qualità evidenzia le caratteristiche genomiche della segale e i geni agronomicamente importanti
PacBio |Illumina |Mappa ottica di Bionano |Assemblaggio del genoma Hi-C |Mappa genetica |Spazzate selettive |Sequenza RNA |ISO-seq |SLAF-seq
Biomarker Technologies ha fornito supporto tecnico sul sequenziamento Pacbio, sul sequenziamento Hi-C e sull'analisi dei dati in questo studio.
Punti salienti
1. È stato ottenuto il primo genoma di segale di alta qualità a livello cromosomico, che ha una dimensione di un singolo cromosoma maggiore di 1 Gb.
2. Rispetto al genoma di Tu, Aet e Hv, nel genoma della segale è stato osservato un recente evento LTR-RT unico, responsabile dell'estensione delle dimensioni del genoma della segale.
3. La divergenza tra segale e grani diploidi è avvenuta dopo la separazione dell'orzo dal grano, con tempi di divergenza per i due eventi pari a circa 9,6 e 15 MYA.
La fosforilazione dei geni FT può controllare il carattere precoce della segale.
4. L'analisi selettiva indica il possibile coinvolgimento di ScID1 nella regolazione della data di intestazione e la sua probabile selezione mediante addomesticamento nella segale
Sfondo
Sfondo
La segale è una preziosa coltura alimentare e foraggera, un'importante risorsa genetica per il miglioramento del grano e del triticale e un materiale indispensabile per efficienti studi genomici comparativi sulle graminacee.La segale weining, una varietà a fioritura precoce coltivata in Cina, è eccezionale per la sua resistenza ad ampio spettro sia all'oidio che alla ruggine striata.Per comprendere le basi genetiche e molecolari dei tratti d'élite della segale e promuovere gli studi genomici e di selezione nella segale e nelle colture correlate, abbiamo qui sequenziato e analizzato il genoma della segale Weining.
Risultati
Genoma della segale
Il genoma della segale è stato costruito combinando letture PacBio SMRT, sequenziamento Illumina a lettura breve, nonché quelle derivanti dalla cattura della conformazione della cromatina (Hi-C), mappatura genetica e analisi BioNano.I contig assemblati (7,74 Gb) rappresentavano il 98,47% della dimensione stimata del genoma (7,86 Gb), con il 93,67% dei contig (7,25 Gb) assegnato a sette cromosomi.Gli elementi ripetitivi costituivano il 90,31% del genoma assemblato.
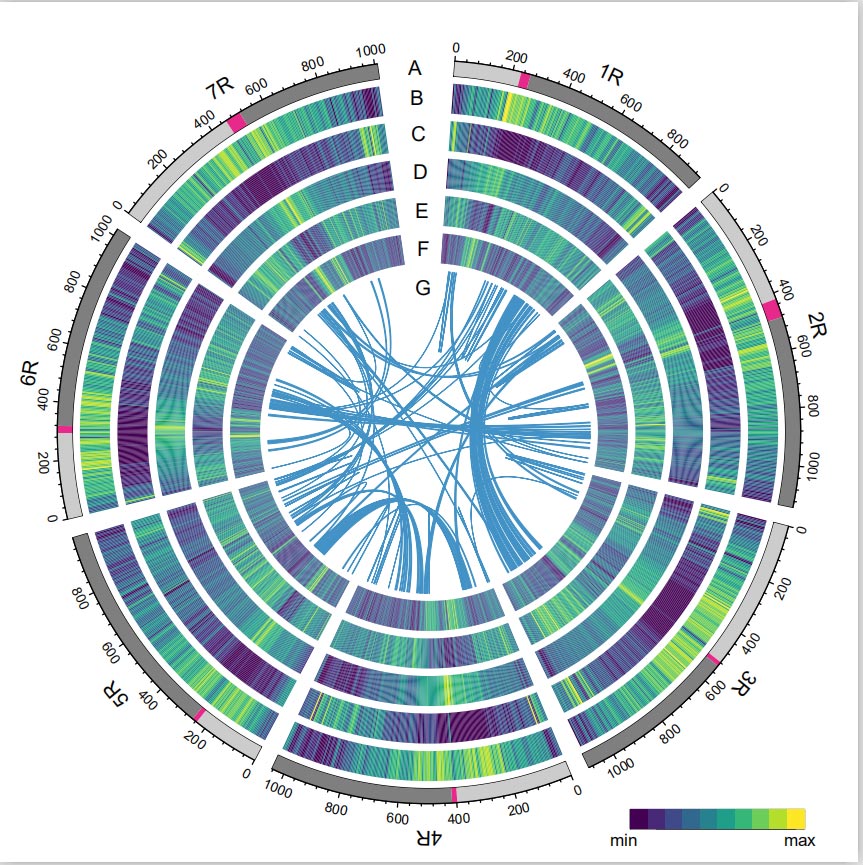
Genoma della segale
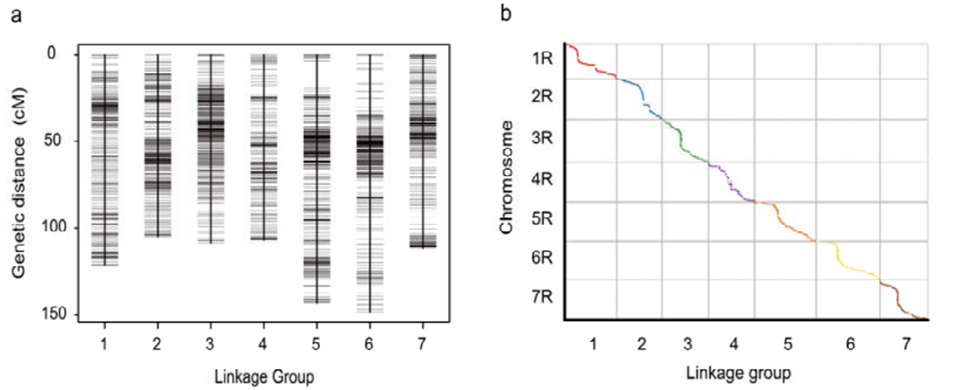
Mappa del collegamento genetico (WJ) sviluppata utilizzando 295 piante F2 derivate dall'incrocio di due varietà autoctone di segale (Weining × Jingzhou)
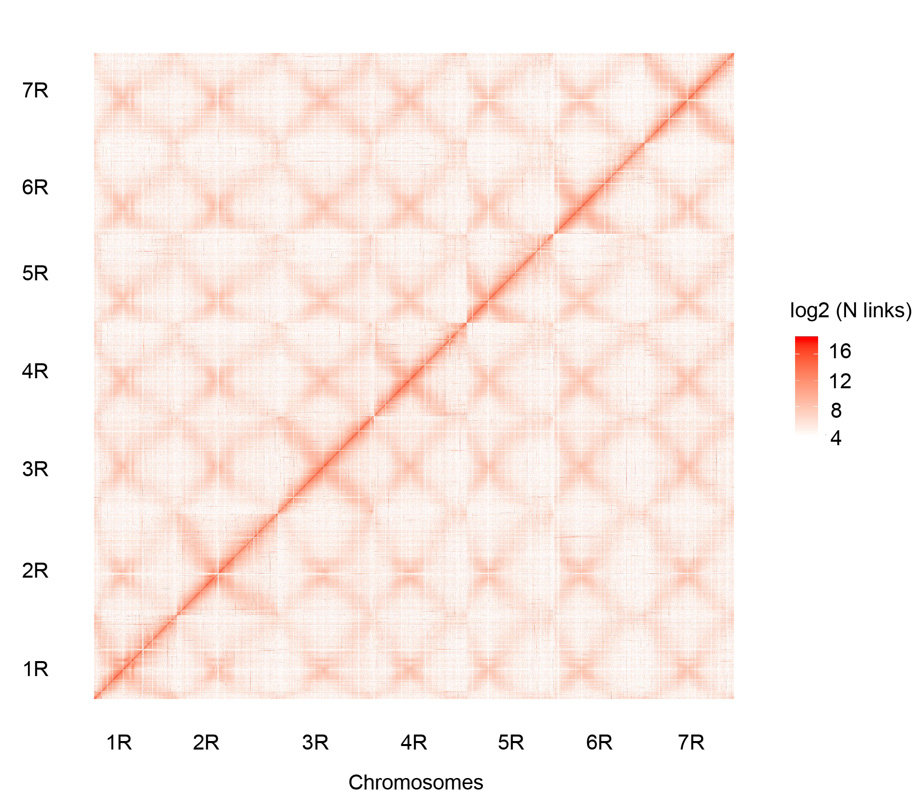
Mappa dei contatti Hi-C dei sette cromosomi di segale Weining assemblati (1R - 7R)
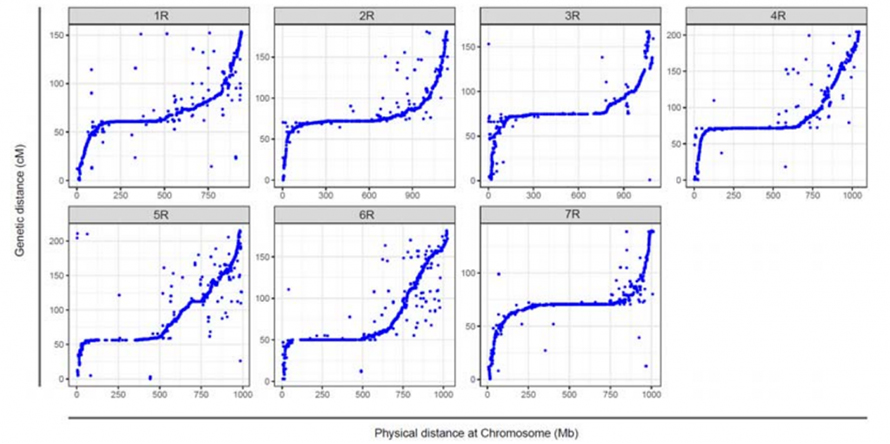
Allineamento tra i sette cromosomi assemblati della segale Weining e i sette gruppi di collegamento della segale sviluppati utilizzando la popolazione RIL Lo7 x Lo255
Il valore dell'indice di assemblaggio LTR (LAI) del genoma della segale è risultato pari a 18,42 e sono stati identificati 1.393 (96,74%) dei 1.440 geni BUSCO altamente conservati. Questi risultati suggeriscono che la sequenza del genoma della segale Weining è di alta qualità sia in e regioni geniche.Sono stati previsti un totale di 86.991 geni codificanti proteine, inclusi 45.596 geni ad alta confidenza (HC) e 41.395 geni a bassa confidenza (LC).
2. Analisi degli TE
Analisi dei TE.Un totale di 6,99 Gb, che rappresentano il 90,31% dell'insieme Weining, sono stati annotati come TE, che comprendevano 2.671.941 elementi appartenenti a 537 famiglie.Questo contenuto di TE era chiaramente superiore a quello precedentemente riportato per Ta (84,70%), Tu (81,42%), Aet (84,40%), WEW (82,20%) o Hv (80,80%).I retrotrasposoni a ripetizione terminale lunga (LTR-RT), inclusi Gypsy, Copia ed elementi RT non classificati, erano i TE dominanti e occupavano l'84,49% del contenuto TE annotato e il 76,29% del genoma di Weining assemblato;I trasposoni del DNA CACTA erano i secondi TE più abbondanti, costituendo l'11,68% del contenuto di TE annotato e il 10,55% del genoma di Weining assemblato.
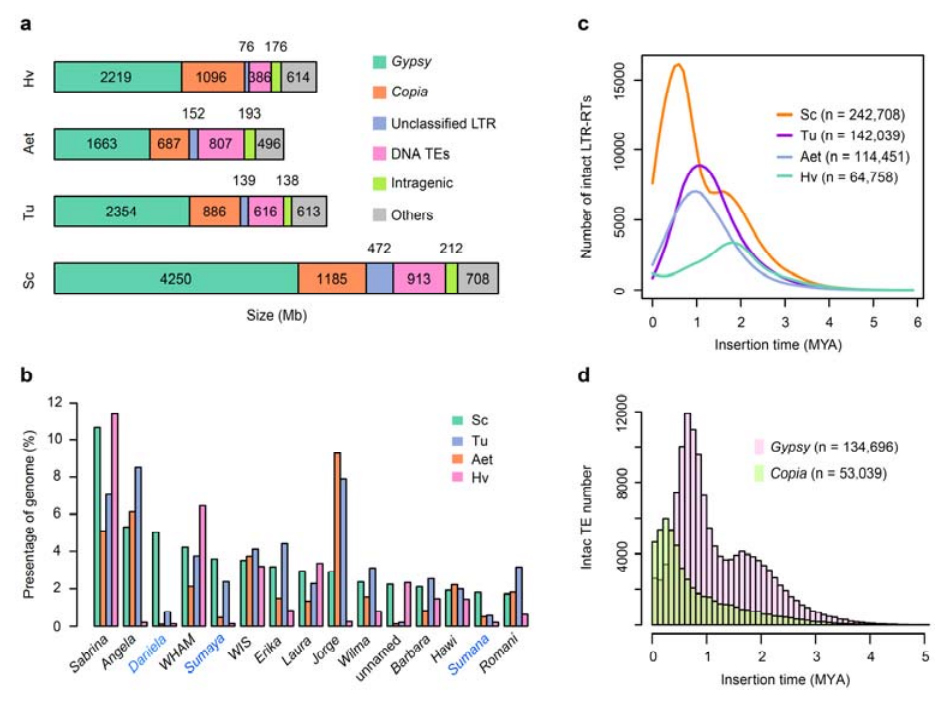
Analisi degli elementi trasposoni della segale
La segale Weining aveva una percentuale relativamente alta di inserimenti recenti di LTR-RT con il picco di amplificazione apparso circa 0,5 milioni di anni fa (MYA), che era il più recente tra le quattro specie;l'altro picco, avvenuto circa 1,7 MYA, era più vecchio e osservato anche nell'orzo.A livello di superfamiglia, sono state trovate esplosioni molto recenti di elementi Copia nella segale Weining a 0,3 MYA, mentre le amplificazioni degli RT zingari hanno modellato in modo dominante il modello di distribuzione bimodale delle dinamiche delle esplosioni LTR-RT.
3. Studio dell'evoluzione del genoma della segale e delle sintenie cromosomiche
La divergenza tra segale e frumenti diploidi ha avuto luogo dopo la separazione dell'orzo dal grano, con tempi di divergenza per i due eventi rispettivamente di circa 9,6 e 15 MYA.1R, 2R, 3R erano interamente collineari rispettivamente con i cromosomi dei gruppi 1, 2 e 3 del grano.4R, 5R, 6R, 7R sono stati trovati esistono fusioni e segmenti su larga scala.
4. Analisi delle duplicazioni geniche e del loro impatto sui geni della biosintesi dell'amido
In particolare, il numero di geni duplicati in tandem (TDG) e di geni duplicati prossimalmente (PDG) della segale Weining era entrambi superiore a quelli trovati per Tu, Aet, Hv, Bd e Os.Anche i geni duplicati trasposti (TrDG) erano più numerosi di quelli trovati specificamente per Tu e Aet.L'espansione del genoma della segale è accompagnata da un numero maggiore di duplicazioni geniche.L’aumento dei TE burst nella segale potrebbe aver portato a un numero elevato di TrDG.
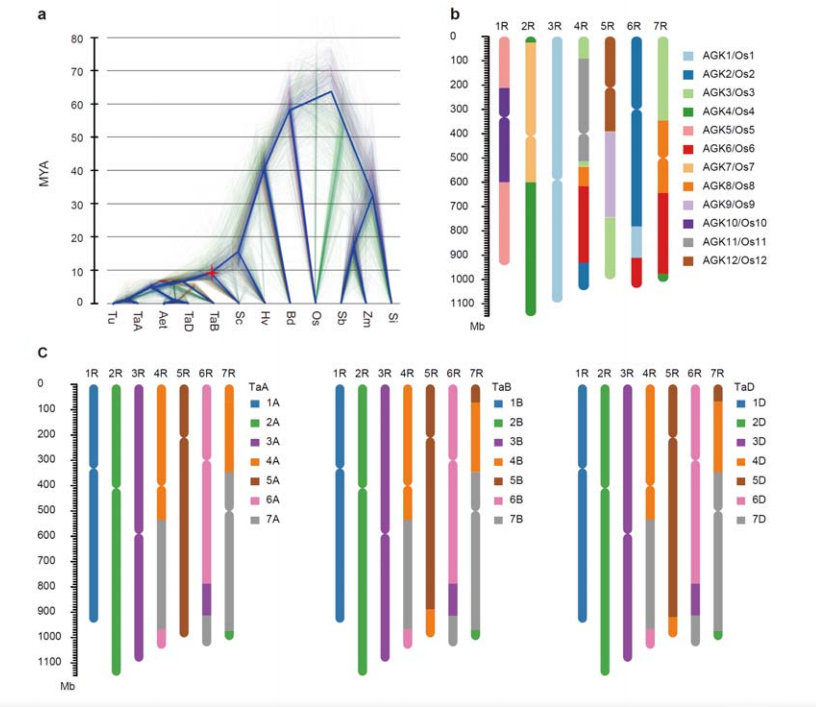
Analisi della sintesi evolutiva e cromosomica del genoma della segale
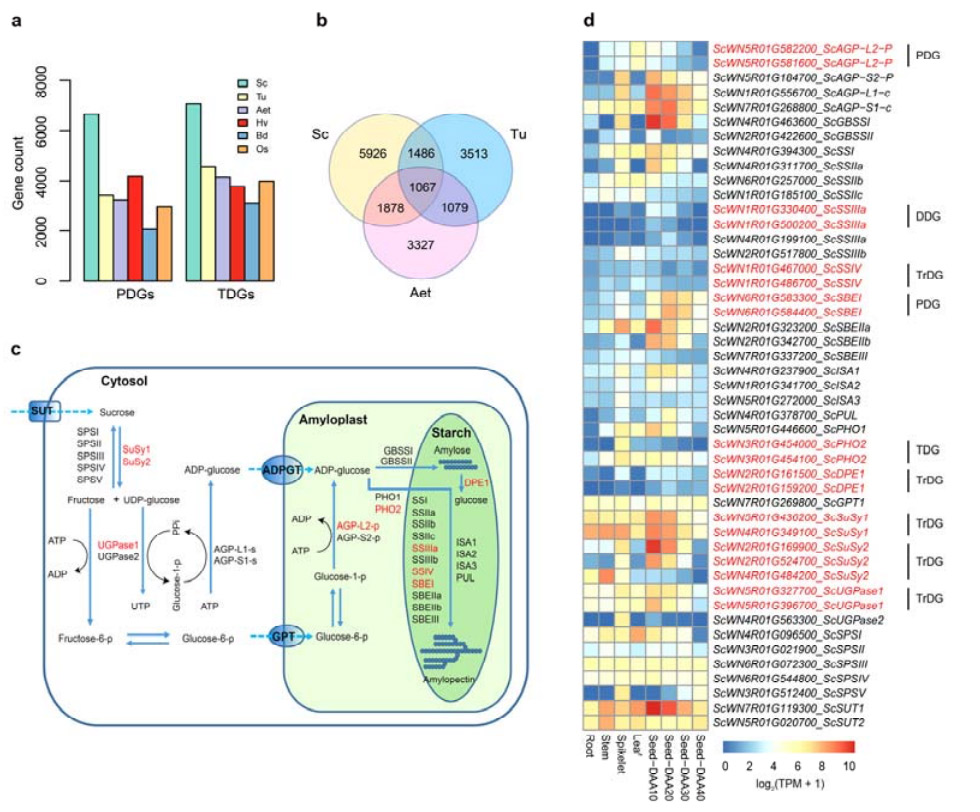
Analisi delle duplicazioni dei geni della segale e del loro impatto sulle diversità dei geni correlati alla biosintesi dell'amido (SBRG)
5. Dissezione dei loci genetici della proteina di stoccaggio dei semi di segale (SSP).
Quattro loci cromosomici (da Sec-1 a Sec-4) che specificano gli SSP della segale sono stati identificati su 1R o 2R.I geni dell’α-gliadina si sono evoluti solo di recente nel grano e in specie strettamente correlate dopo la divergenza del grano dalla segale.
6. Esame del fattore di trascrizione (TF) e dei geni di resistenza alle malattie
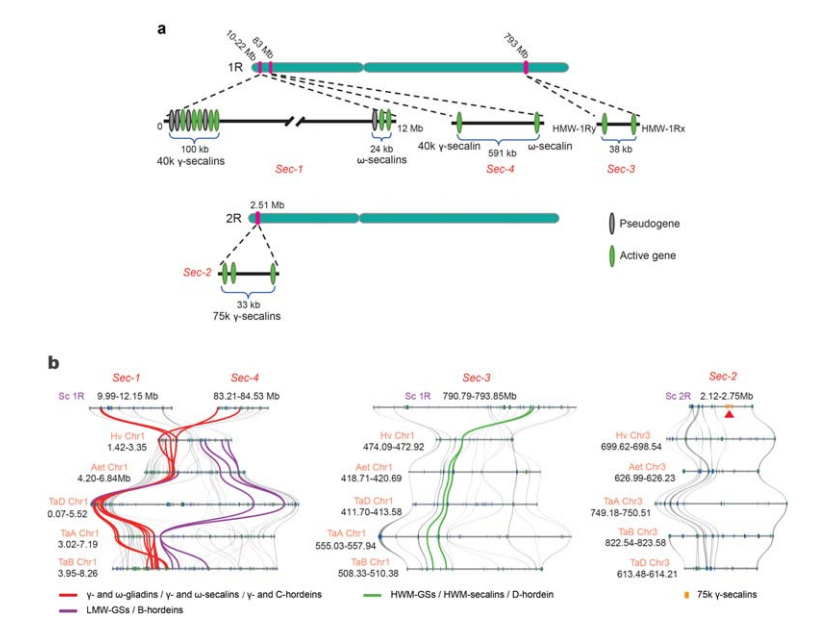
Analisi dei loci delle secaline nella segale
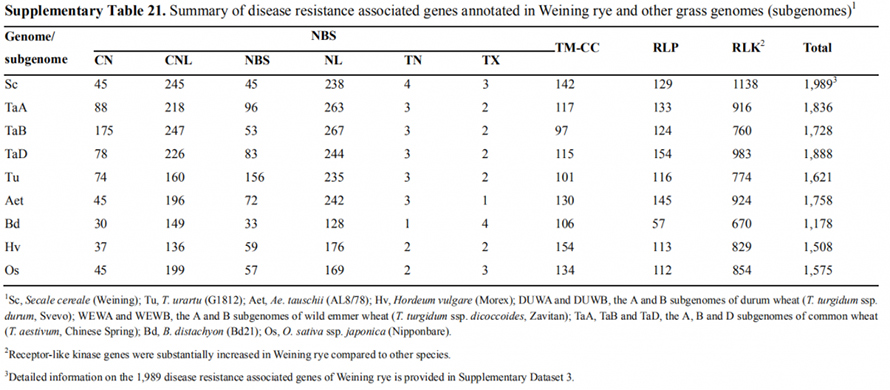
La segale Weining aveva più numerosi geni associati alla resistenza alle malattie (DRA) (1.989, dati supplementari 3) rispetto a Tu (1.621), Aet (1.758), Hv (1.508), Bd (1.178), Os (1.575) e A (1.836 ), sottogenomi B (1.728) e D (1.888) del grano tenero.
7. Studio delle caratteristiche dell'espressione genica associate al tratto precoce della voce
Due geni FT con espressione relativamente alta in condizioni di giornata lunga, ScFT1 e ScFT2, sono stati annotati nell'assemblaggio del genoma di Weining.È stata trovata una relazione con la riduzione del controllo del tempo di due residui aminoacidici della fosforilazione di ScFT2 (S76 e T132)
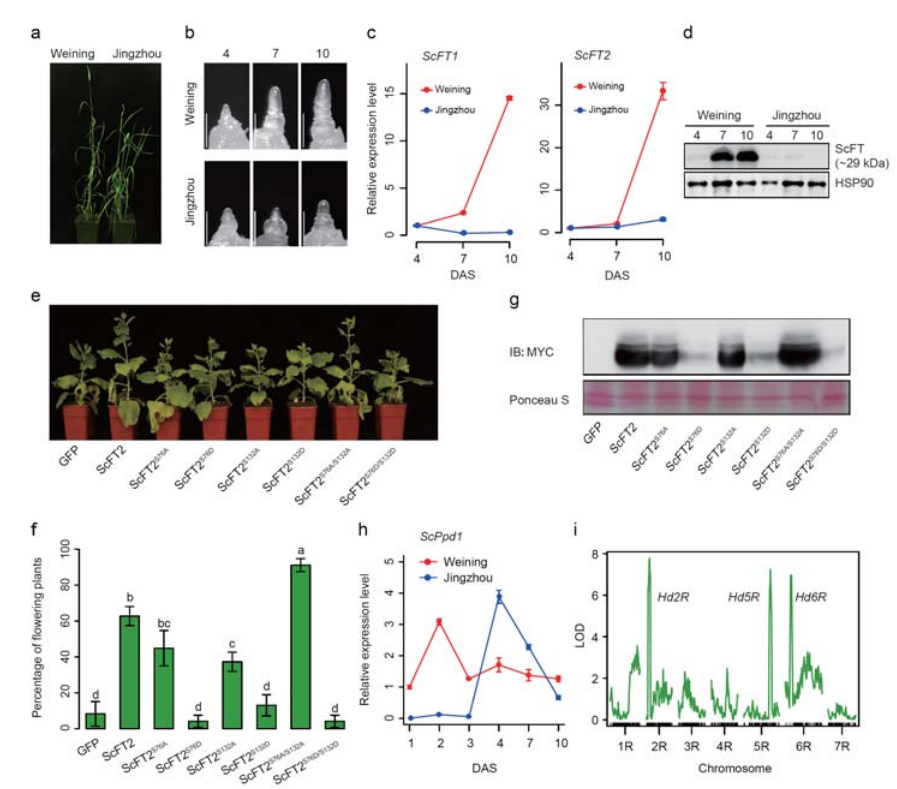
Caratteristiche di sviluppo e di espressione genetica associate al tratto iniziale della segale Weining
8. Estrazione di regioni cromosomiche e loci potenzialmente coinvolti nella domesticazione della segale
Un totale di 123.647 SNP sono stati utilizzati per condurre analisi selettive tra segale coltivata e S. vavilovii.11 segnali di scansione selettivi identificati mediante indice di riduzione (DRI), indice di fissazione (FST) e metodo XP-CLR.È stato riscontrato un possibile coinvolgimento di ScID1 nella regolazione della data di intestazione.
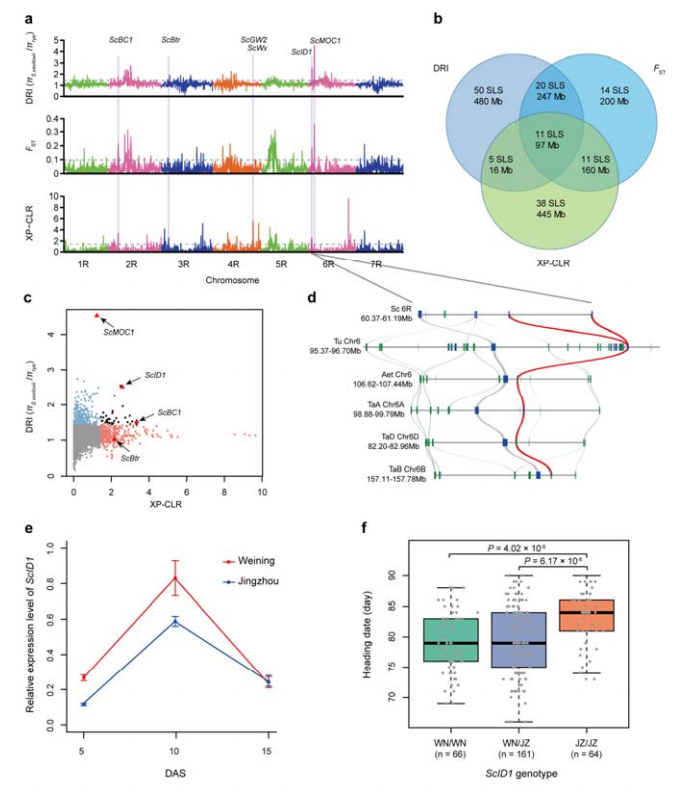
Identificazione e analisi di regioni e loci cromosomici potenzialmente correlati alla domesticazione della segale
Riferimento
Li GW et al.Un assemblaggio del genoma di alta qualità evidenzia le caratteristiche genomiche della segale e i geni agronomicamente importanti.Genetica della natura (2021)
Novità e punti salienti mira a condividere gli ultimi casi di successo con Biomarker Technologies, catturando nuovi risultati scientifici e tecniche importanti applicate durante lo studio.
Orario di pubblicazione: 05-gennaio-2022

