GENOMIEN EVOLUUTIO
luonnon genetiikka
Laadukas genomikokonaisuus korostaa rukiin genomisen ominaisuuksia ja agronomisesti tärkeitä geenejä
PacBio |Illumina |Bionanon optinen kartta |Hi-C Genome Assembly |Geneettinen kartta |Valikoiva pyyhkäisy |RNA-Seq |ISO-seq |SLAF-seq
Biomarker Technologies tarjosi teknistä tukea Pacbio-sekvensoinnille, Hi-C-sekvensoinnille ja data-analyysille tässä tutkimuksessa.
Kohokohdat
1. Saatiin ensimmäinen kromosomitason korkealaatuinen ruisgenomi, jonka yhden kromosomin koko on suurempi kuin 1 Gb.
2. Verrattuna Tu-, Aet- ja Hv-genomiin, rukiin genomissa havaittiin ainutlaatuinen viimeaikainen LTR-RT-tapahtuma, joka oli vastuussa rukiin genomin koon pidentämisestä.
3. Rukiin ja diploidisen vehnän välinen ero tapahtui ohran ja vehnän erottamisen jälkeen, jolloin näiden kahden tapahtuman eroajat olivat noin 9,6 ja 15 MYA.
FT-geenien fosforylaatio voi hallita rukiin varhaista suuntausominaisuutta.
4. Valikoiva pyyhkäisyanalyysi osoittaa ScID1:n mahdollisen osallisuuden nimikepäivämäärän säätelyssä ja sen todennäköisen valinnan kesyttämällä rukiissa
Tausta
Tausta
Ruis on arvokas ravinto- ja rehukasvi, tärkeä geneettinen resurssi vehnän ja ruisvehnän parantamisessa sekä korvaamaton materiaali heinäkasvien tehokkaissa vertailevissa genomiikkatutkimuksissa.Kiinassa viljelty varhain kukkiva Weining-ruislajike on erinomainen laajakirjoises- ti sekä härmäsientä että raidaruostetta vastaan.Ymmärtääksemme rukiin eliittiominaisuuksien geneettistä ja molekyyliperustaa sekä edistääksemme rukiin ja siihen liittyvien viljelykasvien genomi- ja jalostustutkimuksia sekvensoimme ja analysoimme Weining-rukiin genomin.
Saavutukset
Ruis genomi
Ruisgenomi rakennettiin yhdistämällä PacBio SMRT -lukemat, lyhytlukuinen Illumina-sekvensointi sekä kromatiinikonformaation sieppaamisesta (Hi-C), geneettisestä kartoituksesta ja BioNano-analyysistä saadut tulokset.Kokoonpanot (7,74 Gb) edustivat 98,47 % arvioidusta genomin koosta (7,86 Gb), ja 93,67 % jatkuvista (7,25 Gb) oli osoitettu seitsemään kromosomiin.Toistuvat elementit muodostivat 90,31 % kootusta genomista.
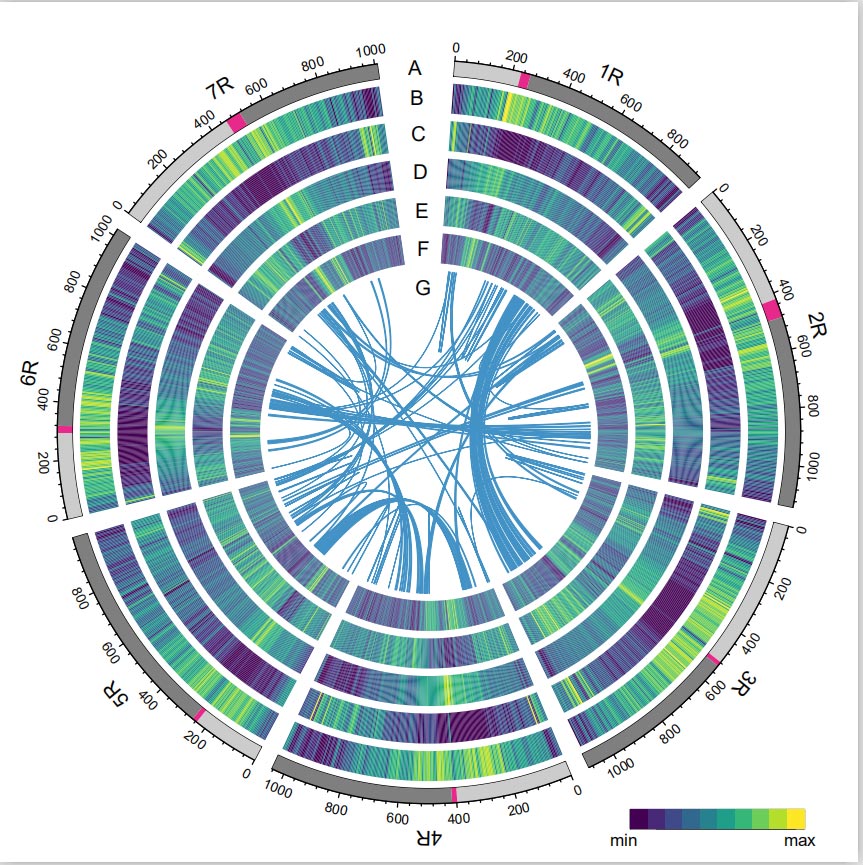
Ruis genomi
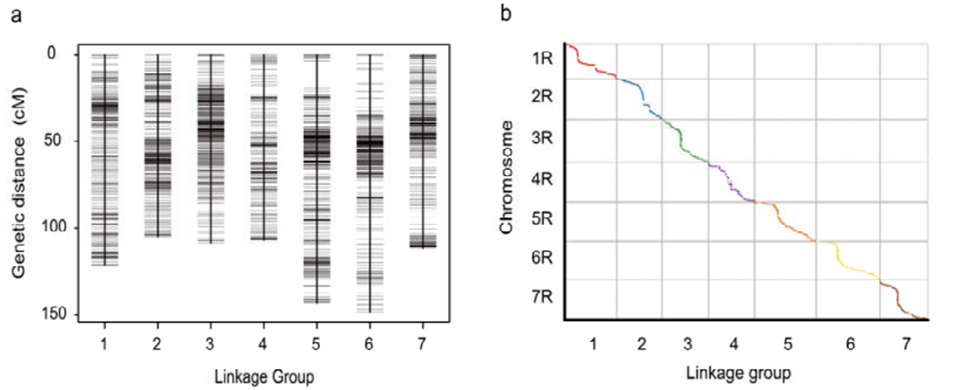
Geneettinen linkityskartta (WJ), joka on kehitetty käyttämällä 295 F2-kasvia, jotka on saatu kahden ruismaalajin (Weining × Jingzhou) risteyttämisestä
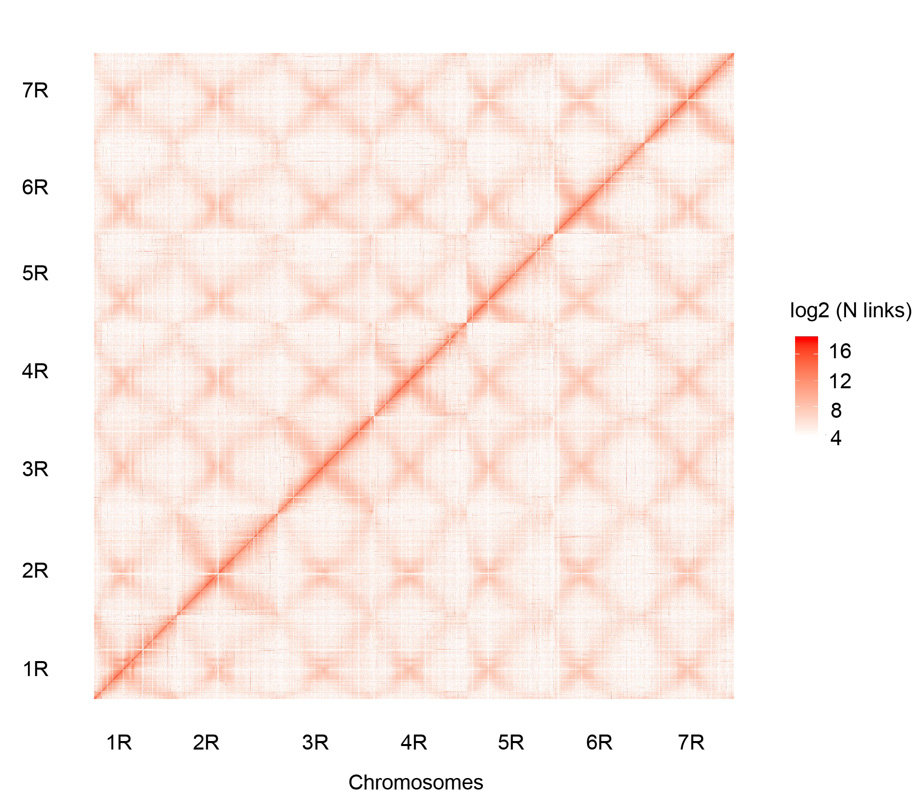
Hi-C-kontaktikartta seitsemästä kootusta Weiningin ruiskromosomista (1R – 7R)
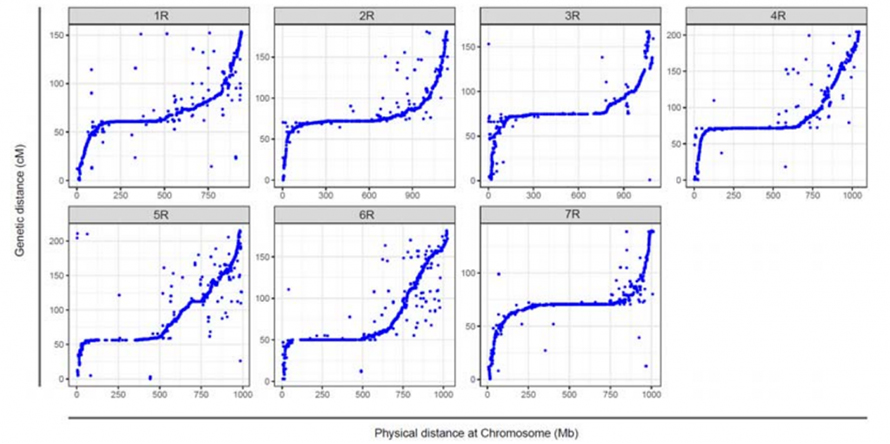
Kohdistus Weiningin rukiin seitsemän kootun kromosomin ja seitsemän ruissidosryhmän välillä, jotka on kehitetty käyttämällä Lo7 x Lo255 RIL-populaatiota
Rukiin genomin LTR Assembly Index (LAI) -arvon havaittiin olevan 18,42 ja 1 393 (96,74 %) 1 440 erittäin konservoituneesta BUSCO-geenistä tunnistettiin. Nämä tulokset viittaavat siihen, että Weiningin rukiin genomisekvenssi on korkealaatuinen molemmissa intergeenisissä geenissä. ja geenialueet.Yhteensä ennustettiin 86 991 proteiinia koodaavaa geeniä, mukaan lukien 45 596 korkean luotettavuuden (HC) geeniä ja 41 395 matalan luottamusgeenin (LC) geeniä.
2. TE:iden analyysi
TE:iden analyysi.Yhteensä 6,99 Gb, mikä edustaa 90,31 % Weiningin kokoonpanosta, merkittiin TE:iksi, jotka sisälsivät 2 671 941 elementtiä, jotka kuuluivat 537 perheeseen.Tämä TE-pitoisuus oli selvästi korkeampi kuin aiemmin raportoitu Ta (84,70 %), Tu (81,42 %), Aet (84,40 %), WEW (82,20 %) tai Hv (80,80 %).Pitkät terminaaliset toistuvat retrotransposonit (LTR-RT), mukaan lukien Gypsy-, Copia- ja luokittelemattomat RT-elementit, olivat hallitsevia TE:itä, ja ne valtasivat 84,49 % annotoidusta TE-sisällöstä ja 76,29 % kootusta Weining-genomista;CACTA DNA:n transposonit olivat toiseksi runsaimmat TE:t, jotka muodostivat 11,68 % annotoidusta TE-sisällöstä ja 10,55 % kootusta Weiningin genomista.
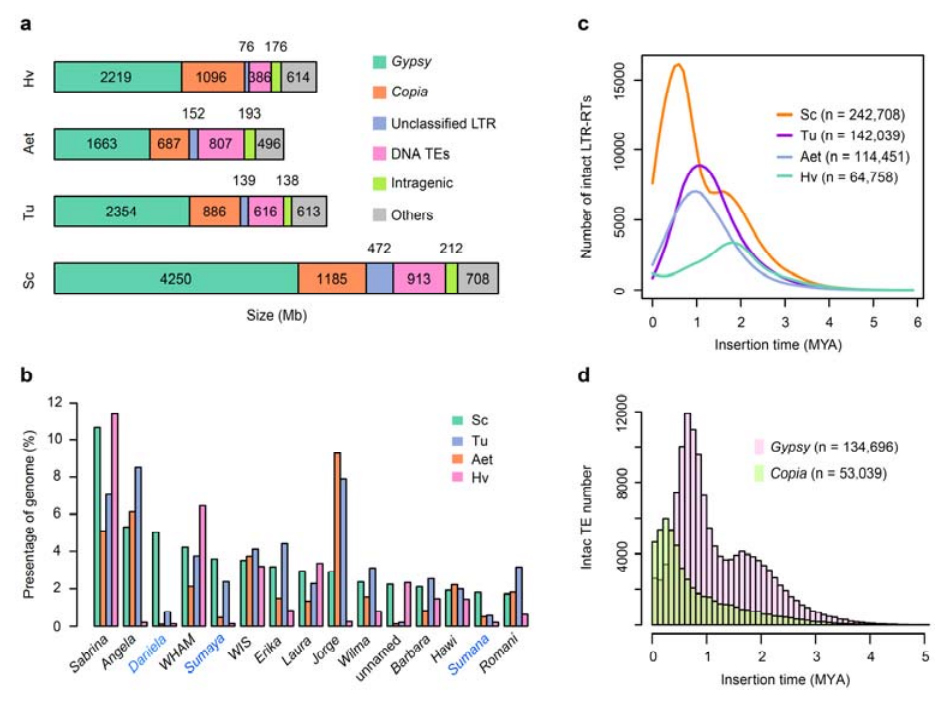
Rukiin transposonielementtien analyysi
Weining-rukiissa oli suhteellisen suuri osuus äskettäin lisätyistä LTR-RT:istä, jolloin vahvistumishuippu ilmestyi noin 0,5 miljoonaa vuotta sitten (MYA), joka oli viimeisin neljästä lajista;toinen huippu, joka oli noin 1,7 MYA, oli vanhempi ja nähtiin myös ohrassa.Superperheen tasolla löydettiin aivan äskettäisiä Copia-elementtien purkauksia Weining-rukiista 0,3 MYA:ssa, kun taas Gypsy RT:iden vahvistukset muokkasivat hallitsevasti LTR-RT-purskedynamiikan bimodaalista jakautumismallia.
3. Rukiin genomin evoluution ja kromosomisynteenien tutkiminen
Ero rukiin ja diploidisen vehnän välillä tapahtui ohran erottamisen jälkeen vehnästä, jolloin näiden kahden tapahtuman eroajat olivat vastaavasti noin 9,6 ja 15 MYA.1R, 2R, 3R olivat täysin kollineaarisia vehnän ryhmien 1, 2 ja 3 kromosomien kanssa, vastaavasti.4R, 5R, 6R, 7R havaittiin, että on olemassa laajamittaisia fuusioita ja segmenttejä.
4. Analyysi geenien päällekkäisyyksistä ja niiden vaikutuksista tärkkelyksen biosynteesigeeneihin
Erityisesti Weiningin rukiin tandemly duplikoituneiden geenien (TDG) ja proksimaalisesti monistuneiden geenien (PDG:t) määrät olivat molemmat korkeammat kuin Tu, Aet, Hv, Bd ja Os.Transponoituja päällekkäisiä geenejä (TrDG:t) oli myös enemmän kuin niitä, jotka löydettiin erityisesti Tulle ja Aetille.Rukiin genomin laajentumiseen liittyy suurempi määrä geenien päällekkäisyyksiä.Rukiin lisääntyneet TE-purskeet ovat saattaneet johtaa kohonneeseen TrDG:iden määrään.
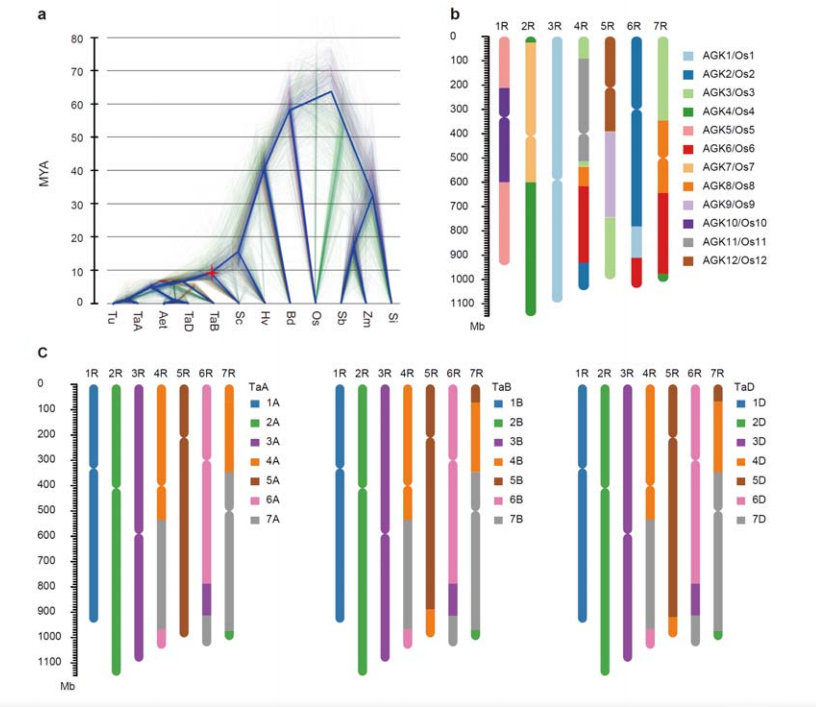
Rukiin genomin evoluutio- ja kromosomisynteenia-analyysit
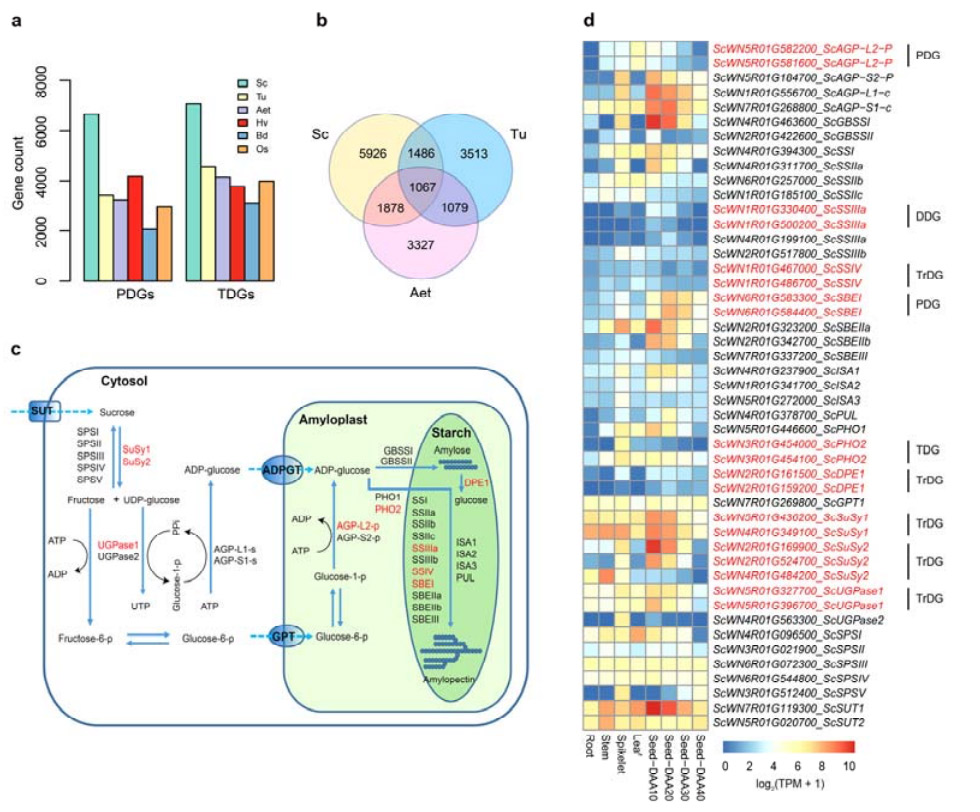
Analyysi rukiin geenien päällekkäisyydestä ja niiden vaikutuksesta tärkkelyksen biosynteesiin liittyvien geenien (SBRG:iden) monimuotoisuuteen
5. Rukiin siementen varastoproteiinin (SSP) geenilokusten dissektio
1R:stä tai 2R:stä on tunnistettu neljä kromosomaalista lokusta (Sec-1 - Sec-4), jotka määrittelevät rukiin SSP:t.α-gliadiinigeenit kehittyivät vasta äskettäin vehnässä ja lähisukulaislajeissa vehnän ja rukiin erottua.
6. Transkriptiotekijän (TF) ja tautiresistenssigeenien tutkiminen
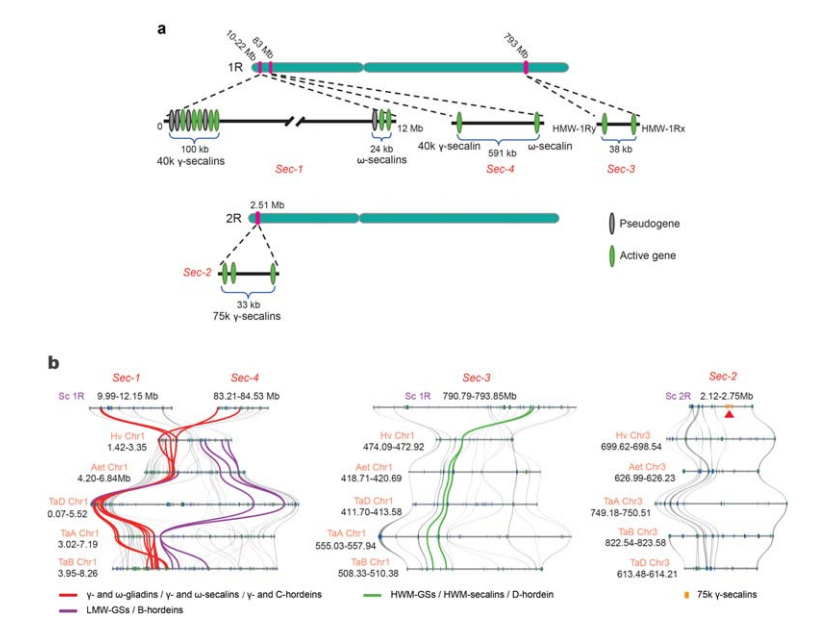
Rukiin sekaliinilokusten analyysi
Weining-rukiissa oli enemmän DRA-geenejä (1 989, lisätiedot 3) kuin Tu (1 621), Aet (1 758), Hv (1 508), Bd (1 178), Os (1 575) ja A (1 836) tavallisen vehnän alagenomit B (1 728) ja D (1 888).
7. Varhaiseen suuntausominaisuuteen liittyvien geenien ilmentymisominaisuuksien tutkiminen
Kaksi FT-geeniä, joilla oli suhteellisen korkea ekspressio pitkän päivän olosuhteissa, ScFT1 ja ScFT2, merkittiin Weiningin genomikokoonpanoon.ScFT2:n (S76 ja T132) fosforylaation kahdella aminohappotähteellä havaittiin yhteys alentavan aikakontrollin kanssa
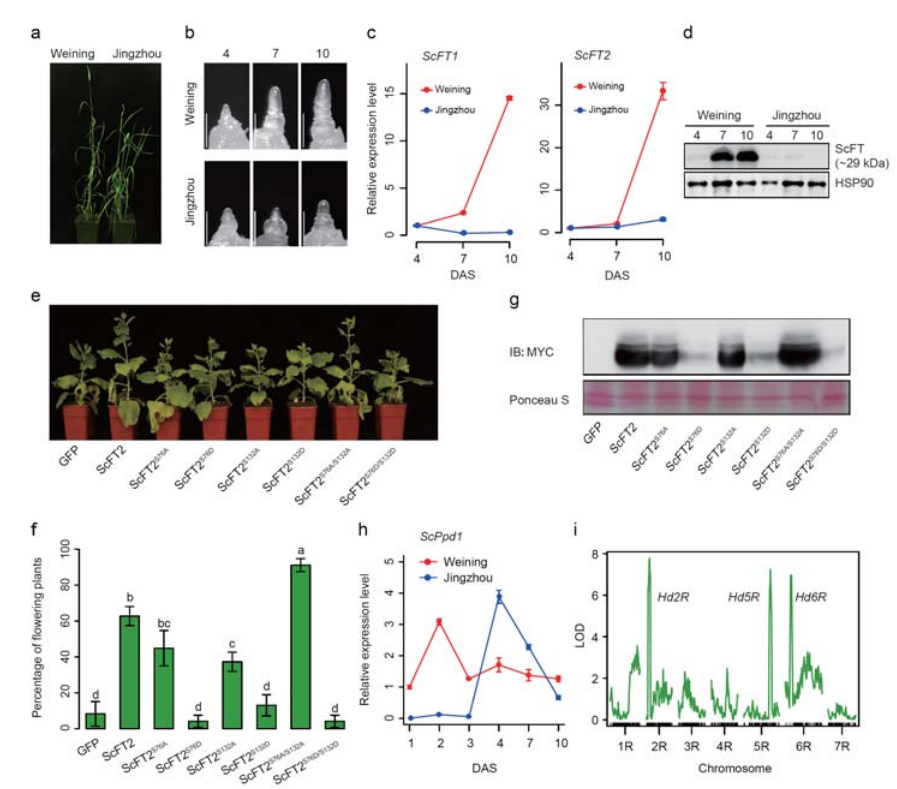
Kehitys- ja geeniekspressiopiirteet, jotka liittyvät Weining-rukiin varhaiseen otsikkoominaisuuteen
8. Rukiin kesytykseen mahdollisesti liittyvien kromosomialueiden ja lokusten louhinta
Yhteensä 123 647 SNP:tä käytettiin selektiiviseen pyyhkäisyanalyysiin viljellyn rukiin ja S. vaviloviin välillä.11 selektiivistä pyyhkäisysignaalia, jotka tunnistetaan vähennysindeksillä (DRI), kiinnitysindeksillä (FST) ja XP-CLR-menetelmällä.ScID1:llä havaittiin mahdollista osallistumista otsikon päivämäärän säätelyyn.
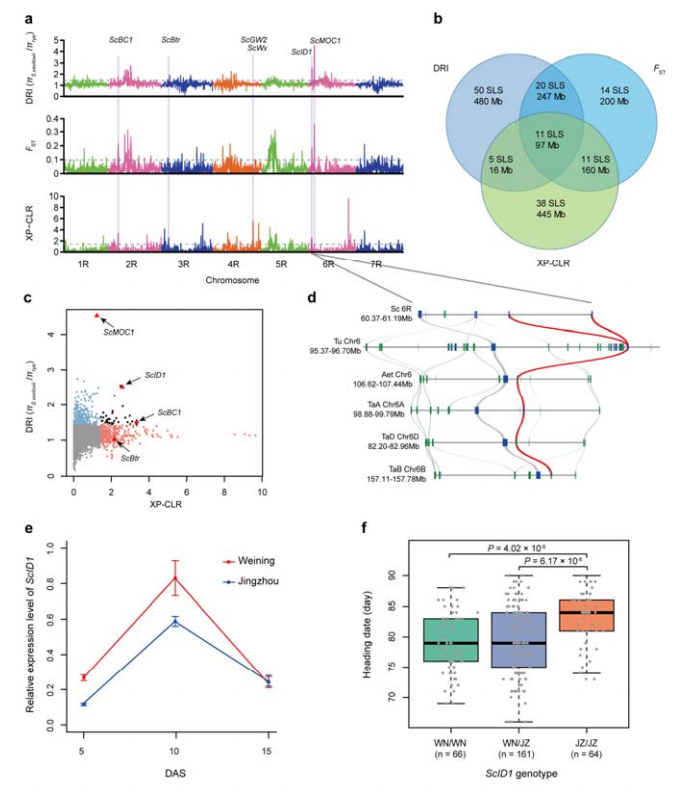
Rukiin kesyttämiseen mahdollisesti liittyvien kromosomialueiden ja lokusten tunnistaminen ja analysointi
Viite
Li GW et ai.Laadukas genomikokonaisuus korostaa rukiin genomiominaisuuksia ja agronomisesti tärkeitä geenejä.Nature Genetics (2021)
Uutisia ja kohokohtia Tavoitteena on jakaa uusimmat onnistuneet tapaukset Biomarker Technologiesin kanssa, vangita uusia tieteellisiä saavutuksia sekä tutkimuksen aikana käytettyjä merkittäviä tekniikoita.
Postitusaika: 05.01.2022

