GENOME EVOLUTION
genetika ng kalikasan
Ang isang mataas na kalidad na genome assembly ay nagha-highlight ng rye genomic na mga katangian at agronomically important genes
PacBio |Illumina |Bionano Optical Map |Hi-C Genome Assembly |Genetic na Mapa |Mga Selective Sweep |RNA-Seq |ISO-seq |SLAF-seq
Ang Biomarker Technologies ay nagbigay ng teknikal na suporta sa Pacbio sequencing, Hi-C sequencing at data analysis sa pag-aaral na ito.
Mga highlight
1. Nakuha ang unang chromosomal-level na mataas na kalidad na Rye genome, na mayroong isang laki ng chromosome na mas malaki sa 1 Gb.
2. Kung ikukumpara sa genome ng Tu, Aet at Hv, isang natatanging kamakailang LTR-RT na kaganapan ang naobserbahan sa Rye genome, na responsable para sa pagpapalawak ng laki ng rye genome.
3. Ang pagkakaiba sa pagitan ng rye at diploid na trigo ay naganap pagkatapos ng paghihiwalay ng barley mula sa trigo, na ang mga oras ng pagkakaiba para sa dalawang kaganapan ay humigit-kumulang 9.6 at 15 MYA .
Maaaring kontrolin ng FT genes phosphorylation ang maagang heading trait sa rye.
4. Ang selective sweep analysis ay nagpapahiwatig ng posibleng pagkakasangkot ng ScID1 sa regulasyon ng petsa ng heading at ang posibleng pagpili nito sa pamamagitan ng domestication sa rye
Background
Background
Ang Rye ay isang mahalagang pagkain at forage crop, isang mahalagang genetic na mapagkukunan para sa pagpapabuti ng trigo at triticale, at isang kailangang-kailangan na materyal para sa mahusay na paghahambing na pag-aaral ng genomics sa mga damo.Ang Weining rye, isang uri ng maagang namumulaklak na nilinang sa China, ay namumukod-tangi dahil sa malawak na spectrum nitong panlaban sa powdery mildew at stripe rust.Upang maunawaan ang genetic at molekular na batayan ng rye elite traits at upang maisulong ang genomic at breeding na pag-aaral sa rye at mga kaugnay na pananim, sini-sequence at sinuri namin dito ang genome ng Weining rye.
Mga nagawa
Rye Genome
Ang Rye genome ay binuo sa pamamagitan ng pagsusuklay ng mga PacBio SMRT reads, maikling-read na pagkakasunud-sunod ng Illumina, pati na rin ang mga mula sa chromatin conformation capture (Hi-C), genetic mapping, at BioNano analysis.Ang mga pinagsama-samang contigs (7.74 Gb) ay nagkakahalaga ng 98.47% ng tinantyang laki ng genome (7.86 Gb), na may 93.67% ng mga contigs (7.25 Gb) na itinalaga sa pitong chromosome.Ang mga paulit-ulit na elemento ay bumubuo ng 90.31% ng pinagsama-samang genome.
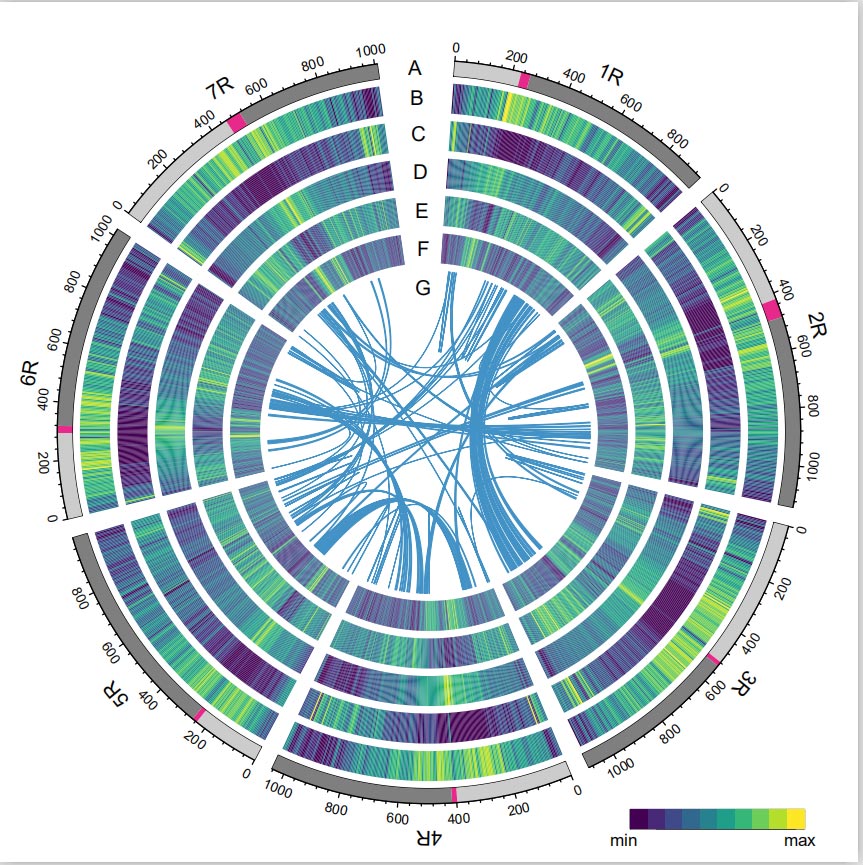
Rye Genome
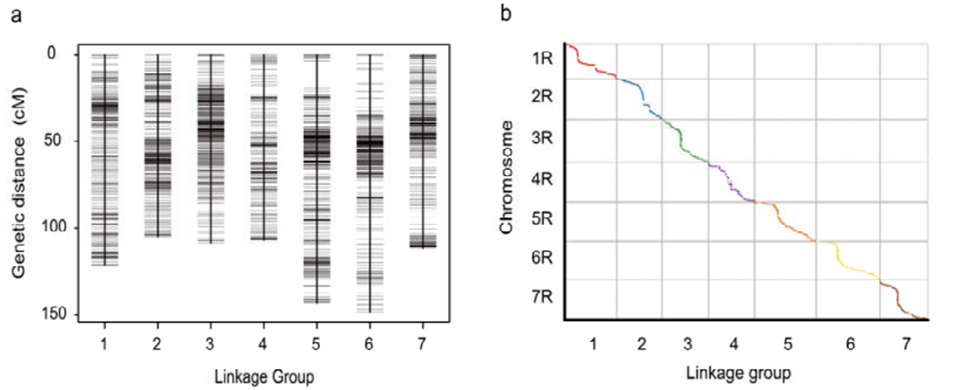
Ang genetic linkage map (WJ) ay binuo gamit ang 295 F2 na halaman na nagmula sa pagtawid ng dalawang rye landraces (Weining × Jingzhou)
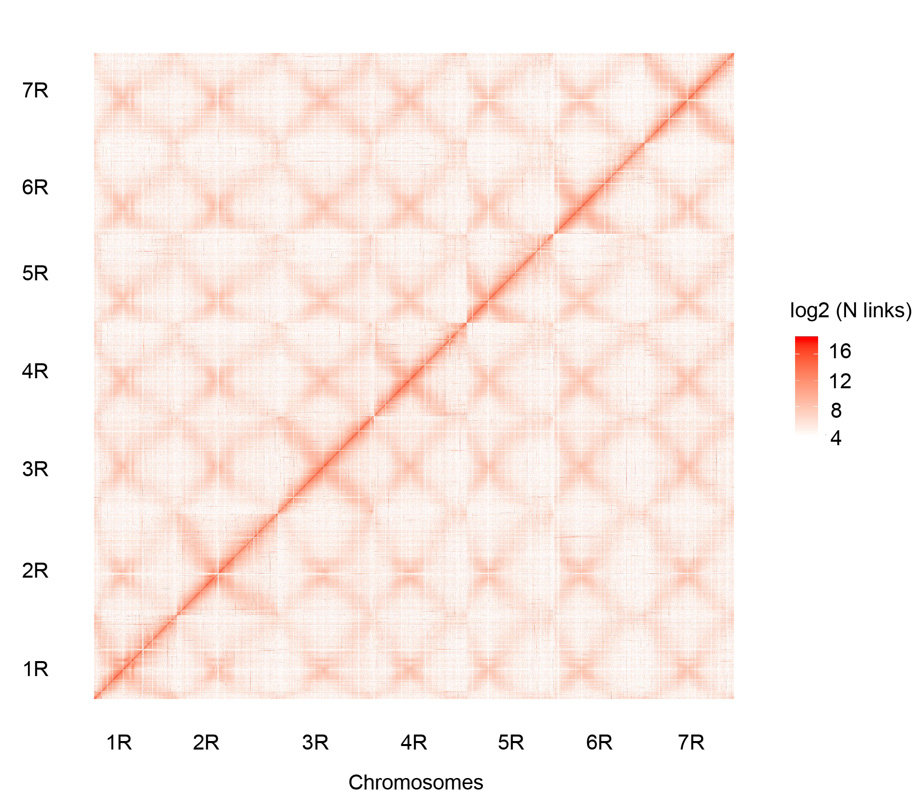
Hi-C contact map ng pitong naka-assemble na Weining rye chromosomes (1R – 7R)
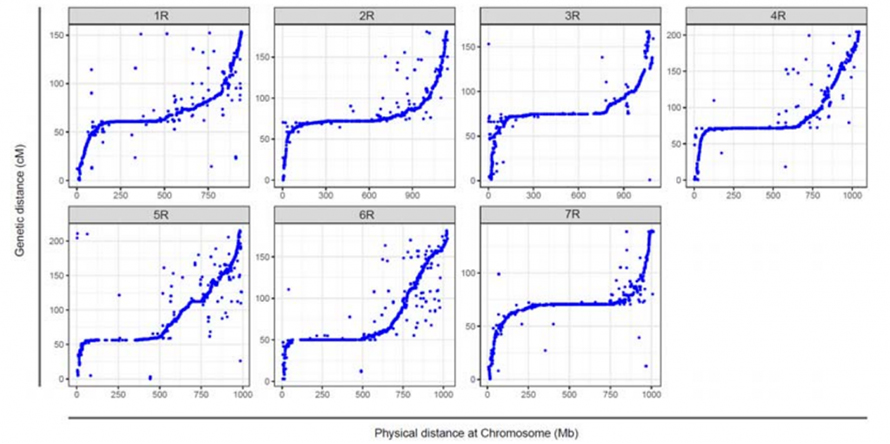
Alignment sa pagitan ng pitong naka-assemble na chromosome ng Weining rye at ng pitong rye linkage group na binuo gamit ang populasyon ng Lo7 x Lo255 RIL
Ang halaga ng LTR Assembly Index (LAI) ng Rye genome ay natagpuang 18.42 at 1,393 (96.74%) ng 1,440 na lubos na napangalagaang BUSCO genes ay natukoy . Iminumungkahi ng mga resultang ito na ang Weining rye genome sequence ay may mataas na kalidad sa parehong intergenic at mga genic na rehiyon.Isang kabuuan ng 86,991 protein-coding genes, kabilang ang 45,596 high-confidence(HC) genes at 41,395 low-confidence genes (LC) genes ang hinulaang.
2. Pagsusuri ng mga TE
Pagsusuri ng mga TE.Isang kabuuan na 6.99 Gb, na kumakatawan sa 90.31% ng Weining assembly, ay na-annotate bilang mga TE, na kinabibilangan ng 2,671,941 elemento na kabilang sa 537 pamilya.Ang nilalamang TE na ito ay malinaw na mas mataas kaysa sa naunang iniulat para sa Ta (84.70%), Tu (81.42%), Aet (84.40%), WEW (82.20%), o Hv (80.80%).Ang mahabang terminal repeat retrotransposons (LTR-RTs), kabilang ang Gypsy, Copia at unclassified RT elements, ay ang nangingibabaw na TEs, at 1ccupied 84.49% ng annotated TE content at 76.29 % ng assembled Weining genome;Ang mga transposon ng CACTA DNA ay ang pangalawang pinaka-masaganang TE, na bumubuo ng 11.68% ng annotated na nilalaman ng TE at 10.55% ng pinagsama-samang Weining genome.
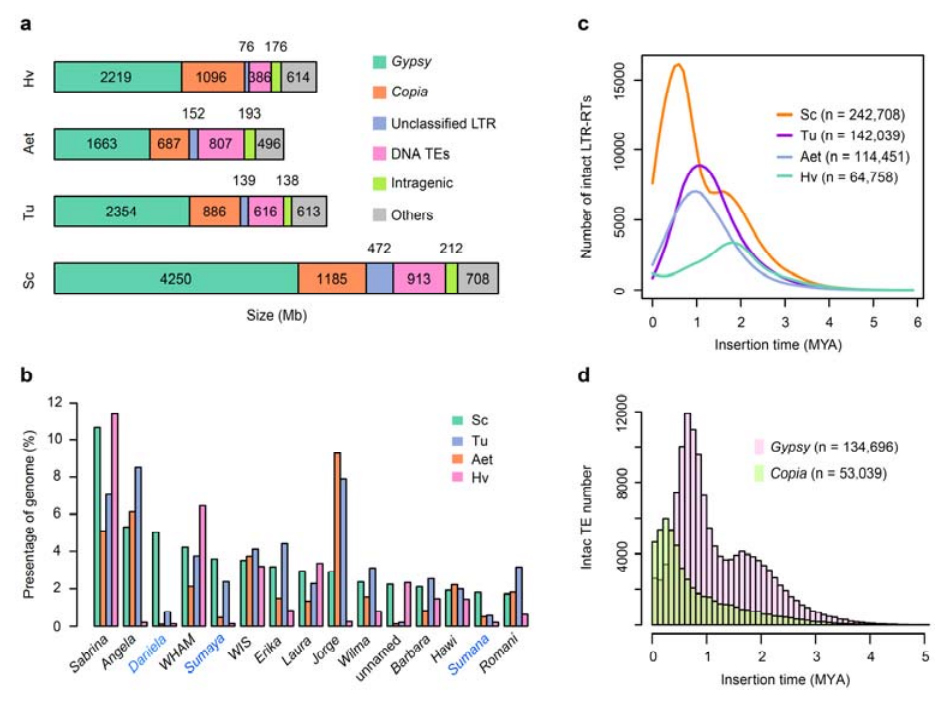
Pagsusuri ng mga elemento ng transposon ng rye
Ang Weining rye ay may medyo mataas na proporsyon ng mga kamakailang pagpapasok ng LTR-RT na may peak of amplification na lumitaw mga 0.5 milyong taon na ang nakalilipas (MYA), na siyang pinakabago sa apat na species;ang iba pang peak, naganap humigit-kumulang 1.7 MYA, ay mas matanda at nakikita rin sa barley.Sa antas ng superfamily, ang pinakahuling pagsabog ng mga elemento ng Copia sa Weining rye sa 0.3 MYA ay natagpuan, habang ang mga amplification ng Gypsy RT ay dominanteng humubog sa bimodal distribution pattern ng LTR-RT burst dynamics.
3. Pagsisiyasat ng rye genome evolution at chromosome syntenies
Ang pagkakaiba sa pagitan ng rye at diploid na trigo ay naganap pagkatapos ng paghihiwalay ng barley mula sa trigo, na ang mga oras ng pagkakaiba para sa dalawang kaganapan ay humigit-kumulang 9.6 at 15 MYA, ayon sa pagkakabanggit.Ang 1R, 2R, 3R ay ganap na collinear sa mga pangkat 1, 2 at 3 chromosome ng trigo, ayon sa pagkakabanggit.4R, 5R, 6R, 7R ay natagpuan na mayroong malakihang pagsasanib at mga segment.
4. Pagsusuri ng mga pagdoble ng gene at ang epekto nito sa mga gene ng starch biosynthesis
Kapansin-pansin, ang mga bilang ng mga tandemly duplicated genes (TDGs) at proximally duplicated genes (PDGs) ng Weining rye ay parehong mas mataas kaysa sa mga natagpuan para sa Tu, Aet, Hv, Bd at Os.Ang mga transposed duplicated genes (TrDGs) ay mas marami rin kaysa sa mga partikular na natagpuan para sa Tu at Aet.Ang pagpapalawak ng genome ng Rye ay sinamahan ng mas mataas na bilang ng mga pagdoble ng gene.Ang tumaas na pagsabog ng TE sa rye ay maaaring humantong sa isang mataas na bilang ng mga TrDG.
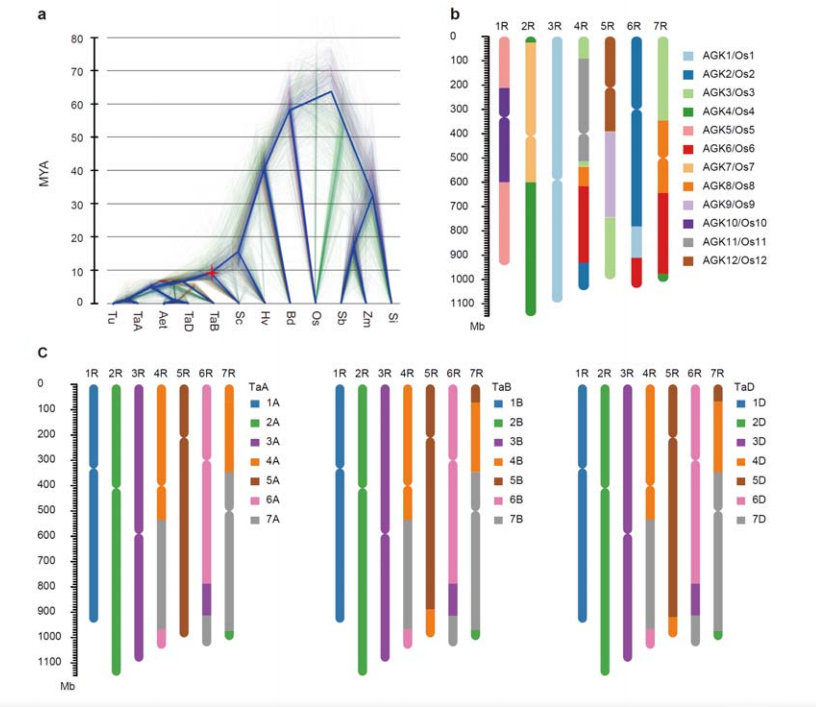
Pagsusuri ng evolutionary at chromosome synteny ng rye genome
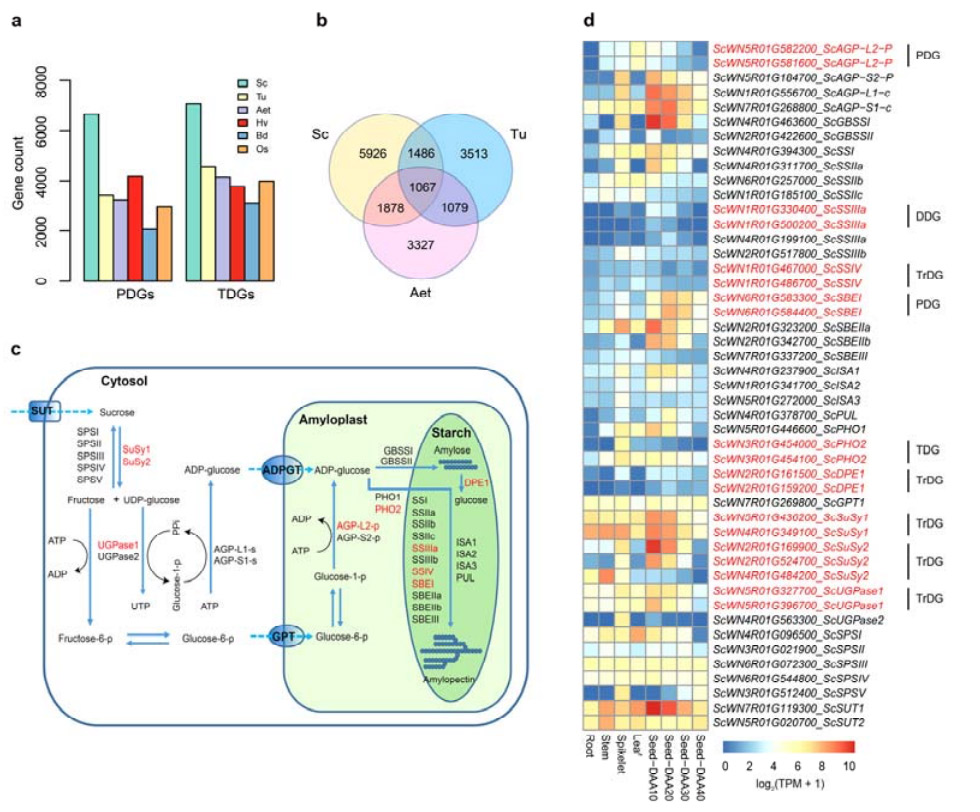
Pagsusuri ng mga duplikasyon ng rye gene at ang epekto nito sa pagkakaiba-iba ng mga starch biosynthesis related genes (SBRGs)
5. Dissection ng rye seed storage protein (SSP) gene loci
Apat na chromosomal loci (Sec-1 hanggang Sec-4) na tumutukoy sa mga rye SSP ay nakilala sa 1R o 2R.Ang mga gene ng α-gliadin ay nag-evolve kamakailan lamang sa trigo at malapit na nauugnay na mga species pagkatapos ng divergence ng trigo mula sa rye.
6. Pagsusuri ng transcription factor (TF) at mga gene na lumalaban sa sakit
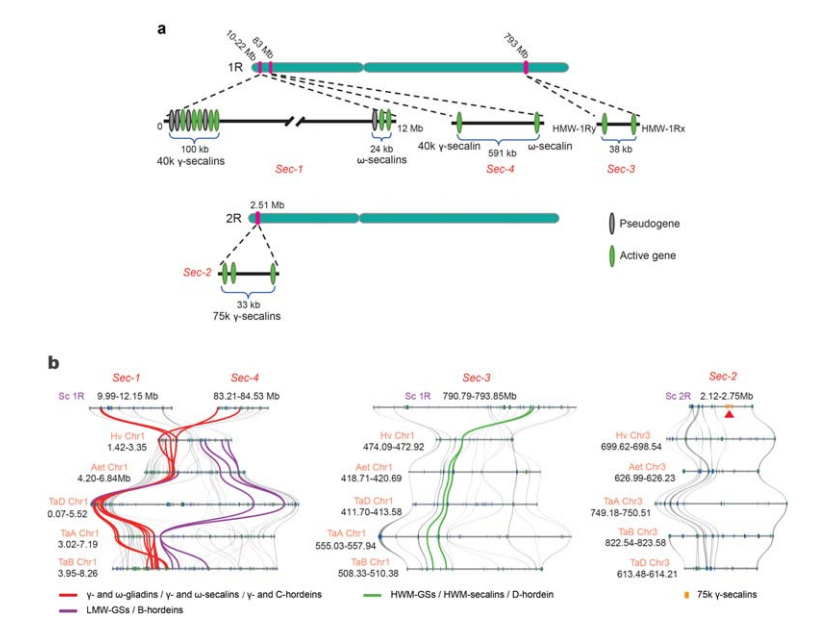
Pagsusuri ng rye secalin loci
Ang Weining rye ay may mas maraming mga gene na nauugnay sa paglaban sa sakit (DRA) (1,989, Karagdagang Data 3) kaysa sa Tu (1,621), Aet (1,758), Hv (1,508), Bd (1,178), Os (1,575), at ang A (1,836). ), B (1,728) at D (1,888) na mga subgenome ng karaniwang trigo.
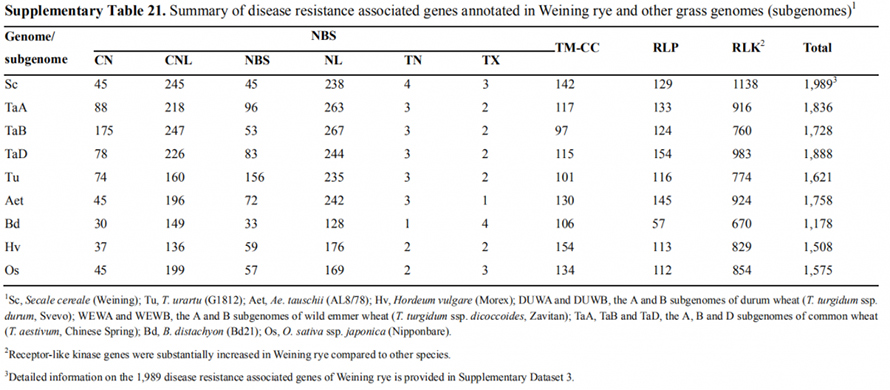
7. Pagsisiyasat ng mga tampok ng pagpapahayag ng gene na nauugnay sa katangian ng maagang heading
Dalawang FT gen na may medyo mataas na pagpapahayag sa ilalim ng mga kondisyon ng mahabang araw, ScFT1 at ScFT2, ay na-annotate sa Weining genome assembly.Dalawang amino acid residues ng ScFT2 (S76 at T132) phosphorylation ang natagpuang kaugnayan sa pagpapababa ng kontrol ng oras
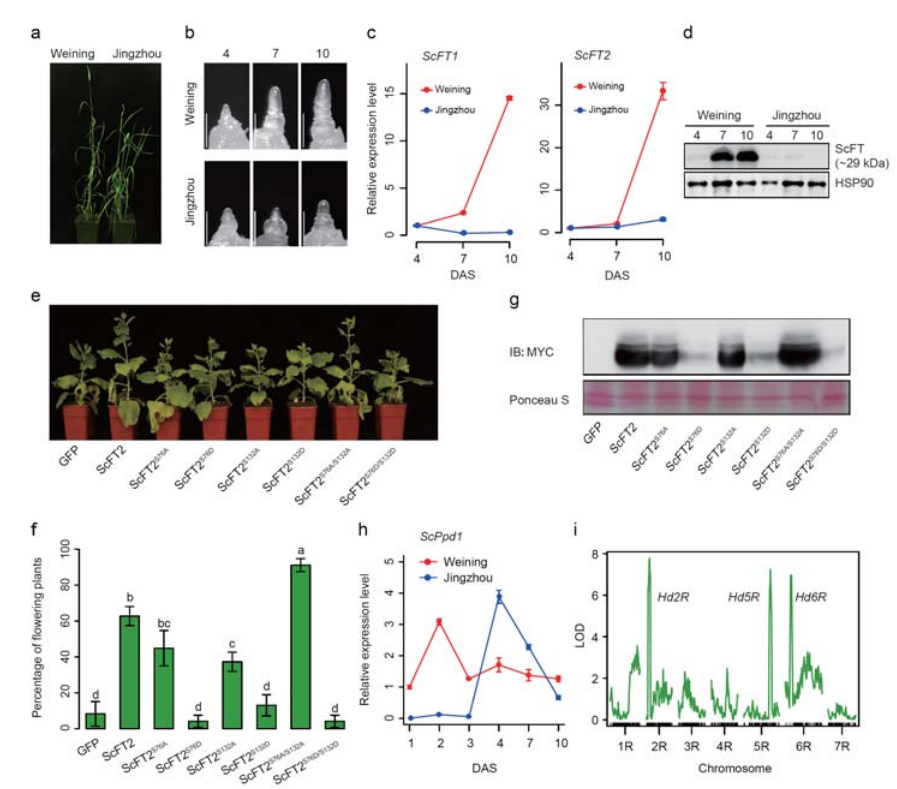
Mga feature ng development at gene expression na nauugnay sa maagang heading na katangian ng Weining rye
8. Pagmimina ng mga chromosomal region at loci na potensyal na kasangkot sa rye domestication
Isang kabuuang 123,647 SNP ang ginamit upang magsagawa ng selevtive sweep analysis sa pagitan ng cultivated rye at S. vavilovii.11 selective sweep signal na natukoy sa pamamagitan ng reduction index (DRI), fixation index (FST) at XP-CLR na paraan.Natagpuan ang ScID1 na posibleng pagkakasangkot sa regulasyon ng petsa ng heading.
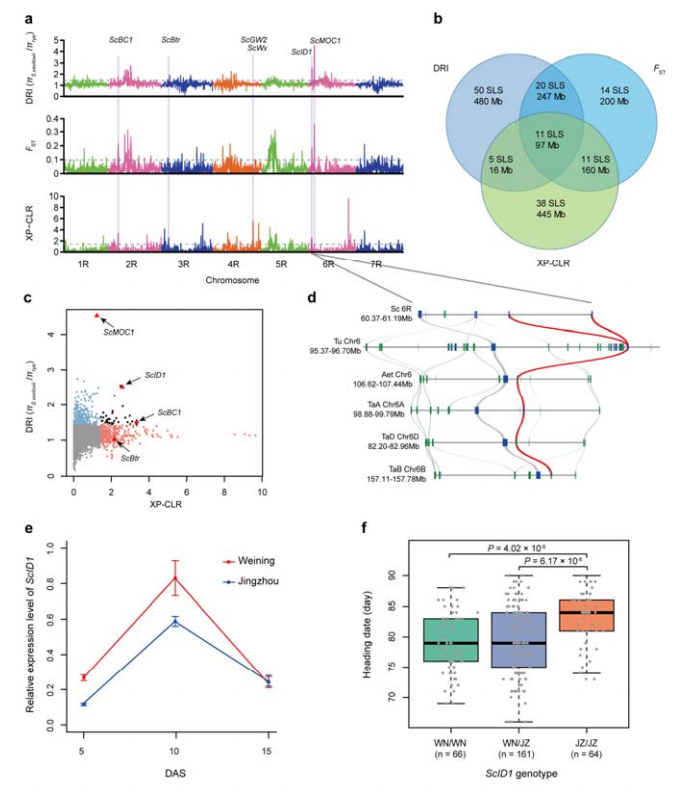
Pagkilala at pagsusuri ng mga chromosomal na rehiyon at loci na potensyal na nauugnay sa rye domestication
Sanggunian
Li GW et al.Ang isang mataas na kalidad na genome assembly ay nagha-highlight ng rye genomic na mga katangian at agronomically important genes.Nature Genetics (2021)
Mga Balita at Highlight naglalayong ibahagi ang pinakabagong matagumpay na mga kaso sa Biomarker Technologies, pagkuha ng mga nobelang siyentipikong tagumpay pati na rin ang mga kilalang pamamaraan na inilapat sa panahon ng pag-aaral.
Oras ng post: Ene-05-2022

