GENOM-EVOLUTION, PANGENOM
Was ist Pangenom?
Die gesammelten Beweise zeigen, dass die Unterschiede zwischen verschiedenen Stämmen einer Art enorm sein können.Ein einziges Genom reicht bei weitem nicht aus, um das Gesamtbild der genetischen Informationen einer einzelnen Art zu erfassen.Der Zweck der Pan-Genom-Studie besteht darin, einen umfassenderen genomischen Graphen einer Art zu erhalten und die Beziehungen zwischen Merkmalen und genetischen Codes zu entschlüsseln, indem eine Genom-De-novo-Assemblierung zahlreicher Stämme durchgeführt wird, was eine tiefere und umfassendere Analyse der Variationen ermöglicht.
Trends der Pan-Genom-Studie
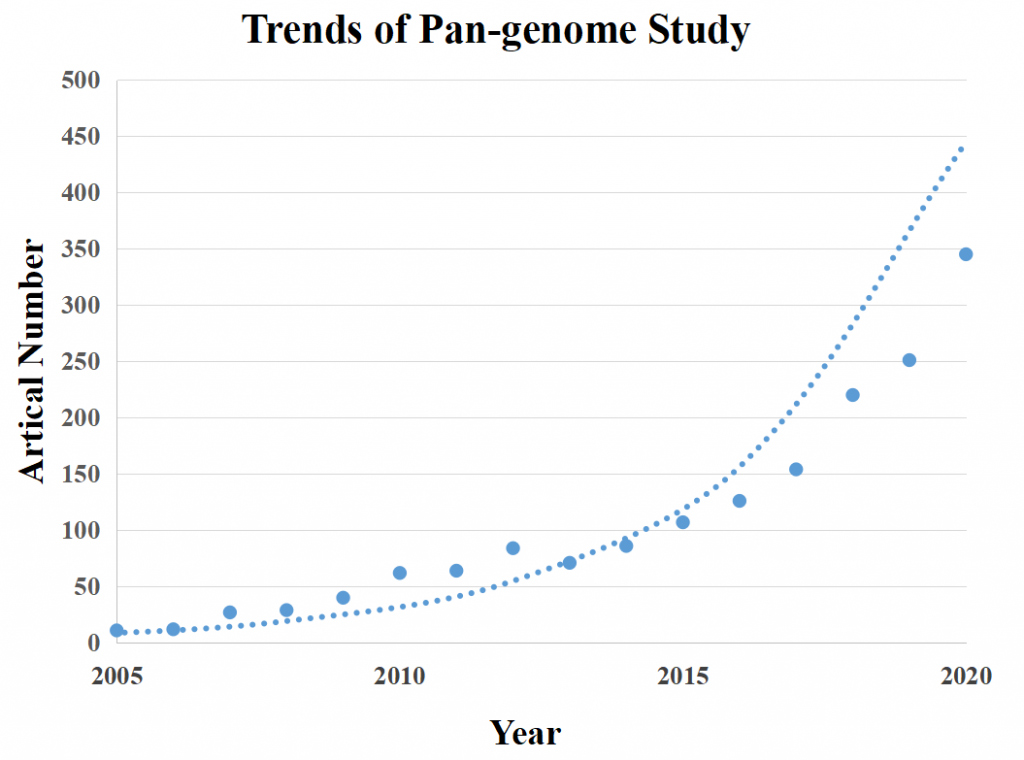
Abbildung 1 Die Trends der veröffentlichten Forschungsarbeiten zum Pan-Genom.
Hinweis: Die Abbildung zeigt das Ergebnis der Verwendung von „Pan-Genom“ als Schlüsselwort für die Suche nach Titeln von Artikeln, die in Zeitschriften der Reihe „Nature“, „Cell“ und „Science“ veröffentlicht wurden
A.Lesevorgänge aus mehreren Proben werden an einer Referenz ausgerichtet und nicht ausgerichtete Lesevorgänge werden zu neuen Contigs zusammengestellt.Durch Hinzufügen dieser neuen Contigs zur ursprünglichen Referenzsequenz kann eine Pangenom-Referenz erstellt werden.Entbehrliche Regionen werden basierend auf der Zuordnung aller Lesevorgänge zum Pangenom bestimmt.
B.Die De-novo-Assemblierung der Genome mehrerer Akzessionen ermöglicht Ansätze zur Ausrichtung des gesamten Genoms, um entbehrliche Genomregionen zu identifizieren.
C.Ein Pan-Genom-Graph kann aus Alignments des gesamten Genoms oder durch De-novo-Graph-Assemblierung erstellt werden, wodurch Varianteninformationen entbehrlicher Regionen effizient als eindeutige Pfade durch den Graphen gespeichert werden.
Wie konstruiert man ein Pan-Genom?
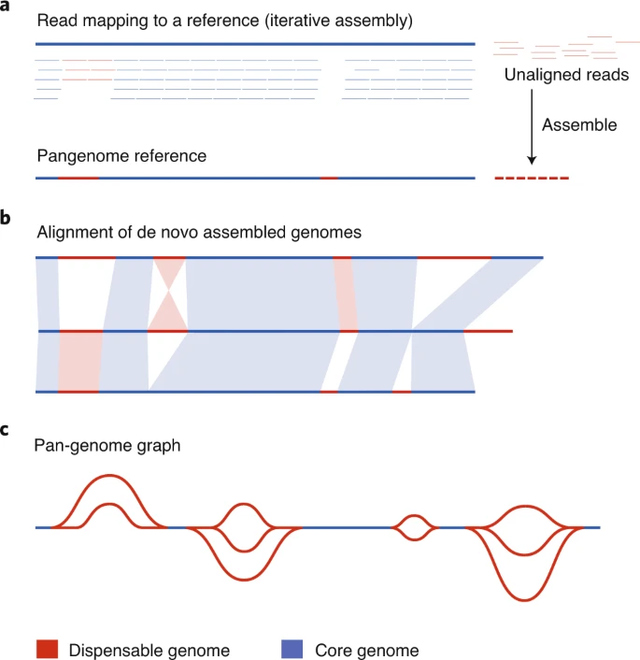
Abbildung 2 Vergleich der Pan-Genom-Ansätze1
A.Lesevorgänge aus mehreren Proben werden an einer Referenz ausgerichtet und nicht ausgerichtete Lesevorgänge werden zu neuen Contigs zusammengestellt.Durch Hinzufügen dieser neuen Contigs zur ursprünglichen Referenzsequenz kann eine Pangenom-Referenz erstellt werden.Entbehrliche Regionen werden basierend auf der Zuordnung aller Lesevorgänge zum Pangenom bestimmt.
B.Die De-novo-Assemblierung der Genome mehrerer Akzessionen ermöglicht Ansätze zur Ausrichtung des gesamten Genoms, um entbehrliche Genomregionen zu identifizieren.
C.Ein Pan-Genom-Graph kann aus Alignments des gesamten Genoms oder durch De-novo-Graph-Assemblierung erstellt werden, wodurch Varianteninformationen entbehrlicher Regionen effizient als eindeutige Pfade durch den Graphen gespeichert werden.
Kürzlich veröffentlichte Pan-Genome
● Vergewaltigungs-Pangenome2

● Tomaten-Pangenome 3

● Reis-Pan-Genom4

● Sonnenblumen-Pan-Genom5

● Sojabohnen-Pan-Genom 6

● Reis-Pan-Genom7

● Pangenom der Gerste8

● Weizen-Pangenom9

● Sorghum-Pangenom10

● Phytoplankton-Pangenom11

Referenz
1. Bayer PE, Golicz AA, Scheben A, Batley J, Edwards D. Pflanzen-Pangenome sind die neue Referenz.Nat-Pflanzen.2020;6(8):914-920.doi:10.1038/s41477-020-0733-0
2. Song JM, Guan Z, Hu J, et al.Acht hochwertige Genome enthüllen die Pan-Genom-Architektur und die Ökotyp-Differenzierung von Brassica napus.Nat-Pflanzen.2020;6(1):34-45.doi:10.1038/s41477-019-0577-7
3. Gao L, Gonda I, Sun H, et al.Das Tomaten-Pan-Genom deckt neue Gene und ein seltenes Allel auf, das den Fruchtgeschmack reguliert.Nat Genet.2019;51(6):1044-1051.doi:10.1038/s41588-019-0410-2
4. Zhao Q, Feng Q, Lu H, et al.Die Pan-Genom-Analyse verdeutlicht das Ausmaß der genomischen Variation in Kultur- und Wildreis [veröffentlichte Korrektur erscheint in Nat Genet.2018 Aug;50(8):1196].Nat Genet.2018;50(2):278-284.doi:10.1038/s41588-018-0041-z
5. Hübner S., Bercovich N., Todesco M. et al.Die Pangenomanalyse von Sonnenblumen zeigt, dass die Hybridisierung den Gengehalt und die Krankheitsresistenz veränderte.Nat-Pflanzen.2019;5(1):54-62.doi:10.1038/s41477-018-0329-0
6. Liu Y, Du H, Li P, et al.Pangenom wilder und kultivierter Sojabohnen.Zelle.2020;182(1):162-176.e13.doi:10.1016/j.cell.2020.05.023
7. Qin P, Lu H, Du H, et al.Pangenomanalyse von 33 genetisch vielfältigen Reisakzessionen deckt verborgene genomische Variationen auf [online vor Druck veröffentlicht, 25. Mai 2021].Zelle.2021;S0092-8674(21)00581-X.doi:10.1016/j.cell.2021.04.046
8. Jayakodi M, Padmarasu S, Haberer G, et al.Das Pangenom der Gerste enthüllt das verborgene Erbe der Mutationszüchtung.Natur.2020;588(7837):284-289.doi:10.1038/s41586-020-2947-8
9. Walkowiak S, Gao L, Monat C, et al.Mehrere Weizengenome offenbaren globale Variation in der modernen Züchtung.Natur.2020;588(7837):277-283.doi:10.1038/s41586-020-2961-x
10. Tao Y, Luo H, Xu J, et al.Umfangreiche Variation im Pan-Genom von kultiviertem und wildem Sorghum [online vor Druck veröffentlicht, 20. Mai 2021].Nat-Pflanzen.2021;10.1038 / s41477-021-00925-x.doi:10.1038/s41477-021-00925-x
11. Fan X, Qiu H, Han W, et al.Das Phytoplankton-Pangenom zeigt einen umfangreichen prokaryotischen horizontalen Gentransfer verschiedener Funktionen.Sci Adv.2020;6(18):eaba0111.Veröffentlicht am 29. April 2020. doi:10.1126/sciadv.aba0111
Zeitpunkt der Veröffentlichung: 04.01.2022

