Metagenomik (NGS)
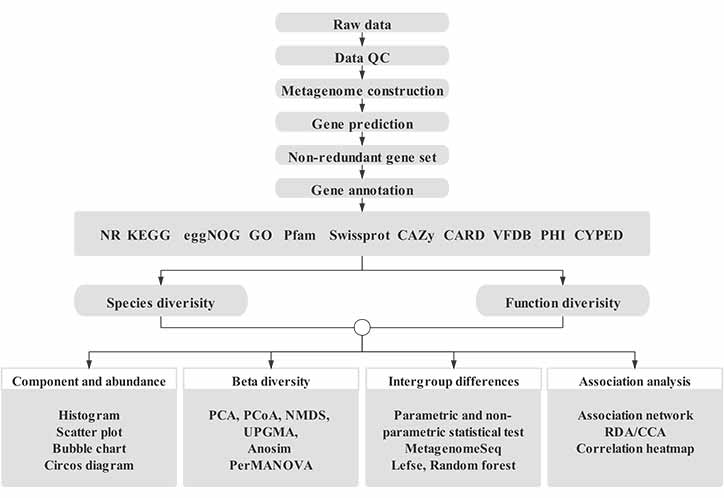
Diese Analyseplattform wurde auf der Grundlage jahrelanger Erfahrung für die Analyse metagenomischer Schrotflintendaten entwickelt.Es besteht aus einem integrierten Workflow, der verschiedene häufig benötigte Metagenomikanalysen enthält, einschließlich Datenverarbeitung, Studien auf Artenebene, Studien auf Genfunktionsebene, Metagenom-Binning usw. Darüber hinaus sind maßgeschneiderte Data-Mining-Tools für den Standardanalyse-Workflow verfügbar, einschließlich Gen- und Artenabfragen , Parametereinstellung, personalisierte Figurengenerierung usw.
Identifizierung des Transkripts
Arbeitsablauf in der Bioinformatik