Génom manusa
genetika alam
Sequencing anu dibaca panjang ngidentipikasi ékspansi ulang GGC di NOTCH2NLC anu aya hubunganana sareng panyakit inklusi intranuklear neuronal
ONT resequencing |Illumina |Sequencing exome sakabeh |CRISPR-Cas9 ONT sasaran sequencing |RNA-seq |ONT 5mC methylation nelepon
Sorotan
1.Ku analisis Patalina dina kulawarga NIID badag, dua wewengkon numbu anu dicirikeun.
2.ONT basis lila-baca sequencing na Cas-9 dimédiasi pengayaan ONT sequencing kapanggih ngabalukarkeun genetik poténsi NIID, GGC ulang ékspansi dina 5 'UTR of NOTCH2NLC.Ulikan ieu ngalaporkeun ékspansi ulang dina gén khusus manusa pikeun kahiji kalina anu mekar ngaliwatan duplikasi ségméntal.
3.RNA sequencing wangsit transkrip antisense abnormal dina awal atawa jero GGC ulang wewengkon ékspansi di NOTCH2NLC.
Latar
NPanyakit inklusi intranuklear euronal (NIID) mangrupikeun panyakit neurodegeneratif anu progresif sareng fatal, anu dicirikeun ku ayana inklusi intranuklear hyaline eosinofilik dina sistem saraf pusat sareng periferal.Manifestasi klinis anu kacida variatifna nyababkeun kasusah anu ageung dina diagnosis dugi ka bubuka biopsi kulit.Sanajan kitu, métode basis histopatologi masih nalangsara ti misdiagnosis, nu nelepon pikeun pamahaman genetik NIID.
Prestasi
Analisis Linkage
Short-read sequencing dumasar sakabeh genom sequencing (WGS) jeung sakabeh exome sequencing (WES) dipigawé dina kulawarga NIID badag (13 kapangaruhan sarta 7 anggota unaffected).Analisis linkage on SNPs sasari tina data ieu ngungkabkeun ngan dua wewengkon numbu: wewengkon 3.5 Mb di 1p36.31-p36.22 (LOD maksimum = 2.32) jeung wewengkon 58.1 Mb di 1p22.1-q21.3 (LOD maksimum: 4.21). ).Nanging, henteu aya SNP patogén atanapi CNV anu diidentifikasi di daérah anu aya hubunganana ieu.
GGC ulang ékspansi di NOTCH2NLC
Nsequencing basis anopore ieu diolah dina 13 kapangaruhan sarta 4 anggota unaffected ti 8 kulawarga (anggota séjén kapangaruhan ieu sequenced ku Pacbio lila dibaca platform sequencing.).Data anu dibaca panjang ngungkabkeun panyakit anu aya hubunganana sareng ékspansi ulangan GGC dina 5 ′ UTR tina pemetaan gén NOTCH2NLC ka 58.1 Mb dikaitkeun wilayah (Gambar 1).Ekspansi ulang ieu ogé dicirikeun dina sadaya 40 kasus NIID sporadis anu diuji ku RP-PCR.
Cas-9 dimédiasi target sequencing on nanopore platform ieu padamelan pikeun ngahontal sinyalna dibaca luhur on NOTCH2NLC ulang (100 X-1,795 X).Runtuyan konsensus ieu sapuk sareng pamanggihan saméméhna ngeunaan ékspansi ulang GGC.Leuwih ti éta, {(GGA) n (GGC) n} ulangan diidentifikasi minangka spidol genetik poténsial pikeun fénotip lemah-dominan (Gambar 2).
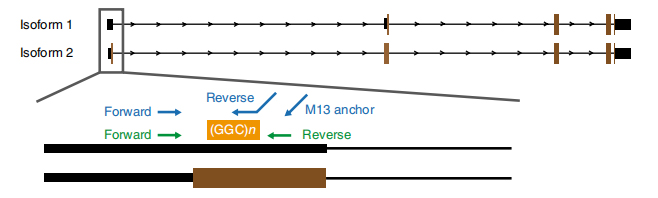
Gambar 1. Panyakit pakait ékspansi ulang dicirikeun dina exon 1 of NOTCH2NLC isoforms.
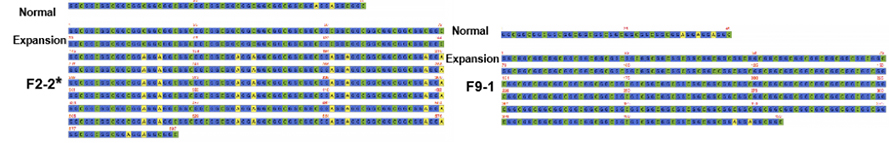
Gambar 2. Runtuyan konsensus tina ulangan NPTCH2NLC di penderita NIID kalayan (*) atanapi tanpa phenotype lemah-dominan.
NGén OTCH2NL mangrupikeun gén khusus pikeun manusa, anu dipercaya maénkeun peran penting dina évolusi otak manusa sareng panyakit saraf.Nanging, tilu gén anu aya hubunganana sareng NOTCH2 (NOTCH2NLA, NOTCH2NLB sareng NOTCH2NLC) kalayan> 99.1% idéntitas sekuen henteu direngsekeun dugi ka rakitan génom manusa panganyarna.Sequencing bébas sintésis jeung lila-dibaca dina platform nanopore geus ditémbongkeun kaunggulan kasohor di resolving wewengkon nu kasaruaan tinggi na (GGC) n repeats kalawan 100% GC-euyeub.
GGC ulang ékspansi di NOTCH2NLC
Tsequencing ranscriptome ieu diolah dina 2 kapangaruhan sarta 2 anggota unaffected.Jero bacaan anu dinormalisasi diitung dina untaian rasa sareng antisense di hulu exon munggaran paralog NOTCH2NL.Transkrip anti-sense abnormal kapanggih ngan dina kasus nu dimaksud, nu diuk di awal atawa di jero wewengkon ékspansi ulang (Puncak Ungu di F1-14 na F1-16 dina Gambar 3.).Salaku tambahan, 54 DEG diidentifikasi sareng sadayana diperkaya dina istilah GO sareng MPO anu aya hubunganana sareng fungsi neuronal.
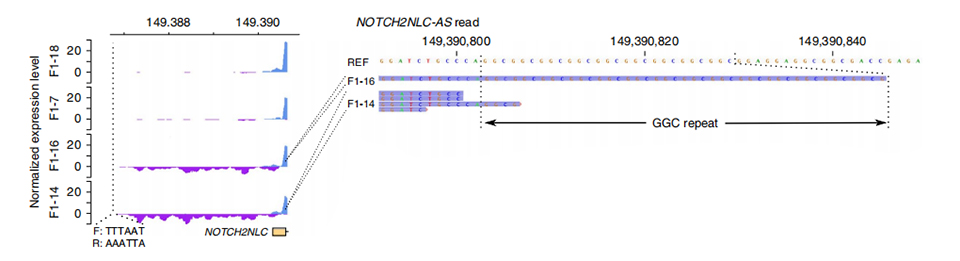
Gambar 3. Dinormalisasi jero bacaan dina hulu exon mimiti NOTCH2NLC dina kasus unaffected (di luhur) jeung kapangaruhan (handap).
Téknologi
Téknologi Nanopore Oxford (ONT)
NSequencing anopore ngabédakeun diri tina platform sequencing séjén, sabab nukléotida dibaca langsung tanpa prosés sintésis DNA.Nalika untaian tunggal DNA ngaliwatan pori protéin ukuranana nano (nanopore), nukléotida béda ngahasilkeun arus ionik béda, nu bisa direbut tur dipindahkeun kana runtuyan basa.Platform sequencing ONT sorangan henteu nunjukkeun wates téknis anu jelas dina panjang maca DNA.Ku alatan éta, Ultra-long reads (ULRs) sadia pikeun assembly génom kualitas luhur.Sumawona, bacaan anu panjang pisan ieu, anu cukup panjang pikeun nyebrang fitur sekuen kompléks atanapi variasi struktural, ngabantosan ngatasi watesan sekuen anu dibaca pondok di dieu.

Urutan nanopore
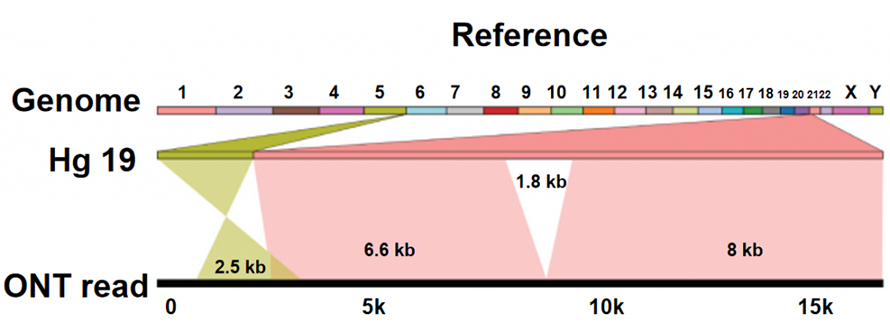
Idéntifikasi variasi struktur (SV).
Ssequencing bébas sintésis sakitu legana dilestarikan informasi methylation DNA dina citakan.Méthylated A, T, C jeung G ngahasilkeun arus ionik béda ti un-methylated, nu bisa dibaca langsung ku platform.Urutan nanopore nguatkeun profil génom sadayana 5mC sareng 6mA dina résolusi nukléotida tunggal.
Rujukan
Jun Sone, et.al.Sequencing lila-baca nangtukeun ékspansi ulang GGC di NOTCH2NLC pakait sareng kasakit inklusi intranuclear neuronal.Nature Genetics (2019)
Tech jeung Highlights Tujuanana pikeun ngabagikeun aplikasi suksés panganyarna tina téknologi sequencing throughput tinggi anu béda dina sagala rupa arena panalungtikan ogé ideu cemerlang dina desain ékspérimén sareng pertambangan data.
waktos pos: Jan-06-2022

