GENOMICA UMANE
genetica di a natura
A sequenza di lettura longa identifica espansioni di ripetizione GGC in NOTCH2NLC assuciata à a malatia di inclusione intranucleare neuronale
ONT resequencing |Illumina |Sequenza di l'esoma tutale |CRISPR-Cas9 ONT sequencing targeted |RNA-seq |Chjama di metilazione ONT 5mC
Highlights
1.By Analisi di Linkage nantu à una grande famiglia NIID, duie regioni ligati sò stati identificati.
2.A sequenza di longa lettura basata in ONT è a sequenza ONT di arricchimentu mediata da Cas-9 hà scupertu una causa genetica potenziale di NIID, espansione di ripetizioni GGC in 5' UTR di NOTCH2NLC.Stu studiu hà riportatu espansioni ripetute in geni specifichi di l'omu per a prima volta chì anu evolutu attraversu duplicazioni segmentali.
A sequenza di 3.RNA hà revelatu trascrizioni antisensu anormali in u principiu o in e regioni di espansione GGC ripetite in NOTCH2NLC.
Sfondate
NA malatia d'inclusione intranucleare euronal (NIID) hè una malatia neurodegenerativa progressiva è fatale, chì hè carattarizata da a presenza di inclusioni intranucleari ialine eosinofili in i sistemi nervosi cintrali è periferici.E so manifestazioni cliniche assai variate susciteghjanu grandi difficultà in u diagnosticu finu à l'intruduzioni di a biòpsia di a pelle.In ogni casu, i metudi basati in l'istopatologia sò sempre soffrenu di misdiagnosis, chì dumanda una cunniscenza genetica di NIID.
Rializazioni
Analisi di ligami
SHort-read sequencing basatu in tuttu u genoma sequencing (WGS) è sequencing exome sanu (WES) hè stata realizata nantu à una grande famiglia NIID (13 affettati è 7 membri micca affettati).L'analisi di linkage nantu à SNP estratti da queste dati hà revelatu solu duie regioni ligati: una regione di 3.5 Mb à 1p36.31-p36.22 (LOD massimu = 2.32) è una regione di 58.1 Mb à 1p22.1-q21.3 (LOD massimu: 4.21). ).Tuttavia, nisun SNP o CNV patogeni sò stati identificati in queste regioni ligati.
GGC ripete espansioni in NOTCH2NLC
NA sequenza basata in anopore hè stata processata nantu à 13 affettati è 4 membri micca affettati da 8 famiglie (un altru membru affettatu hè statu sequenziatu da a piattaforma di sequenza di lettura longa Pacbio).I dati di lettura longa anu revelatu espansioni ripetute GGC associate à a malatia in u 5' UTR di a mappatura di u gene NOTCH2NLC à a regione ligata à 58,1 Mb (Figura 1).Queste espansioni ripetute sò state identificate ancu in tutti i 40 casi sporadici di NIID testati da RP-PCR.
CA sequenza di destinazione mediata da as-9 nantu à a piattaforma nanopore hè stata impiegata per ottene una copertura di lettura più alta nantu à a ripetizione NOTCH2NLC (100 X-1,795 X).Queste sequenze di cunsensu accunsenu bè cù i scuperti precedenti nantu à l'espansione di ripetizione di GGC.Inoltre, {(GGA) n (GGC) n} n ripetizioni sò stati identificati cum'è un marcatore geneticu potenziale per u fenotipu dominante di debule (Figura 2).
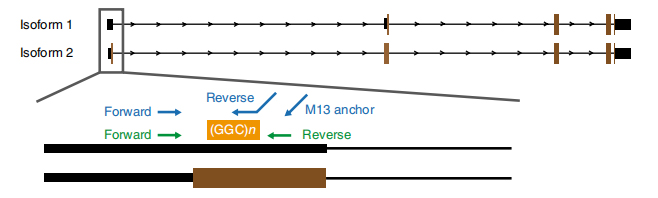
Figura 1. L'espansione ripetuta assuciata à a malatia identificata nantu à l'exon 1 di l'isoforme NOTCH2NLC.
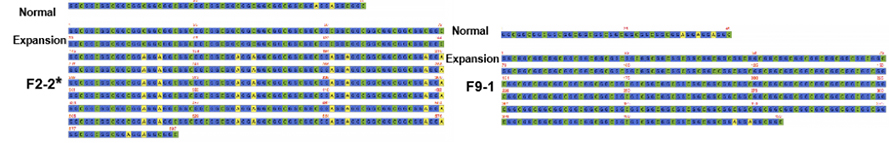
Figura 2. Sequenze di cunsensu di NPTCH2NLC ripetite in pazienti NIID cù (*) o senza fenotipu dominante di debule.
NI geni OTCH2NL sò geni specifichi di l'omu, chì si crede chì ghjucanu un rolu vitale in l'evoluzione di u cervellu umanu è e malatie neurologiche.In ogni casu, trè geni cunnessi NOTCH2 (NOTCH2NLA, NOTCH2NLB è NOTCH2NLC) cù identità di sequenza> 99.1% ùn sò stati risolti finu à l'ultima assemblea di genoma umanu.A sequenza senza sintesi è a longa lettura nantu à a piattaforma nanopore anu dimustratu vantaghji notevuli in a risoluzione di e regioni di alta similarità è (GGC)n ripetizioni cù 100% GC-rich.
GGC ripete espansioni in NOTCH2NLC
TA sequenza di ranscriptome hè stata processata nantu à 2 membri affettati è 2 micca affettati.A prufundità di lettura nurmalizata hè stata calculata nantu à i filamenti sensu è antisensu in upstream di i primi esoni di paralogi NOTCH2NL.Trascrizioni anti-sensu anormali sò stati trovati solu in i casi affettati, chì si trovanu in u principiu o in a regione di espansione ripetuta (Purple picchi in F1-14 è F1-16 in Figura 3.).Inoltre, 54 DEG sò stati identificati è tutti sò stati arricchiti in termini GO è MPO in relazione à e funzioni neuronali.
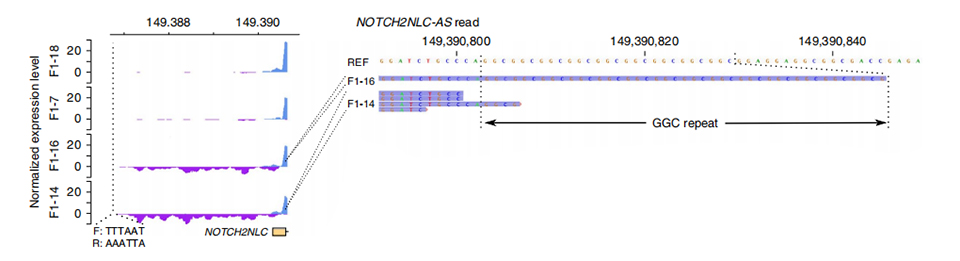
Figura 3. Profundità di lettura normalizzata in upstream di u primu exon di NOTCH2NLC in casi micca affettati (sopra) è affettati (sottu).
Tecnulugia
Oxford Nanopore Teghnologies (ONT)
NA sequencing anopore si distingue da altre piattaforme di sequenza, in chì i nucleotidi sò letti direttamente senza prucessu di sintesi di DNA.Cum'è una sola fila di DNA passa per un poru di proteina nano-sized (nanopore), i diversi nucleotidi generanu diverse correnti ioniche, chì ponu esse catturati è trasferiti in sequenza di basi.A piattaforma di sequenza ONT stessa ùn mostra micca un limite tecnicu apparente nantu à a durata di a lettura di l'ADN.Dunque, letture Ultra-long (ULR) sò dispunibuli per l'assemblea di genoma di alta qualità.Inoltre, queste letture estremamente lunghe, chì sò abbastanza longu per attraversà funzioni di sequenza cumplessa o variazioni strutturali, aiutanu à superà e limitazioni di a sequenza di lettura corta quì.

Sequenza di nanopori
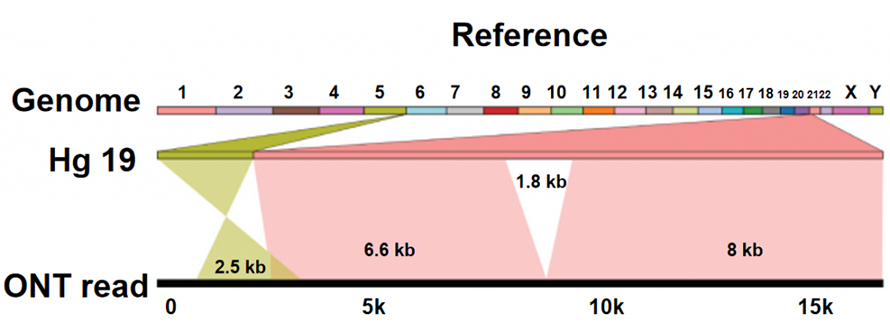
Identificazione di variazioni di struttura (SV).
SA sequenza senza sintesi hà largamente preservatu l'infurmazioni di metilazione di DNA nantu à u mudellu.A, T, C è G metilati generanu correnti ioniche distinti da quelli non metilati, chì ponu esse letti direttamente da a piattaforma.A sequenza di nanopori permette a profilazione di tuttu u genoma di 5mC è 6mA à una risoluzione di un nucleotide.
Riferimentu
Jun Sone, et.al.A sequenza di lettura longa identifica espansioni di ripetizione GGC in NOTCH2NLC assuciata à a malatia di inclusione intranucleare neuronale.Genetica di a natura (2019)
Tecnulugia è Highlights hà u scopu di sparta l'applicazioni più recenti di successu di diverse tecnulugia di sequenza d'alta produzzione in diverse aree di ricerca, è ancu idee brillanti in u disignu sperimentale è l'estrazione di dati.
Tempu di posta: 06-Jan-2022

