MENSELIJKE GENOMICA
natuur genetica
Lang gelezen sequencing identificeert uitbreidingen van GGC-herhalingen in NOTCH2NLC geassocieerd met neuronale intranucleaire inclusieziekte
ONT-herschikking |Illumina |Hele exome-sequencing |CRISPR-Cas9 ONT gerichte sequencing |RNA-seq |ONT 5mC methylatieoproep
Hoogtepunten
1. Door koppelingsanalyse van een grote NIID-familie werden twee gekoppelde regio's geïdentificeerd.
2.ONT-gebaseerde long-read sequencing en Cas-9-gemedieerde verrijking ONT-sequencing ontdekte een potentiële genetische oorzaak van NIID, GGC-herhalingsuitbreidingen in 5' UTR van NOTCH2NLC.Deze studie rapporteerde voor het eerst herhaalde uitbreidingen in mensspecifieke genen die evolueerden door segmentale duplicaties.
3.RNA-sequencing onthulde abnormale antisense-transcripten in het begin of binnen GGC-herhalingsexpansiegebieden in NOTCH2NLC.
Achtergrond
Neuronale intranucleaire inclusieziekte (NIID) is een progressieve en fatale neurodegeneratieve ziekte, die wordt gekenmerkt door de aanwezigheid van eosinofiele hyaliene intranucleaire insluitsels in het centrale en perifere zenuwstelsel.De zeer variabele klinische manifestaties ervan veroorzaken grote problemen bij de diagnose tot de introductie van huidbiopten.Op histopathologie gebaseerde methoden lijden echter nog steeds onder verkeerde diagnoses, wat vraagt om een genetisch begrip van NIID.
Prestaties
Koppelingsanalyse
Sop hort-read sequencing gebaseerde Whole Genome Sequencing (WGS) en Whole Exome Sequencing (WES) werden uitgevoerd op een grote NIID-familie (13 getroffen en 7 niet-getroffen leden).Koppelingsanalyse op SNP's geëxtraheerd uit deze gegevens bracht slechts twee gekoppelde regio's aan het licht: een regio van 3,5 Mb op 1p36.31-p36.22 (maximale LOD = 2,32) en een regio van 58,1 Mb op 1p22.1-q21.3 (maximale LOD: 4,21 ).Er werden echter geen pathogene SNP's of CNV's geïdentificeerd in deze gekoppelde regio's.
GGC herhaaluitbreidingen in NOTCH2NLC
Nop anopore gebaseerde sequencing werd verwerkt op 13 getroffen en 4 niet-getroffen leden uit 8 families (een ander getroffen lid werd gesequenced door Pacbio long read sequencing-platform).Lang gelezen gegevens onthulden ziekte-geassocieerde GGC-herhalingsuitbreidingen in de 5'-UTR van NOTCH2NLC-gentoewijzing naar een gekoppeld gebied van 58,1 Mb (Figuur 1).Deze herhaalde uitbreidingen werden ook geïdentificeerd in alle 40 sporadische NIID-gevallen die met RP-PCR werden getest.
Cas-9-gemedieerde doelsequencing op een nanoporieplatform werd gebruikt om een hogere leesdekking te bereiken op NOTCH2NLC-herhaling (100 X-1.795 X).Deze consensussequenties kwamen goed overeen met eerdere bevindingen over GGC-herhalingsuitbreidingen.Bovendien werden {(GGA)n (GGC)n}n-herhalingen geïdentificeerd als een potentiële genetische marker voor zwakte-dominant fenotype (Figuur 2).
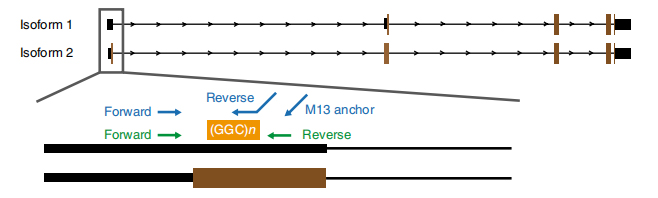
Figuur 1. Ziekte-geassocieerde herhalingsexpansie geïdentificeerd op exon 1 van NOTCH2NLC-isovormen.
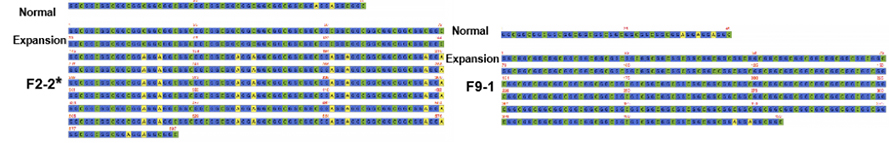
Figuur 2. Consensussequenties van NPTCH2NLC-herhaling bij NIID-patiënten met(*) of zonder zwakte-dominant fenotype
NOTCH2NL-genen zijn mensspecifieke genen waarvan wordt aangenomen dat ze een cruciale rol spelen in de evolutie van het menselijk brein en neurologische ziekten.Drie NOTCH2-gerelateerde genen (NOTCH2NLA, NOTCH2NLB en NOTCH2NLC) met >99,1% sequentie-identiteit werden echter pas opgelost tijdens de laatste assemblage van het menselijk genoom.Synthesevrije en langgelezen sequencing op een nanoporieplatform hebben opmerkelijke voordelen laten zien bij het oplossen van regio's met hoge gelijkenis en (GGC)n-herhalingen met 100% GC-rijk.
GGC herhaaluitbreidingen in NOTCH2NLC
Transcriptoomsequencing werd verwerkt op 2 getroffen en 2 niet-getroffen leden.Genormaliseerde leesdiepte werd berekend op sense- en antisense-strengen stroomopwaarts van de eerste exons van NOTCH2NL-paralogen.Abnormale antisense-transcripten werden alleen gevonden in getroffen gevallen, die zich in het begin of binnen het herhaalde expansiegebied bevinden (paarse pieken in F1-14 en F1-16 in figuur 3).Bovendien werden 54 DEG's geïdentificeerd en ze waren allemaal verrijkt met GO- en MPO-termen gerelateerd aan neuronale functies.
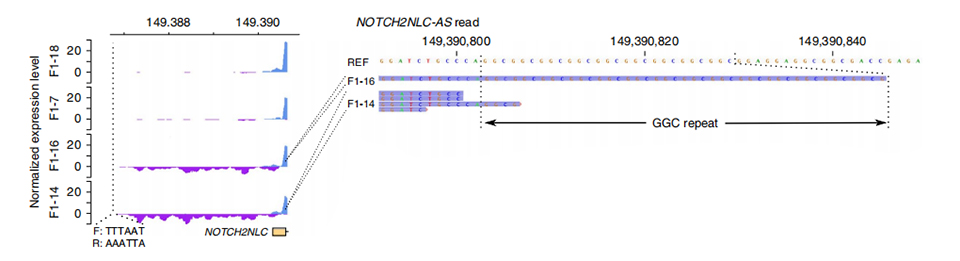
Figuur 3. Genormaliseerde leesdiepte stroomopwaarts van het eerste exon van NOTCH2NLC in niet-getroffen (boven) en getroffen (onder) gevallen.
Technologie
Oxford Nanopore-technologie (ONT)
Nanopore-sequencing onderscheidt zich van andere sequencing-platforms doordat de nucleotiden direct worden uitgelezen zonder DNA-syntheseproces.Terwijl een enkele DNA-streng door een eiwitporie van nanogrootte (nanoporie) gaat, genereren verschillende nucleotiden verschillende ionenstromen, die kunnen worden opgevangen en overgedragen in een reeks basen.Het ONT-sequencingplatform zelf vertoont geen duidelijke technische limiet op de lengte van het DNA-lezen.Daarom zijn Ultra-long reads (ULR's) beschikbaar voor genoomassemblage van hoge kwaliteit.Bovendien helpen deze extreem lange leesbewerkingen, die lang genoeg zijn om complexe sequentiekenmerken of structurele variaties te doorkruisen, hier de beperkingen van korte leessequenties te overwinnen.

Nanoporiënsequencing
Identificatie van structuurvariatie (SV).
Ssynthesevrije sequencing behield grotendeels DNA-methylatie-informatie op de sjabloon.Gemethyleerde A, T, C en G genereren andere ionenstromen dan niet-gemethyleerde, die rechtstreeks door het platform kunnen worden gelezen.Nanopore-sequencing maakt profilering van het hele genoom mogelijk van zowel 5mC als 6mA met een resolutie van één nucleotide.
Referentie
Jun Sone, enz.al.Lang gelezen sequencing identificeert uitbreidingen van GGC-herhalingen in NOTCH2NLC geassocieerd met neuronale intranucleaire inclusieziekte.Natuurgenetica (2019)
Technologie en hoogtepunten heeft tot doel de meest recente succesvolle toepassing van verschillende high-throughput sequencing-technologieën in verschillende onderzoeksarena's te delen, evenals briljante ideeën op het gebied van experimenteel ontwerp en datamining.
Posttijd: 06-jan-2022

