MENSCH GENOMIK
Naturgenetik
Laang gelies Sequenzéierung identifizéiert GGC Widderhuelung Expansioune bei NOTCH2NLC verbonne mat neuronaler intranuklearer Inklusiounskrankheet
ONT resequencing |Illumina |Ganz Exome Sequenzéierung |CRISPR-Cas9 ONT gezielte Sequenzéierung |RNA-seq |ONT 5mC Methylatioun ruffen
Highlights
1.By Verknüpfungsanalyse op enger grousser NIID Famill goufen zwee verlinkte Regiounen identifizéiert.
2.ONT-baséiert laang gelies Sequencing a Cas-9 vermëttelt Beräicherung ONT Sequencing entdeckt eng potenziell genetesch Ursaach vun NIID, GGC widderhuelen Expansioune am 5' UTR vun NOTCH2NLC.Dës Etude huet widderholl Expansioune a mënschlech spezifesch Genen fir d'éischte Kéier gemellt, déi sech duerch segmental Duplikatioune evoluéiert hunn.
3.RNA Sequencing opgedeckt anormal Antisense Transkriptiounen am Ufank oder bannent GGC widderhuelen Expansioun Regiounen an NOTCH2NLC.
Hannergrond
NEuronal Intranuklear Inklusiounskrankheet (NIID) ass eng progressiv a fatal neurodegenerativ Krankheet, déi duerch d'Präsenz vun eosinophilen hyaline intranuklearen Inklusiounen an zentrale a periphere Nervensystemer charakteriséiert ass.Seng héich variabel klinesch Manifestatiounen erhéijen grouss Schwieregkeeten bei der Diagnostik bis d'Aféierung vun der Hautbiopsie.Wéi och ëmmer, Histopathologie-baséiert Methoden leiden nach ëmmer vu falscher Diagnostik, wat e genetesch Verständnis vun NIID fuerdert.
Leeschtungen
Verknëppung Analyse
SHort-liesen sequencing baséiert ganze Genom sequencing (WGS) a ganz exome sequencing (WES) war op enger grousser NIID Famill gesuergt (13 betraff a 7 net beaflosst Memberen).Verknüpfungsanalyse op SNPs, déi aus dësen Daten extrahéiert goufen, hunn nëmmen zwou verlinkte Regiounen opgedeckt: eng 3.5 Mb Regioun bei 1p36.31-p36.22 (maximal LOD=2.32) an eng 58.1 Mb Regioun bei 1p22.1-q21.3 (maximal LOD: 4.21) ).Wéi och ëmmer, keng pathogen SNPs oder CNVs goufen an dëse verlinkte Regiounen identifizéiert.
GGC widderhuelen Expansioune an NOTCH2NLC
NAnopore-baséiert Sequencing gouf op 13 betraffene a 4 onaffektéierte Memberen aus 8 Famillen veraarbecht (en aneren betraffene Member gouf vun der Pacbio laang gelies Sequenzéierungsplattform sequenzéiert.).Laang-liesen Donnéeën opgedeckt Krankheet assoziéiert GGC widderhuelen Expansioune am 5 'UTR vun NOTCH2NLC Gentherapie Kaart ze 58,1 Mb verbonne Regioun (Dorënner 1).Dës Widderhuelung Expansioune goufen och an all 40 sporadeschen NIID Fäll getest duerch RP-PCR identifizéiert.
Cas-9 mediéiert Zilsequenzéierung op der Nanopore Plattform gouf benotzt fir méi héich Liesdeckung op NOTCH2NLC Widderhuelung (100 X-1,795 X) z'erreechen.Dës Konsenssequenzen waren gutt mat fréiere Befunde iwwer GGC widderhuelen Expansioune eens.Ausserdeem goufen {(GGA) n (GGC) n} n Widderhuelunge als potenziell genetesch Marker fir Schwäch-dominante Phänotyp identifizéiert (Dorënner 2).
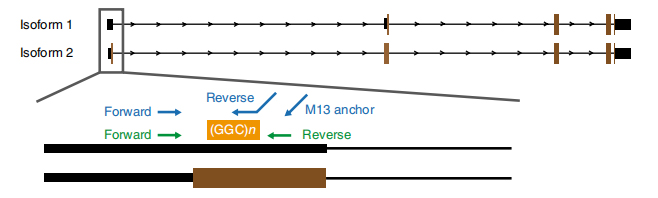
Figur 1. Krankheet verbonne widderhuelen Expansioun identifizéiert op Exon 1 vun NOTCH2NLC isoforms.
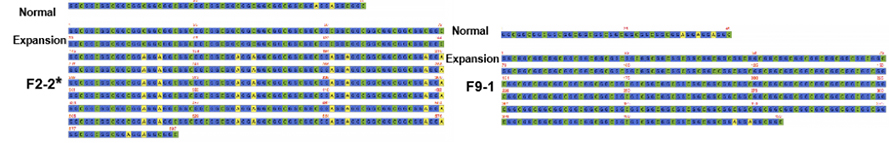
Figur 2. Konsens Sequenzen vun NPTCH2NLC Widderhuelung bei NIID Patienten mat (*) oder ouni Schwächt dominante Phänotyp
NOTCH2NL Genen si mënschlech spezifesch Genen, déi gegleeft sinn eng vital Roll an der mënschlecher Gehirn Evolutioun an neurologescher Krankheeten ze spillen.Wéi och ëmmer, dräi NOTCH2-Zesummenhang Genen (NOTCH2NLA, NOTCH2NLB an NOTCH2NLC) mat> 99,1% Sequenz Identitéit goufen net bis déi lescht mënschlech Genom Assemblée geléist.Synthese-gratis a laang gelies Sequenzéierung op der Nanopore Plattform hunn bemierkenswäert Virdeeler gewisen fir Regioune mat héijer Ähnlechkeet ze léisen an (GGC)n Wiederholungen mat 100% GC-räich.
GGC widderhuelen Expansioune an NOTCH2NLC
TRanscriptome Sequenzéierung gouf op 2 betraffene an 2 onaffektéierte Memberen veraarbecht.Normaliséiert Liesdéift gouf berechent op Sënn an Antisense Strécke am Upstream vun den éischten Exonen vun NOTCH2NL Paralogen.Abnormal Anti-Sënn Transkriptiounen goufen nëmmen a betraffene Fäll fonnt, déi am Ufank oder an der Widderhuelung Expansiounsregioun sëtzen (Purple Peaks an F1-14 a F1-16 an der Figur 3.).Zousätzlech goufen 54 DEGs identifizéiert an all goufen an GO an MPO Begrëffer beräichert am Zesummenhang mat neuronale Funktiounen.
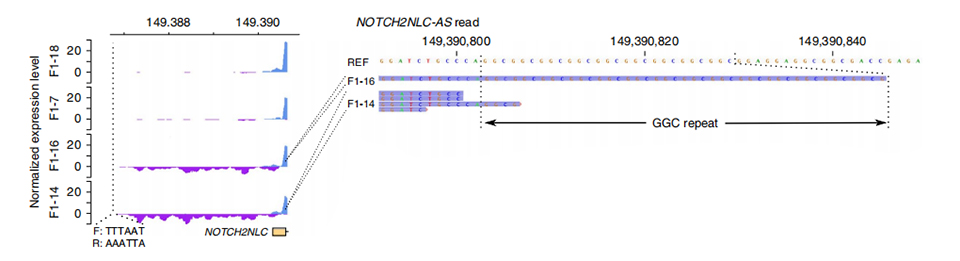
Figur 3. Normaliséiert liesen Déift op upstream vun der éischter Exon vun NOTCH2NLC an net beaflosst (uewen) a betraff (ënner) Fäll.
Technologie
Oxford Nanopore Technologies (ONT)
NAnopore Sequenzéierung ënnerscheet sech vun anere Sequenzéierungsplattformen, an datt d'Nukleotiden direkt ouni DNA Syntheseprozess gelies ginn.Wéi eng eenzeg Strang DNA duerch eng Nano-Gréisst Proteinpore (Nanopore) passéiert, generéieren verschidden Nukleotiden verschidde ionesch Stréimunge, déi a Sequenz vu Basen ageholl kënne ginn.ONT Sequenzéierungsplattform selwer weist keng scheinbar technesch Limit op der Längt vun der DNA Liesung.Dofir sinn Ultra-laang Liesungen (ULRs) verfügbar fir Genom Assemblée vu héich Qualitéit.Ausserdeem hëllefen dës extrem laang Liesungen, déi laang genuch sinn fir komplex Sequenzfeatures oder strukturell Variatioun ze iwwerwannen, d'Limitatiounen vun der Kuerzliese Sequenzéierung hei ze iwwerwannen.

Nanopore Sequenzéierung
Struktur Variatioun (SV) Identifikatioun
Synthesis-gratis Sequenzéierung gréisstendeels konservéiert DNA Methylatiounsinformatioun op Schabloun.Methyléiert A, T, C a G generéieren ënnerschiddlech ionesch Stréimunge vun on-methyléierten, déi direkt vun der Plattform gelies kënne ginn.Nanopore Sequencing erméiglecht d'ganz Genomprofiléierung vu béide 5mC a 6mA bei Single-Nukleotid Opléisung.
Referenz
Jun Sone, et al.al.Laang gelies Sequenzéierung identifizéiert GGC Widderhuelung Expansioune bei NOTCH2NLC verbonne mat neuronaler intranuklearer Inklusiounskrankheet.Naturgenetik (2019)
Tech an Highlights zielt fir déi lescht erfollegräich Uwendung vu verschiddenen High-Throughput-Sequenzéierungstechnologien a verschiddene Fuerschungsarena ze deelen, souwéi genial Iddien am experimentellen Design an Datemining.
Post Zäit: Jan-06-2022

