MENSCHLICHE GENOMIK
Naturgenetik
Long-Read-Sequenzierung identifiziert GGC-Wiederholungsexpansionen in NOTCH2NLC, die mit einer neuronalen intranukleären Einschlusserkrankung verbunden sind
ONT-Resequenzierung |Illumina |Sequenzierung des gesamten Exoms |CRISPR-Cas9 ONT gezielte Sequenzierung |RNA-seq |ONT 5mC Methylierungsaufruf
Höhepunkte
1. Durch die Verknüpfungsanalyse einer großen NIID-Familie wurden zwei verknüpfte Regionen identifiziert.
2. ONT-basierte Long-Read-Sequenzierung und Cas-9-vermittelte Anreicherungs-ONT-Sequenzierung entdeckten eine mögliche genetische Ursache für NIID- und GGC-Wiederholungsexpansionen in der 5′-UTR von NOTCH2NLC.Diese Studie berichtete erstmals über wiederholte Erweiterungen in humanspezifischen Genen, die sich durch segmentale Duplikationen entwickelten.
3. Die RNA-Sequenzierung ergab abnormale Antisense-Transkripte am Anfang oder innerhalb der GGC-Repeat-Expansionsregionen in NOTCH2NLC.
Hintergrund
NDie euronale intranukleäre Einschlusskrankheit (NIID) ist eine fortschreitende und tödliche neurodegenerative Erkrankung, die durch das Vorhandensein eosinophiler hyaliner intranukleärer Einschlüsse im zentralen und peripheren Nervensystem gekennzeichnet ist.Seine sehr unterschiedlichen klinischen Manifestationen führen zu großen Schwierigkeiten bei der Diagnose bis zur Einführung einer Hautbiopsie.Bei histopathologischen Methoden kommt es jedoch immer noch zu Fehldiagnosen, die ein genetisches Verständnis von NIID erfordern.
Erfolge
Verknüpfungsanalyse
SAn einer großen NIID-Familie (13 betroffene und 7 nicht betroffene Mitglieder) wurde eine auf Kurzlesesequenzierung basierende Sequenzierung des gesamten Genoms (WGS) und Sequenzierung des gesamten Exoms (WES) durchgeführt.Die Verknüpfungsanalyse der aus diesen Daten extrahierten SNPs ergab nur zwei verknüpfte Regionen: eine 3,5-MB-Region bei 1p36.31-p36.22 (maximale LOD = 2,32) und eine 58,1-MB-Region bei 1p22.1-q21.3 (maximale LOD: 4,21). ).In diesen verknüpften Regionen wurden jedoch keine pathogenen SNPs oder CNVs identifiziert.
GGC wiederholt Erweiterungen in NOTCH2NLC
NAnopore-basierte Sequenzierung wurde bei 13 betroffenen und 4 nicht betroffenen Mitgliedern aus 8 Familien durchgeführt (ein weiteres betroffenes Mitglied wurde von der Long-Read-Sequenzierungsplattform Pacbio sequenziert).Langfristig gelesene Daten zeigten krankheitsbedingte GGC-Wiederholungsexpansionen in der 5'-UTR der NOTCH2NLC-Genkartierung in der 58,1-Mb-verknüpften Region (Abbildung 1).Diese wiederholten Erweiterungen wurden auch in allen 40 durch RP-PCR getesteten sporadischen NIID-Fällen identifiziert.
CDie as-9-vermittelte Zielsequenzierung auf einer Nanoporenplattform wurde eingesetzt, um eine höhere Leseabdeckung bei NOTCH2NLC-Wiederholungen (100 x bis 1.795 x) zu erreichen.Diese Konsenssequenzen stimmten gut mit früheren Erkenntnissen zu GGC-Wiederholungsexpansionen überein.Darüber hinaus wurden {(GGA)n (GGC)n}n Wiederholungen als potenzieller genetischer Marker für den schwächedominanten Phänotyp identifiziert (Abbildung 2).
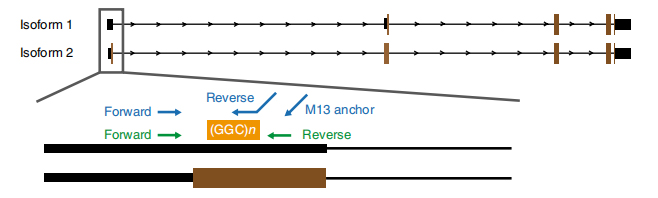
Abbildung 1. Krankheitsbedingte Wiederholungsexpansion, identifiziert auf Exon 1 der NOTCH2NLC-Isoformen.
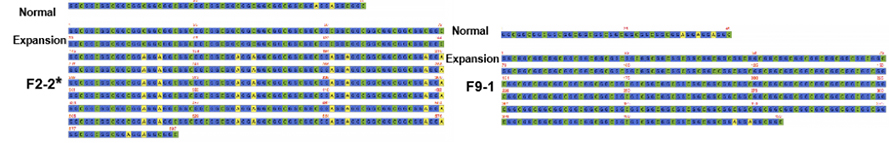
Abbildung 2. Konsenssequenzen der NPTCH2NLC-Wiederholung bei NIID-Patienten mit (*) oder ohne schwächedominantem Phänotyp
NOTCH2NL-Gene sind menschenspezifische Gene, von denen angenommen wird, dass sie eine entscheidende Rolle bei der Evolution des menschlichen Gehirns und bei neurologischen Erkrankungen spielen.Allerdings wurden drei mit NOTCH2 verwandte Gene (NOTCH2NLA, NOTCH2NLB und NOTCH2NLC) mit einer Sequenzidentität von >99,1 % erst bei der letzten Zusammenstellung des menschlichen Genoms aufgelöst.Synthesefreie und Long-Read-Sequenzierung auf einer Nanoporenplattform haben bemerkenswerte Vorteile bei der Auflösung von Regionen mit hoher Ähnlichkeit und (GGC)n-Wiederholungen mit 100 % GC-reich gezeigt.
GGC wiederholt Erweiterungen in NOTCH2NLC
TDie Ranskriptomsequenzierung wurde bei zwei betroffenen und zwei nicht betroffenen Mitgliedern durchgeführt.Die normalisierte Lesetiefe wurde für Sense- und Antisense-Stränge vor den ersten Exons von NOTCH2NL-Paralogs berechnet.Abnormale Antisense-Transkripte wurden nur in betroffenen Fällen gefunden, die am Anfang oder innerhalb der Wiederholungsexpansionsregion liegen (violette Peaks in F1-14 und F1-16 in Abbildung 3).Darüber hinaus wurden 54 DEGs identifiziert und alle mit GO- und MPO-Begriffen im Zusammenhang mit neuronalen Funktionen angereichert.
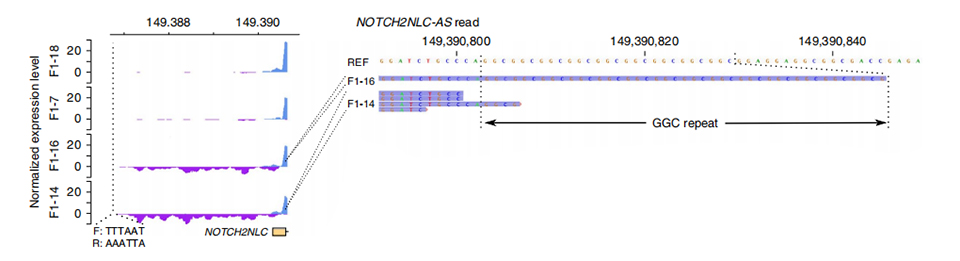
Abbildung 3. Normalisierte Lesetiefe vor dem ersten Exon von NOTCH2NLC in nicht betroffenen (oben) und betroffenen (unten) Fällen.
Technologie
Oxford Nanopore Teghnologies (ONT)
NDie Anopore-Sequenzierung unterscheidet sich von anderen Sequenzierungsplattformen dadurch, dass die Nukleotide direkt ohne DNA-Syntheseprozess abgelesen werden.Wenn ein einzelner DNA-Strang eine Proteinpore (Nanopore) in Nanogröße passiert, erzeugen verschiedene Nukleotide unterschiedliche Ionenströme, die eingefangen und in eine Basensequenz übertragen werden können.Die ONT-Sequenzierungsplattform selbst weist keine erkennbare technische Grenze für die Länge des DNA-Lesens auf.Daher stehen Ultra-Long Reads (ULRs) für die Genomassemblierung in hoher Qualität zur Verfügung.Darüber hinaus tragen diese extrem langen Lesevorgänge, die lang genug sind, um komplexe Sequenzmerkmale oder strukturelle Variationen zu überqueren, dazu bei, die Einschränkungen der Sequenzierung mit kurzen Lesevorgängen zu überwinden.

Nanoporensequenzierung
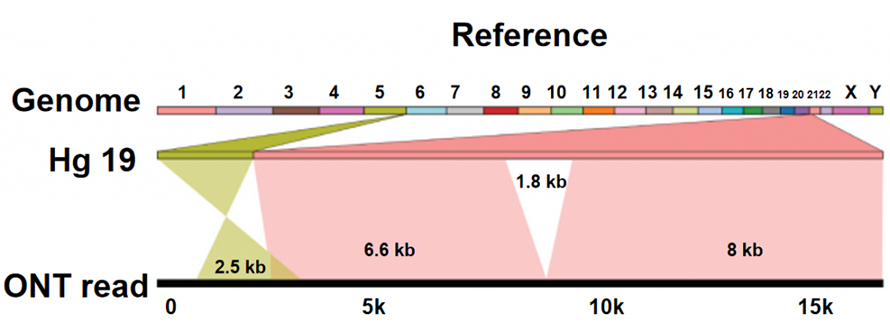
Identifizierung von Strukturvariationen (SV).
SDurch die synthesefreie Sequenzierung blieben die DNA-Methylierungsinformationen auf der Vorlage weitgehend erhalten.Methyliertes A, T, C und G erzeugen im Vergleich zu nicht methylierten unterschiedliche Ionenströme, die direkt von der Plattform abgelesen werden können.Die Nanoporensequenzierung ermöglicht die Profilierung des gesamten Genoms von 5 mC und 6 mA mit Einzelnukleotidauflösung.
Referenz
Jun Sone et.al.Long-Read-Sequenzierung identifiziert GGC-Wiederholungsexpansionen in NOTCH2NLC, die mit einer neuronalen intranukleären Einschlusserkrankung verbunden sind.Naturgenetik (2019)
Technik und Highlights Ziel ist es, die neuesten erfolgreichen Anwendungen verschiedener Hochdurchsatz-Sequenzierungstechnologien in verschiedenen Forschungsbereichen sowie brillante Ideen im experimentellen Design und beim Data Mining zu teilen.
Zeitpunkt der Veröffentlichung: 06.01.2022

