LJUDSKA GENOMIKA
genetika prirode
Dugo čitano sekvenciranje identificira GGC ponovljene ekspanzije u NOTCH2NLC povezane s neuronskom intranuklearnom inkluzijskom bolešću
ONT ponovno sekvenciranje |Illumina |Sekvenciranje cijelog egzoma |CRISPR-Cas9 ONT ciljano sekvenciranje |RNA-seq |ONT 5mC metilacija poziva
Highlights
1. Analizom povezivanja na velikoj porodici NIID identifikovana su dva povezana regiona.
2. ONT sekvenciranje zasnovano na dugom čitanju i obogaćivanje posredovano Cas-9 ONT sekvenciranje otkrilo je potencijalni genetski uzrok ekspanzije ponavljanja NIID, GGC u 5′ UTR NOTCH2NLC.Ova studija je po prvi put izvijestila o ponovljenim ekspanzijama gena specifičnih za ljude koji su evoluirali kroz segmentne duplikacije.
3. Sekvenciranje RNK otkrilo je abnormalne antisense transkripte na početku ili unutar GGC ponovljenih ekspanzijskih regiona u NOTCH2NLC.
Pozadina
Neuronal intranuclear inclusion disease (NIID) je progresivna i fatalna neurodegenerativna bolest, koju karakteriše prisustvo eozinofilnih hijalinskih intranuklearnih inkluzija u centralnom i perifernom nervnom sistemu.Njegove vrlo varijabilne kliničke manifestacije stvaraju velike poteškoće u dijagnostici do uvođenja biopsije kože.Međutim, metode zasnovane na histopatologiji još uvijek pate od pogrešne dijagnoze, što zahtijeva genetsko razumijevanje NIID-a.
Dostignuća
Linkage Analysis
SSekvenciranje cijelog genoma (WGS) i sekvenciranje cijelog egzoma (WES) izvršeno je na velikoj porodici NIID (13 oboljelih i 7 nepogođenih članova).Analiza veze na SNP-ovima izvučena iz ovih podataka otkrila je samo dva povezana regiona: region od 3,5 Mb na 1p36.31-p36.22 (maksimalni LOD=2,32) i region od 58,1 Mb na 1p22.1-q21.3 (maksimalni LOD: 4,21 ).Međutim, u ovim povezanim regijama nisu identificirani patogeni SNP ili CNV.
GGC ponavljanje proširenja u NOTCH2NLC
NSekvenciranje zasnovano na anopore je obrađeno na 13 zahvaćenih i 4 neporažena člana iz 8 porodica (još jedan zahvaćeni član je sekvencioniran Pacbio platformom za dugo čitanje sekvenciranja.).Dugo čitani podaci otkrili su ekspanzije GGC ponavljanja povezanih sa bolešću u 5′ UTR mapiranja gena NOTCH2NLC u 58,1 Mb povezanu regiju (Slika 1).Ove ponovljene ekspanzije su takođe identifikovane u svih 40 sporadičnih NIID slučajeva testiranih RP-PCR.
CAs-9 posredovano ciljno sekvenciranje na platformi nanopora je korišćeno da bi se postigla veća pokrivenost čitanja na ponavljanju NOTCH2NLC (100 X-1,795 X).Ove konsenzusne sekvence su se dobro slagale sa prethodnim nalazima o GGC ponovljenim ekspanzijama.Štaviše, {(GGA)n (GGC)n}n ponavljanja su identifikovana kao potencijalni genetski marker za fenotip koji dominira slabošću (Slika 2).
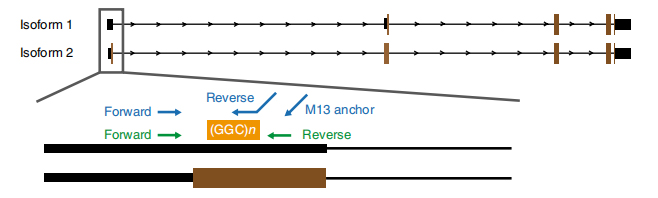
Slika 1. Ponovljena ekspanzija povezana s bolešću identificirana na egzonu 1 izoforma NOTCH2NLC.
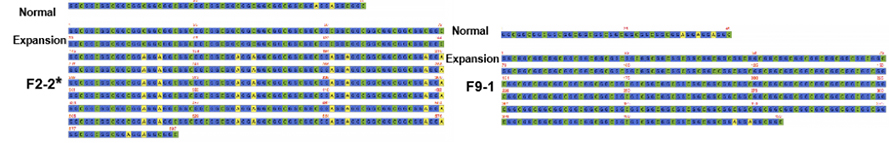
Slika 2. Konsenzus sekvence ponavljanja NPTCH2NLC kod pacijenata sa NIID sa (*) ili bez fenotipa koji dominira slabošću
NOTCH2NL geni su geni specifični za ljude, za koje se vjeruje da igraju vitalnu ulogu u evoluciji ljudskog mozga i neurološkim bolestima.Međutim, tri gena srodna NOTCH2 (NOTCH2NLA, NOTCH2NLB i NOTCH2NLC) sa >99,1% identičnosti sekvence nisu riješena do najnovijeg sklopa ljudskog genoma.Sekvenciranje bez sinteze i dugo čitanje na platformi nanopora pokazalo je značajne prednosti u rješavanju područja visoke sličnosti i (GGC)n ponavljanja sa 100% bogatim GC.
GGC ponavljanje proširenja u NOTCH2NLC
Tsekvenciranje rankriptoma je obrađeno na 2 zahvaćena i 2 neporažena člana.Normalizovana dubina čitanja izračunata je na čulnim i antisens lancima uzvodno od prvih egzona paraloga NOTCH2NL.Abnormalni anti-sense transkripti pronađeni su samo u zahvaćenim slučajevima, koji se nalaze na početku ili unutar regiona ponovljene ekspanzije (ljubičasti vrhovi na F1-14 i F1-16 na slici 3.).Pored toga, identifikovano je 54 DEG-a i svi su obogaćeni terminima GO i MPO koji se odnose na neuronske funkcije.
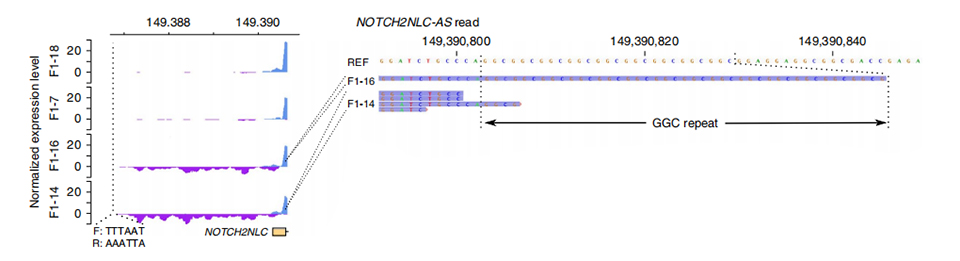
Slika 3. Normalizovana dubina čitanja uzvodno od prvog egzona NOTCH2NLC u slučajevima bez uticaja (iznad) i pogođenim (ispod).
Tehnologija
Oxford Nanopore Technologies (ONT)
Nanopore sekvenciranje razlikuje se od drugih platformi sekvenciranja po tome što se nukleotidi čitaju direktno bez procesa sinteze DNK.Kako jednolančana DNK prolazi kroz proteinske pore nano veličine (nanopore), različiti nukleotidi stvaraju različite jonske struje, koje se mogu uhvatiti i prenijeti u niz baza.Sama ONT platforma za sekvenciranje ne pokazuje očigledno tehničko ograničenje dužine očitavanja DNK.Stoga su ultra-duga čitanja (ULR) dostupna za sastavljanje genoma visokog kvaliteta.Štaviše, ova izuzetno duga čitanja, koja su dovoljno dugačka da pređu složene karakteristike sekvence ili strukturne varijacije, pomažu da se prevladaju ograničenja sekvenciranja kratkog čitanja.

Sekvenciranje nanopora
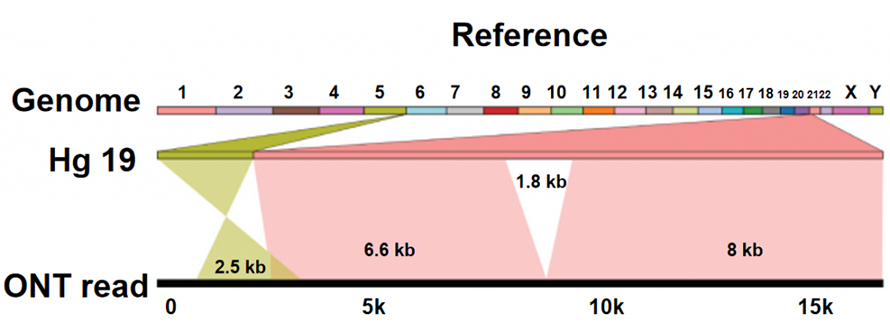
Identifikacija varijacije strukture (SV).
SSekvenciranje bez sinteze u velikoj mjeri je sačuvalo informacije o metilaciji DNK na šablonu.Metilirani A, T, C i G generiraju različite jonske struje od nemetiliranih, koje se mogu očitati direktno na platformi.Sekvenciranje nanopora omogućava profiliranje cijelog genoma i 5mC i 6mA pri rezoluciji jednog nukleotida.
Referenca
Jun Sone, et.al.Dugo čitano sekvenciranje identifikuje ekspanzije ponavljanja GGC-a u NOTCH2NLC povezane sa bolešću neuronske intranuklearne inkluzije.Prirodna genetika (2019)
Tech and Highlights ima za cilj dijeljenje najnovije uspješne primjene različitih tehnologija sekvenciranja visoke propusnosti u različitim istraživačkim arenama, kao i briljantnih ideja u eksperimentalnom dizajnu i rudarenju podataka.
Vrijeme objave: Jan-06-2022

