GENOMICA UMANA
genetica della natura
Il sequenziamento a lettura lunga identifica le espansioni di ripetizioni GGC in NOTCH2NLC associate alla malattia da inclusioni intranucleari neuronali
ONT risequenziamento |Illumina |Sequenziamento dell'intero esoma |Sequenziamento mirato CRISPR-Cas9 ONT |RNA-seq |Chiamata di metilazione ONT 5mC
Punti salienti
1. Mediante l'analisi di collegamento su un'ampia famiglia NIID, sono state identificate due regioni collegate.
2.Sequenziamento a lettura lunga basato su ONT e arricchimento mediato da Cas-9 Il sequenziamento ONT ha scoperto una potenziale causa genetica di NIID, espansioni di ripetizioni GGC in 5 'UTR di NOTCH2NLC.Questo studio ha riportato per la prima volta espansioni ripetute in geni specifici dell'uomo che si sono evoluti attraverso duplicazioni segmentali.
3. Il sequenziamento dell'RNA ha rivelato trascrizioni antisenso anomale all'inizio o all'interno delle regioni di espansione ripetuta GGC in NOTCH2NLC.
Sfondo
NLa malattia da inclusioni intranucleari euronali (NIID) è una malattia neurodegenerativa progressiva e fatale, caratterizzata dalla presenza di inclusioni intranucleari ialine eosinofile nel sistema nervoso centrale e periferico.Le sue manifestazioni cliniche altamente variabili sollevano grandi difficoltà nella diagnosi fino all'introduzione della biopsia cutanea.Tuttavia, i metodi basati sull’istopatologia sono ancora affetti da diagnosi errate, che richiedono una comprensione genetica del NIID.
Risultati
Analisi dei collegamenti
SIl sequenziamento dell'intero genoma basato sul sequenziamento hort-read (WGS) e il sequenziamento dell'intero esoma (WES) sono stati eseguiti su una vasta famiglia NIID (13 membri affetti e 7 membri non affetti).L'analisi di collegamento sugli SNP estratti da questi dati ha rivelato solo due regioni collegate: una regione da 3,5 Mb a 1p36.31-p36.22 (LOD massimo = 2,32) e una regione da 58,1 Mb a 1p22.1-q21.3 (LOD massimo: 4,21 ).Tuttavia, in queste regioni collegate non sono stati identificati SNP o CNV patogeni.
Espansioni ripetute di GGC in NOTCH2NLC
NIl sequenziamento basato su anopori è stato elaborato su 13 membri affetti e 4 membri non affetti provenienti da 8 famiglie (un altro membro affetto è stato sequenziato dalla piattaforma di sequenziamento a lettura lunga Pacbio).I dati a lunga lettura hanno rivelato espansioni di ripetizioni GGC associate alla malattia nel 5' UTR della mappatura del gene NOTCH2NLC nella regione collegata a 58,1 Mb (Figura 1).Queste espansioni ripetute sono state identificate anche in tutti i 40 casi NIID sporadici testati mediante RP-PCR.
CIl sequenziamento del target mediato da as-9 su piattaforma di nanopori è stato impiegato per ottenere una copertura di lettura più elevata sulla ripetizione NOTCH2NLC (100 X-1.795 X).Queste sequenze consenso concordavano bene con i risultati precedenti sulle espansioni delle ripetizioni GGC.Inoltre, le ripetizioni {(GGA)n (GGC)n}n sono state identificate come potenziale marcatore genetico per il fenotipo dominante la debolezza (Figura 2).
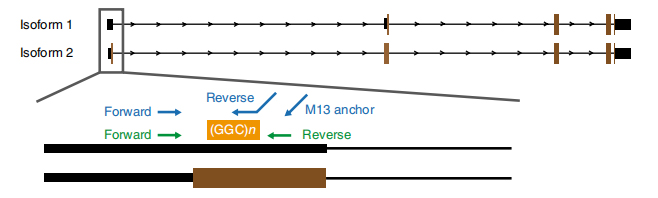
Figura 1. Espansione ripetuta associata alla malattia identificata sull'esone 1 delle isoforme NOTCH2NLC.
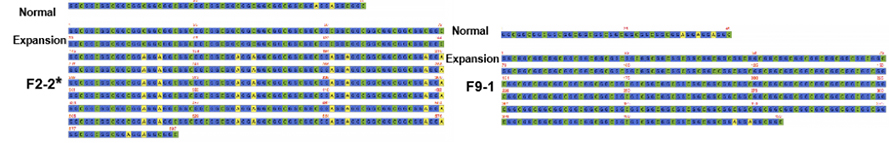
Figura 2. Sequenze consenso di NPTCH2NLC ripetute in pazienti NIID con (*) o senza fenotipo con debolezza dominante
NI geni OTCH2NL sono geni specifici dell'uomo, che si ritiene svolgano un ruolo vitale nell'evoluzione del cervello umano e nelle malattie neurologiche.Tuttavia, tre geni correlati a NOTCH2 (NOTCH2NLA, NOTCH2NLB e NOTCH2NLC) con un'identità di sequenza >99,1% non sono stati risolti fino all'ultimo assemblaggio del genoma umano.Il sequenziamento senza sintesi e a lunga lettura su piattaforma di nanopori ha mostrato notevoli vantaggi nella risoluzione di regioni ad elevata somiglianza e ripetizioni (GGC)n con il 100% di ricchezza in GC.
Espansioni ripetute di GGC in NOTCH2NLC
Til sequenziamento del ranscrittoma è stato elaborato su 2 membri affetti e 2 membri non affetti.La profondità di lettura normalizzata è stata calcolata sui filamenti senso e antisenso a monte dei primi esoni dei paraloghi NOTCH2NL.Trascrizioni antisenso anomale sono state riscontrate solo nei casi affetti, che si trovano all'inizio o all'interno della regione di espansione ripetuta (picchi viola in F1-14 e F1-16 nella Figura 3).Inoltre, sono stati identificati 54 DEG e tutti sono stati arricchiti in termini GO e MPO relativi alle funzioni neuronali.
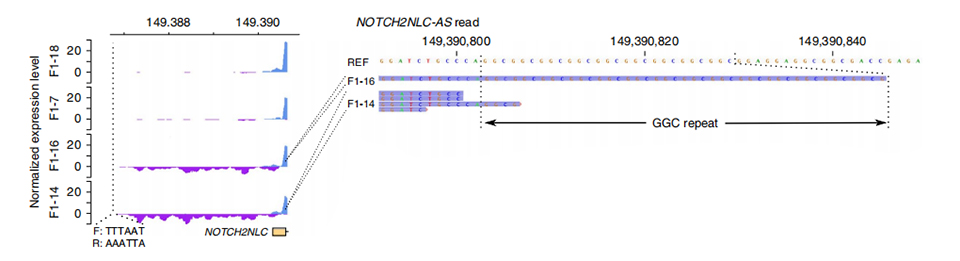
Figura 3. Profondità di lettura normalizzata a monte del primo esone di NOTCH2NLC nei casi non affetti (sopra) e affetti (sotto).
Tecnologia
Tecnologie dei nanopori di Oxford (ONT)
NIl sequenziamento di anopori si distingue dalle altre piattaforme di sequenziamento, in quanto i nucleotidi vengono letti direttamente senza processo di sintesi del DNA.Quando un singolo filamento di DNA passa attraverso un poro proteico di dimensioni nanometriche (nanoporo), diversi nucleotidi generano diverse correnti ioniche, che possono essere catturate e trasferite in una sequenza di basi.La stessa piattaforma di sequenziamento ONT non mostra limiti tecnici evidenti sulla lunghezza della lettura del DNA.Pertanto, sono disponibili letture ultra lunghe (ULR) per l'assemblaggio del genoma di alta qualità.Inoltre, queste letture estremamente lunghe, sufficientemente lunghe da attraversare caratteristiche di sequenze complesse o variazioni strutturali, aiutano a superare i limiti del sequenziamento di letture brevi in questo caso.

Sequenziamento dei nanopori
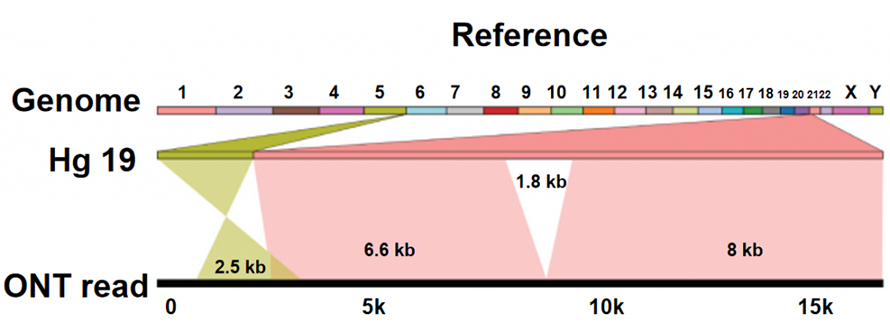
Identificazione della variazione della struttura (SV).
Sil sequenziamento privo di sintesi ha ampiamente conservato le informazioni sulla metilazione del DNA sul modello.A, T, C e G metilati generano correnti ioniche distinte da quelle non metilate, che possono essere lette direttamente dalla piattaforma.Il sequenziamento dei nanopori consente la profilazione dell'intero genoma sia di 5 mC che di 6 mA con risoluzione a singolo nucleotide.
Riferimento
Jun Sone, et.al.Il sequenziamento a lettura lunga identifica le espansioni di ripetizioni GGC in NOTCH2NLC associate alla malattia da inclusioni intranucleari neuronali.Genetica della natura (2019)
Tecnologia e punti salienti mira a condividere le più recenti applicazioni di successo di diverse tecnologie di sequenziamento ad alto rendimento in vari ambiti di ricerca, nonché idee brillanti nella progettazione sperimentale e nel data mining.
Orario di pubblicazione: 06 gennaio 2022

