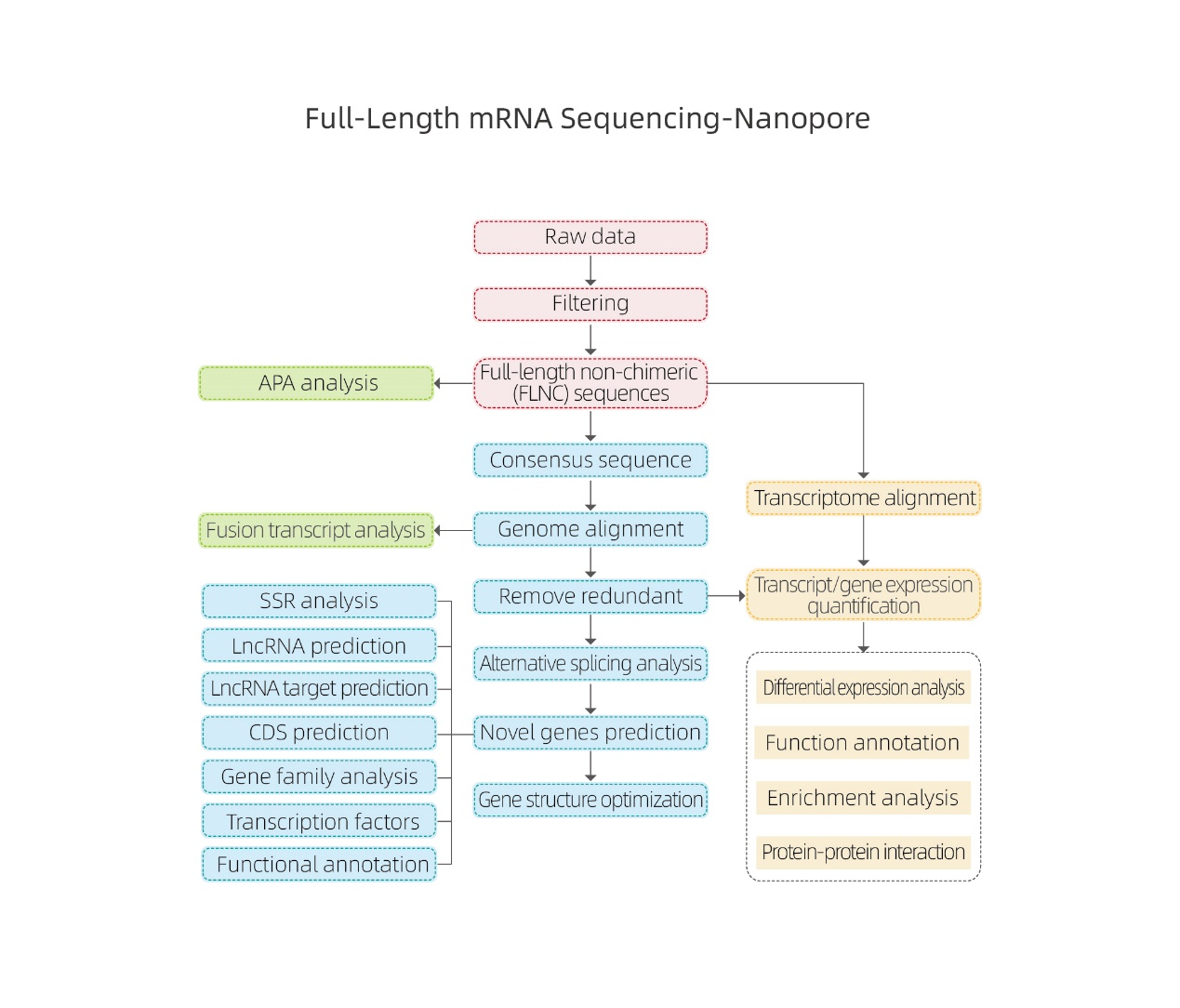
Voll-Längt mRNA Sequencing-Nanopore
Service Virdeeler
● Niddereg Sequenz Viraussetzung
● Volllängt cDNA Moleküle opzeweisen
● Manner Donnéeën erfuerderlech fir déiselwecht Unzuel vun Transkriptiounen ze decken
● Identifikatioun vu multiple Isoformen pro Gen
● Ausdrockquantifikatioun am Isoformniveau
Service Spezifikatioune
| Bibliothéik | Plattform | Recommandéiert Daterendung (Gb) | Qualitéitskontroll |
| cDNA-PCR (Poly-A beräichert) | Nanopore PromethION P48 | 6 Gb/Prouf (je no Arten) | Volllängt Verhältnis> 70% Duerchschnëtt Qualitéit Score: Q10
|
Bioinformatik Analysen
●Raw Datenveraarbechtung
● Transkript Identifikatioun
● Alternativ Splicing
● Ausdrockquantifikatioun am Genniveau an Isoformniveau
● Differential Ausdrock Analyse
● Funktiounsannotatioun a Beräicherung (DEGs an DETs)
Prouf Ufuerderunge a Liwwerung
Sample Ufuerderunge:
Nukleotiden:
| Konzentratioun (ng/μl) | Betrag (μg) | Rengheet | Integritéit |
| ≥ 100 | ≥ 0,6 | OD260/280=1,7-2,5 OD260/230=0,5-2,5 Limitéiert oder keng Protein oder DNA Kontaminatioun op Gel gewisen. | Fir Planzen: RIN≥7.0; Fir Déieren: RIN≥7,5; 5.0≥28S/18S≥1.0; limitéiert oder keng Basislinn Héicht |
Tissue: Gewiicht (dréchen): ≥1 g
* Fir Tissue méi kleng wéi 5 mg, empfeelen mir Flash gefruerene (a flëssege Stickstoff) Tissueprobe ze schécken.
Zellsuspension: Zellzuel = 3 × 106- 1 × 107
* Mir recommandéieren gefruer Zell lysate ze Schëff.Am Fall datt Zelle méi kleng wéi 5 × 10 zielt5, Flash gefruer a flëssege Stickstoff ass recommandéiert, wat am léifsten fir Mikro Extraktioun ass.
Bluttprouwen: Volume ≥1 ml
Recommandéiert Sample Liwwerung
Behälter: 2 ml Zentrifuge Röhre (Zinnfolie ass net recommandéiert)
Probe Etikettéierung: Grupp + replizéieren zB A1, A2, A3;B1, B2, B3...
Versand: 2, Dréchent Äis: D'Probe mussen a Poschen gepackt ginn an an dréchen Äis begruewe ginn.
- RNAstable Réier: RNA Proben kënnen an RNA Stabiliséierungsröhre gedréchent ginn (zB RNAstable®) a bei Raumtemperatur verschéckt ginn.
Service Work Flow
Nukleotiden:

Echantillon Liwwerung

Bibliothéik Bau

Sequenzéieren

Donnéeën Analyse

No-Verkaf Servicer
Service Work Flow
Tissue:

Experimenter Design

Echantillon Liwwerung

RNA Extraktioun

Bibliothéik Bau

Sequenzéieren

Donnéeën Analyse

No-Verkaf Servicer
1.Differential Ausdrock Analyse -Vulkan Komplott
Differential Ausdrock Analyse kann a béid Genniveau veraarbecht ginn fir differenziell ausgedréckte Genen (DEGs) ze identifizéieren an am Isoformniveau fir differentiell z'identifizéieren
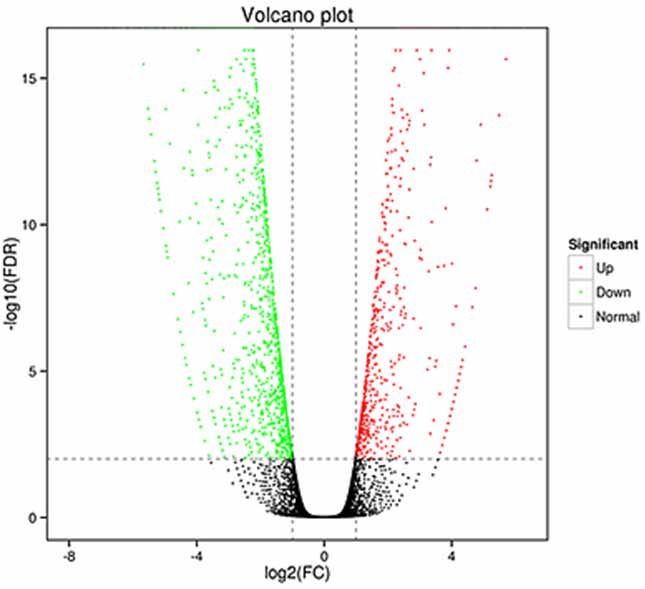
ausgedréckte Transkriptiounen (DETs)
2.Hierarchesch Clustering Hëtztkaart
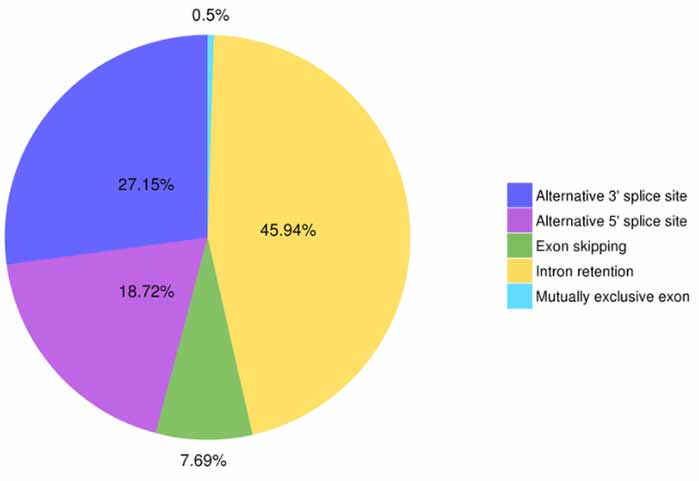
3.Alternativ Splicing Identifikatioun a Klassifikatioun
Fënnef Aarte vun alternativ Splicing Eventer kënne vun Astalavista virausgesot ginn.
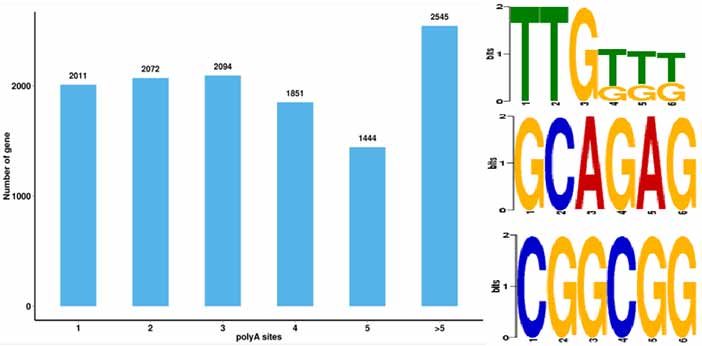
4.Alternativ Poly-Adenylatioun (APA) Eventer Identifikatioun a Motiv op 50 bp Upstream vu Poly-A
BMK Fall
Alternativ Splicing Identifikatioun an Isoform-Niveau Quantifikatioun duerch Nanopore Volllängt Transkriptom Sequenzéierung
Verëffentlecht:Naturkommunikatioun, 2020
Sequenzéierungsstrategie:
Gruppéierung: 1. CLL-SF3B1 (WT);2. CLL-SF3B1 (K700E Mutatioun);3. Normal B-Zellen
Sequenzéierungsstrategie: MinION 2D Bibliothéik Sequenzéierung, PromethION 1D Bibliothéik Sequenzéierung;kuerz liesen Daten aus selwechten Echantillon
Sequenzéierungsplattform: Nanopore MinION;Nanopore PromethION;
Schlëssel Resultater
1.Isoform-Niveau Alternativ Splicing Identifikatioun
Laang gelies Sequenzen erméiglecht d'Identifikatioun vu mutanten SF3B1K700E-verännert Splice Siten op Isoform-Niveau.35 alternativ 3'SSs an 10 alternativ 5'SSs goufe wesentlech differenziell tëscht SF3B1 gespléckt.K700Ean SF3B1WT.33 vun de 35 Ännerunge goufen nei duerch laang gelies Sequenzen entdeckt.
2.Isoform-Niveau Alternativ Splicing Quantifikatioun
Ausdrock vun Intron Retention (IR) Isoformen am SF3B1K700Ean SF3B1WTquantifizéiert baséiert op Nanopore Sequenzen, enthüllt eng global Down-Regulatioun vun IR Isoformen am SF3B1K700E.
Referenz
Tang AD, Soulette CM, Baren MJV, et al.Volllängt Transkriptkarakteriséierung vun der SF3B1 Mutatioun bei chronescher lymphozyter Leukämie verroden Downregulatioun vun erhale Intronen [J].Natur Kommunikatiounen.